Zellabstammung



Die Zellabstammung bezeichnet die Entwicklungsgeschichte einer Zelle in einem Gewebe oder Organ vom Embryo an.[1] Dabei differenzieren embryonale Stammzellen zu spezialisierten Zellen. Die graphische Darstellung der Zellabstammung wird als Zellstammbaum bezeichnet.
Eigenschaften
Die jeweilige Zellabstammung entsteht aufgrund der spatiotemporalen Genexpression. Eutele Lebewesen besitzen eine festgelegte Anzahl und Entwicklungsschritten von Zellen, z. B. beim Fadenwurm Caenorhabditis elegans (1031 im männlichen Fadenwurm bzw. nach Apoptose 959 Zellen im hermaphroditischen Fadenwurm).[2][3] Für die vollständige Beschreibung der Zellabstammung von C. elegans erhielt John Sulston 2002 den Nobelpreis für Physiologie oder Medizin.
Menschen
Beim Menschen können aus einer befruchteten Eizelle oder aus einer totipotenten Stammzelle mindestens 210 Zelltypen entstehen. Die Anzahl der Zellen je Zelltyp ist individuell variabel.
Methoden
Die Zellabstammung kann z. B. durch eine klonale Analyse oder durch mikroskopische Beobachtung ermittelt werden,[4] teilweise auch in Verbindung mit einer Immunfärbung oder als Einzelzellanalyse.[5] Durch Konfokalmikroskopie können Gewebe mit etwas größerer Schichtdicke untersucht werden.[6]
Geschichte
Ab dem Jahr 1870 wurden erstmals die Muster der Zellteilungen und die Spezialisierung der Zellen in Nematoden und Ascidien durch Charles Otis Whitman beschrieben.[6][7] Er beobachtete, dass in diesen Organismen das Muster und die Spezialisierung individuell invariabel sind. In anderen Organismen sind die Anzahl der Zellen und ihre Entwicklung nicht so sehr festgelegt, vermutlich aufgrund äußerer Einflüsse.[6]
Weblinks
Einzelnachweise
- ↑ Collins English Dictionary - Complete & Unabridged 10th Edition.. HarperCollins Publishers., (Abgerufen am 2. Juni 2014).
- ↑ J.E. Sulston, H.R. Horvitz: Post-embryonic cell lineages of the nematode, Caenorhabditis elegans. In: Developmental Biology. 56, Nr. 1, 1977, S. 110–56. doi:10.1016/0012-1606(77)90158-0. PMID 838129.
- ↑ J. Kimble, D. Hirsh: The postembryonic cell lineages of the hermaphrodite and male gonads in Caenorhabditis elegans. In: Developmental Biology. 70, Nr. 2, 1979, S. 396–417. doi:10.1016/0012-1606(79)90035-6. PMID 478167.
- ↑ F. Amat, P. J. Keller: Towards comprehensive cell lineage reconstructions in complex organisms using light-sheet microscopy. In: Development, growth & differentiation. Band 55, Nummer 4, Mai 2013, ISSN 1440-169X, S. 563–578, doi:10.1111/dgd.12063, PMID 23621671.
- ↑ T. M. Gibson, C. A. Gersbach: The role of single-cell analyses in understanding cell lineage commitment. In: Biotechnology journal. Band 8, Nummer 4, April 2013, ISSN 1860-7314, S. 397–407, doi:10.1002/biot.201200201, PMID 23520130, PMC 3753774 (freier Volltext).
- ↑ a b c A. D. Chisholm: Cell Lineage Archiviert vom Original am 9. April 2011. In: Encyclopedia of Genetics. 2001, S. 302–310. doi:10.1006/rwgn.2001.0172. Abgerufen am 2. Juni 2014.
- ↑ Charles Galperin: From Cell Lineage to Developmental Genetics. In: History and Philosophy of the Life Sciences. 20, Nr. 3, 1998, S. 301–350.
Auf dieser Seite verwendete Medien
Autor/Urheber: Zorn, A.M., Liver development (October 31, 2008), StemBook, ed. The Stem Cell Research Community, StemBook, doi/10.3824/stembook.1.25.1, http://www.stembook.org., Lizenz: CC BY 3.0
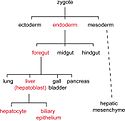
The cell lineage steps during hepatic development (red) from uncommitted endoderm to functional adult hepatocytes and biliary epithelium.
Autor/Urheber: Murtaugh, L.C. and Kopinke, D., Pancreatic stem cells (July 11, 2008), StemBook, ed. The Stem Cell Research Community, StemBook, doi/10.3824/stembook.1.3.1, http://www.stembook.org., Lizenz: CC BY 3.0
Pancreas specification within the foregut endoderm is first indicated by expression of the transcription factor Pdx1, which marks the progenitors of all exocrine and endocrine cell types. As the embryonic organ grows, multipotent progenitor cells also express the transcription factor Ptf1a and the digestive enzyme Cpa1. These progenitors are later segregated into specific sub-lineages, prior to differentiation. Acinar cells arise from precursors that express high levels of Ptf1a and Cpa1, while islet cells arise from precursors that transiently express the transcription factor Neurog3. Although Neurog3-expressing cells arise from duct-like epithelial progenitors (see Figure 2), the lineage relationship between these structures and the ducts of the mature organ is unclear (question marks).
Complete cell lineage of C. elegans.
The history of cellular divisions that sequentially tracks the parents of a particular cell up to its original ancestor, the zygote cell, is called cell lineage. Simple organisms such as the C. elegans worm tend to have a deterministic cell lineage, meaning that for any given embryo, the cells divide following exactly the same pattern. More complex organisms tend to have a deterministic lineage only in the early stages of embryogenesis and then, as large numbers of cells of similar type are required, they tend to generate these cells by following non-deterministic, although likely sterotypic, patterns.
Studying the cell lineage of an organism is important because it helps in the understanding of how a particular cell defines its fate in the organism. It also helps to understand how mutations and environmental factors may perturb the embryogenesis process and result in malformed organisms. In the context of studying gene functionality, the cell lineage also provides a baseline layer upon which the behavior of genes can be laid out across space and time as the organism develops.