Viroplasma

Als Viroplasma (englisch viroplasm) bezeichnet man jenen Ort innerhalb einer virusinfizierten Zelle, an dem die eigentliche Produktion von Virusbestandteilen stattfindet (Virusfabrik, Virionenfabrik, englisch virus factory, virion factory, VF, oder virus assembly site, viral assembly site, assembly site, VAS, AS).[1][2][3] Meist ist das Viroplasma an morphologischen Veränderungen im Vergleich zu einer nicht-infizierten Zelle im Elektronenmikroskop zu erkennen bzw. zu vermuten; in vielen Fällen fehlt jedoch der direkte Nachweis. Ein Beispiel ist hierfür das erst kürzlich beschriebene sogenannte „membranöse Netz“ (englisch membranous web) in Zellen, die mit einem Replikon des Hepatitis-C-Virus transfiziert wurden. Hier zeigte sich das Viroplasma als eine Zusammenballung von Membrankomplexen innerhalb des Cytoplasmas.
Bei einigen Viren kann das Viroplasma bereits im Lichtmikroskop erkannt werden, entweder nach bestimmten Färbemethoden oder durch deutliche cytologische Veränderungen. Diese Veränderungen wurden schon in der Anfangszeit der Zellpathologie als „Einschlusskörperchen“ (englisch inclusion body, X-body) beschrieben und später der Virusvermehrung zugeschrieben. Zum Teil handelt es sich um Ablagerungen von viralen Proteinen oder Viruspartikeln, meist jedoch um besonders ausgedehnte und veränderte Regionen des Endoplasmatischen Retikulums oder des Golgi-Apparates, die der Virusvermehrung dienen. Ein Beispiel für diese cytoplasmatischen Einschlusskörperchen sind die Negri-Körper (benannt nach ihrem Entdecker, dem Pathologen Adelchi Negri) in Zellen, die das Rabiesvirus vermehren.
Das Viroplasma kann im Cytoplasma als auch im Zellkern (Karyoplasma) lokalisiert sein. Als nukleäres Viroplasma findet es sich im Zellkern jedoch nur bei Viren, zu deren Vermehrung die zellulären DNA- oder RNA-Polymerasen benötigt werden (z. B. bei Polyomaviren und Herpesviren).
Quellen
- Reyes R. Novoa, Gloria Calderita et al.: Virus factories: associations of cell organelles for viral replication and morphogenesis. Biology of the Cell (2005) 97: S. 147–172, PMID 15656780
Einzelnachweise
- ↑ Haruna Takahashi, Sho Fukaya, Chihong Song, Kazuyoshi Murata, Masaharu Takemura: Morphological and Taxonomic Properties of the Newly Isolated Cotonvirus japonicus, a New Lineage of the Subfamily Megavirinae. In: ASM Journals: Journal of Virology, Band 95, Nr. 18, 25. August 2021; doi:10.1128/JVI.00919, ResearchGate.
- ↑ Graziele Oliveira, Bernard La Scola, Jônatas Abrahão: Giant virus vs amoeba: fight for supremacy, in: Virol J 16, 126, 4. November 2019, doi:10.1186/s12985-019-1244-3, PDF
- ↑ Yang Liu, Bich Ngoc Tran, Fan Wang, Puey Ounjai, Jinlu Wu, Choy L. Hew: Visualization of Assembly Intermediates and Budding Vacuoles of Singapore Grouper Iridovirus in Grouper Embryonic Cells, in: Sci Rep 6, 18696, 4, Januar 2016, doi:10.1038/srep18696
Weblinks
- Masaharu Takemura: Medusavirus Ancestor in a Proto-Eukaryotic Cell: Updating the Hypothesis for the Viral Origin of the Nucleus, in: Front. Microbiol. 11:571831, 3. September 2020, doi:10.3389/fmicb.2020.571831
- Thomas G. Laughlin, Amar Deep, Amy M. Prichard, Christian Seitz, Yajie Gu, Eray Enustun, Sergey Suslov, Kanika Khanna, Erica A. Birkholz, Emily Armbruster, J. Andrew McCammon, Rommie E. Amaro, Joe Pogliano, Kevin D. Corbett, Elizabeth Villa: Architecture and self-assembly of the jumbo bacteriophage nuclear shell. In: Nature, 3. August 2022; doi:10.1038/s41586-022-05013-4. Dazu:
- Tessa Koumoundouros: Giant Viruses Called 'Jumbo Phages' Could Help Us Fight Antibiotic Resistance. Auf: sciencealert vom 5. August 2022.
Siehe auch
- Phikzvirus §Phagenkern (englisch phage nucleus)
Auf dieser Seite verwendete Medien
(c) Graham Beards at the English Wikipedia, CC BY-SA 3.0
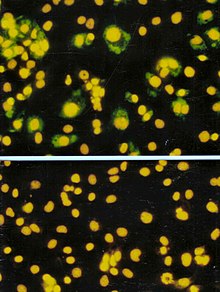
Cells infected with rotavirus (top) and labelled with a monoclonal antibody tagged with Fluorescein Isothiocyanate (FITC) compared with uninfected cells (bottom).
Autor/Urheber: Paramecium, Lizenz: CC BY-SA 3.0
Immunfluoreszenz-Aufnahme des Virusstammes CP7, des Bovinen Viralen Diarrhöe Virus (BVDV). Die Zellkerne wurden mit DAPI angefärbt. Das Anfärben des NS3 Proteins erfolgte mittels primärem Antikörper zur Proteinbindung und sekundärem Antikörper zum Anfärben (anti-mouse-Rhodamin Red, Alexa 555)