Tomato-bushy-stunt-Virus
| Tomato-bushy-stunt-Virus | ||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 Das Kapsid des Tomato-bushy-stunt-Virus | ||||||||||||||||||||||
| Systematik | ||||||||||||||||||||||
| ||||||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||||||
| ||||||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||||||
| Tomato bushy stunt virus | ||||||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||||||
| TBSV | ||||||||||||||||||||||
| Links | ||||||||||||||||||||||
|
Das Tomato-bushy-stunt-Virus (selten auch Tomatenzwergbusch-Virus,[4] englisch Tomato bushy stunt virus, TBSV; Spezies Tombusvirus lycopersici) bezeichnet, ist ein Pflanzenvirus aus der Familie Tombusviridae, Unterfamilie Procedovirinae. Die Spezies ist die ehemalige Typusart der Gattung Tombusvirus.[5][6][7]
TBSV ist ein Einzelstrang-RNA-Virus, das erstmals 1935 in Tomaten gefunden wurde. Es befällt hauptsächlich Gemüsepflanzen, wird aber allgemein nicht als wirtschaftlich signifikantes Pflanzenpathogen angesehen. Je nach Wirt verursacht TBSV Wachstumsstörungen, Blattflecken und deformierte oder fehlende Früchte. Das Virus ist in der Natur wahrscheinlich bodengebunden, kann aber auch mechanisch übertragen werden, beispielsweise durch kontaminierte Schneidwerkzeuge. TBSV wurde als Modellsystem in der virologischen Forschung zum Lebenszyklus von Pflanzenviren verwendet, insbesondere bei experimentellen Infektionen der Modellwirtspflanze Nicotiana benthamiana.[8][9]
Das verkümmerte (englisch to stunt ‚verkümmern‘), „buschige“ Aussehen der Tomatenpflanzen, in denen das Virus erstmals entdeckt wurde, gab dem Erreger seinen Namen.
Wirtspflanzen

TBSV verfügt unter experimentellen Bedingungen über ein breites Wirtsspektrum und infiziert Berichten zufolge über 120 verschiedene Pflanzenarten aus 20 Familien. Unter natürlichen Bedingungen ist sein Verbreitungsgebiet jedoch viel enger und umfasst im Allgemeinen Erntegemüse und Zierpflanzen. Es wurde erstmals in Tomatenpflanzen nachgewiesen, ist aber auch als Parasit von Apfel, Artischocke, Kirsche, Weinrebe, Hopfen, Pfeffer und Wald-Erdbeeren[4]dokumentiert. Obwohl TBSV bei Tomatenpflanzen zu erheblichen Ertragsverlusten führt, wird es nicht als wirtschaftlich bedeutender Erreger angesehen.[9][10] Es ist jedoch ein sehr gut etabliertes Modellsystem für die Untersuchung von Pflanzenviren, üblicherweise durch experimentelle Infektion von Nicotiana benthamiana oder N. clevelandii, Verwandten der Tabakpflanze, bei denen TBSV eine systemische Infektion verursachen kann. Die gemeinsame Modellpflanze Arabidopsis thaliana ist jedoch kein Wirt.[8][9] Interessanterweise kann TBSV unter Laborbedingungen auch in Hefe (Bierhefe Saccharomyces cerevisiae) replizieren.[11]
Infektion und ihre Anzeichen
Die Anzeichen von TBSV sind wirtsabhängig. Lokale Infektionen können nekrotische oder chlorotische Läsionen verursachen. Systemische Infektionen können zu Wachstumsstörungen, deformierten oder fehlenden Früchten und beschädigten Blättern führen wie beispielsweise dem verkümmerten, „buschigen“ Aussehen der Tomatenpflanzen. In der Landwirtschaft kann der Ertrag deutlich reduziert werden. Bei einigen Wirten, insbesondere bei N. benthamiana, kann TBSV tödliche systemische Nekrose verursachen.[9][10]
Übertragung
Es wird angenommen, dass TBSV in der Wildnis passiv übertragen wird, hauptsächlich über den Boden und Wasser. Es sind keine Vektororganismen bekannt, insbesondere ist eine Übertragung durch Blattläuse, Milben oder den Tröpfchenpilz Olpidium brassicae (Olpidiaceae) ausdrücklich ausgeschlossen.[10] Es wurde jedoch beobachtet, dass das eng verwandte Cucumber-necrosis-Virus (CNV), ebenfalls Virusgattung Tombusvirus, von Zoosporen der nahe verwandten Tröpfchenpilsspezies O. bornovanus übertragen wird, so dass die Übertragung von TBSV durch einen bislang unbekannten Vektor weiterhin möglich ist.[9] TBSV kann auch durch Samen oder durch mechanische Inokulation (Beimpfung) übertragen werden.[9][10] In experimentellen Tests kann das Virus die Passage durch das menschliche Verdauungssystem überleben, wenn es in der Nahrung aufgenommen wird, und bleibt ansteckend. Daher vermutet man, dass eine Ausbreitung durch das Abwasser erfolgen könnte.[12]
Verbreitung und Gegenmaßnahmen
TBSV ist in Mittel- und Westeuropa, Nordafrika sowie Nord- und Südamerika ziemlich weit verbreitet.[9][10] Es gibt zwar keine Empfehlungen für spezifischen Bekämpfungsmaßnahmen. Die von der University of California herausgegebenen Richtlinien zur Schädlingsbekämpfung empfehlen jedoch, Felder mit einer TBSV-Vorgeschichte zu meiden oder lange Fruchtfolgen zu verwenden.[13]
Systematik
TBSV ist die Typusart der Gattung Tombusvirus in der Familie Tombusviridae, Unterfamilie Procedovirinae.[2][14] Sowohl die Gattung als auch die Familie haben ihren Namen von einer Abkürzung für englisch tomato bushy stunt virus abgeleitet.[15] Liste der Mitglieder der Gattung (Stand Mai 2024):[6][7]
Familie Tombusviridae
- Unterfamilie Procedovirinae
- Gattung Tombusvirus
- Spezies Tombusvirus algeriaense mit Grapevine Algerian latent virus (GALV)
- Spezies Tombusvirus bulgariaense mit Cucumber Bulgarian latent virus alias Cucumber Bulgarian virus (CBLV-Bulgaria)
- Spezies Tombusvirus cucumis mit Cucumber necrosis virus (CNV, Cucumber-necrosis-Virus)
- Spezies Tombusvirus cymbidii mit Cymbidium ringspot virus (CyRV)
- Spezies Tombusvirus cynarae mit Artichoke mottled crinkle virus (AMCV)
- Spezies Tombusvirus dianthi mit Carnation Italian ringspot virus (CIRV)
- Spezies Tombusvirus havelfluminis mit Havel River virus (HRV)
- Spezies Tombusvirus latofluminis mit Lato River virus (LRV)
- Spezies Tombusvirus limonii mit Limonium flower distortion virus (LFDV)
- Spezies Tombusvirus lycopersici (ehem. Typusspezies) mit Tomato bushy stunt virus (TBSV, Tomato-bushy-stunt-Virus), Isolate (Auswahl) nach National Center for Biotechnology Information (NCBI)[16]
- TBSV strain A23
- TBSV strain b8
- TBSV strain BS-3
- TBSV strain CHERRY
- TBSV strain ja6
- TBSV strain ja9
- TBSV strain type
- Spezies Tombusvirus melongenae mit
- Eggplant mottled crinkle virus (EMCV)
- Lisianthus necrosis virus L (LNV-L)
- Lisianthus necrosis virus Zantedeschia (LNV-Zantedeschia), Wirt: Zantedeschia jucunda
- Pear latent virus (PLV)
- Spezies Tombusvirus moroccoense mit
- Moroccan pepper virus (MPV)
- Lettuce necrotic stunt virus (LNSV)
- Spezies Tombusvirus neckarfluminis mit Neckar River virus (NRV)
- Spezies Tombusvirus necropelargonii mit Pelargonium necrotic spot virus (PNSV)
- Spezies Tombusvirus pelargonii mit Pelargonium leaf curl virus (PLCV)
- Spezies Tombusvirus petuniae mit Petunia asteroid mosaic virus (PAMV)
- Spezies Tombusvirus siktefluminis mit Sikte waterborne virus (SWBV)
- Gattung Tombusvirus
Aufbau

Die Virusteilchen (Virionen) von TBSV sind nicht umhüllt, und mit ikosaedrischer Geometrie (T=3-Symmetrie). Das Kapsid ist aus 180 Untereinheiten eines einzelnen Kapsidproteins zusammengesetzt. Seine Struktur wurde ab den späten 1950er Jahren durch Röntgenkristallographie eingehend untersucht. Dabei wurde die ikosaedrische Symmetrie erstmals von dem Strukturbiologen Donald Caspar identifiziert, der auch Pionierarbeit bei der Erforschung des Tabakmosaikvirus leistete.[17] 1978 wurde von einem Forscherteam um Stephen C. Harrison eine Darstellung mit nahezu atomarer Auflösung erzielt.[18][19]
Genom und Proteom

TBSV ist ein einzelsträngiges RNA-Virus mit positiver Polarität und einem linearen Genom mit einer Länge von etwa 4800 nt.[20][21] Dem RNA-Strang fehlen der 3'-Polyadenin-Schwanz und die 5'-Kappe.
Das Genom enthält fünf Gene die eine Replikase kodieren, die wie folgt zusammengesetzt ist:[9]
- zwei Proteinen (p33 und p92),
- ein Kapsidprotein (CP oder p41 genannt),
- zwei weiteren Proteine: dem RNA-Silencing-Suppressor p19 und dem Movement-Protein p22,
- möglicherweise gibt es ein weiteres Gen unbekannter Funktion mit der Bezeichnung pX.[9]
Die beiden Proteine p19 und p22 werden von überlappenden Genen exprimiert, die so angeordnet sind, dass der offene Leserahmen (englisch open reading frame, ORF) von p19 vollständig im ORF von p22 liegt.[22]
p33 und p92
Die beiden Proteine p33 und p92 bilden zusammen den viralen Replikase-Komplex. Dabei ist p33 ist kleiner als p92. Letzteres wird durch ribosomales Durchlesen des p33-Stoppcodons erzeugt, was zu einer gemeinsamen N-terminalen Aminosäuresequenz und einem großen Überschuss an p33 im Vergleich zu p92 führt. Die p33-Proteine binden kooperativ einzelsträngige Nukleinsäuren, während das p92-Protein eine RNA-abhängige RNA-Polymerase (RdRp) ist. Beide sind für die Virusproliferation (die Vermehrung der Virusteilchen) essentiell. Beide Proteine sind mit Zellmembranen assoziiert.[9]
p41 (Kapsidprotein)
Das Kapsidprotein p41 (alias CP) ist ein doppeltes Jelly-Roll-Protein (englisch jelly roll fold). Das ikosaedrischen Kapsid zusammensetzt setzt sich aus 180 Kopien des Proteins zusammen. Um das Virus lokal in benachbarte Pflanzenzellen zu verbreiten, ist die Bildung von Virionen nicht zwingend notwendig, da sich Ribonukleoprotein-Partikel mit dem viralen genetischen Material über Plasmodesmata (Plasmodesmen) zu den unmittelbaren Nachbarzellen ausbreiten können. Für eine systemische Infektion (anderer Pflanzen) durch voll funktionsfähige Virionen wird jedoch das Kapsidprotein benötigt.[9]
p19
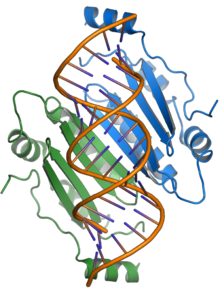
Das Protein p19 ist ein Pathogenitätsfaktor und unterdrückt den RNA-Silencing-Signalweg, eine häufige Form der antiviralen Abwehr seitens der Wirte. Das p19-Protein bindet kurze sRNAs („kleine RNA“s) und verhindert deren Einbau in den RNA-induzierten Silencing-Komplex (RISC), wodurch die Virusvermehrung in der Wirtspflanze ermöglicht wird.[8][23][24] Das Vorhandensein von p19 ist bei einigen Wirten für eine systemische oder tödliche Infektion erforderlich; im experimentellen Wirt N. benthamiana wird die letale systemische Nekrose als Ergebnis der TBSV-Infektion weitgehend durch p19 vermittelt.[9][23]
p22 (Movement-Protein)
Das Protein p22 ist ein Movement-Protein, das für die Ausbreitung des Virus von Zelle zu Zelle erforderlich ist. Es handelt sich um ein RNA-bindendes Protein, das mit der Zellwand assoziiert ist und die Bewegung von viralem genetischem Material von einer Zelle zu seinem Nachbarn durch verbindende Plasmodesmen erleichtert.[9][25]
Replikation
Obwohl dem RNA-Strang des Genoms der 3'-Polyadenin-Schwanz und die 5'-Kappe fehlen, werden die Proteine p33 und p92 direkt aus genomischer RNA translatiert. Wenn das Genom repliziert wird, entstehen zwei subgenomische[26] RNA-Moleküle, die als Messenger-RNA (mRNA) fungieren: Eines, aus dem das Kapsidprotein p41 (CP) exprimiert wird, und eines, aus dem die Proteine p19 und p22 exprimiert werden. Die überlappenden Gene p19 und p22 werden beide über Effekte des Leaky-Scannings übersetzt.[9] Zudem wurden mehrere Wechselwirkungen zwischen linear gut getrennten und weit auseinanderliegenden Bereichen des Genoms identifiziert mit funktioneller Bedeutung für eine effiziente Replikation.[22]
DI-Partikel
DI-Moleküle (englisch defective interfering RNA) sind RNAs, die aus dem viralen Genom hergestellt werden, die aber aufgrund von Verkürzungen und anderen Fehlern nicht in der Lage sind, Zellen (wie ein Virus) alleine zu infizieren. Stattdessen müssen sie (wie ein Satellitenvirus) mit einem intakten Helfervirus koinfiziert werden. TBSV-Infektionen produzieren unter experimentellen Bedingungen häufig eine signifikante Anzahl von DIs aus vollständigen und funktionalen Teilen des Genoms; allerdings wurde dieses Verhalten in freier Wildbahn nicht beobachtet. Ihre Produktion ist wahrscheinlich hostspezifisch. Infektionen, die zu DIs führen, haben in der Regel milderen Verlauf.[9][27][28]
siRNAs
Eine Variante der kleinen sRNAs ist die siRNA (englisch small interfering RNA). Diese entsteht, wenn die Pflanze Virus-Erbgut erkannt und zerstückelt. Zusammen mit bestimmten Proteinen (Wegweiser- oder Helferproteine) der Pflanze können diese sich an die Virus-RNA binden und diese zum Schutz der Pflanze inaktivieren; ein Effekt, der aber nur bei einer sehr kleinen Zahl von siRNAs möglich ist. 2019 angestellte Versuche, geeignete siRNAs mit Hilfen von Agrobacterium tumefaciens in die N. benthamiana-Pflanzen einzuschleußen, und so eine Art Immunisierung der Pflanzen zu bewirken, verliefen erfolgversprechend. Diese Bakterien sind in der Lage, die Zellwand der Pflanzenzellen zu überwinden und ins Innere einzudringen.[29][30][31]
Einzelnachweise
- ↑ a b c d ICTV: ICTV Taxonomy history: Carrot mottle mimic virus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ a b ICTV Master Species List 2018b v2 MSL #34v, März 2019
- ↑ P. Hopper, S. C. Harrison, R. T. Sauer: Structure of tomato bushy stunt virus. V. Coat protein sequence determination and its structural implications. In: Journal of Molecular Biology. 177. Jahrgang, Nr. 4, 25. August 1984, S. 701–713, doi:10.1016/0022-2836(84)90045-7, PMID 6481803 (englisch).
- ↑ a b Penka Kaitasowa, Violeta Kondakowa: Immunelektronenmikroskopischer Nachweis des Tomatenzwergbusch-Virus (tomato bushy stunt virus) in Erdbeeren: (Kurze Mitteilung), in: Arch. Phytopathol. Pflanzenschutz, Berlin 20. Dezember 1988, doi:10.1080/03235408909438914, online 12. September 2009
- ↑ Brian W. J. Mahy, Marc H. V. Van Regenmortel: Desk Encyclopedia of Plant and Fungal Virology. Academic Press, 2009, ISBN 978-0-12-375148-5, S. 445– (englisch, google.com [abgerufen am 26. August 2019]).
- ↑ a b ICTV: Taxonomy Browser.
- ↑ a b ICTV: Virus Metadata Resource (VMR).
- ↑ a b c Herman B. Scholthof: The Tombusvirus-encoded P19: from irrelevance to elegance. In: Nature Reviews Microbiology. 4. Jahrgang, Nr. 5, 6. März 2006, S. 405–411, doi:10.1038/nrmicro1395, PMID 16518419 (englisch).
- ↑ a b c d e f g h i j k l m n o Y. Yamamura, H. B. Scholthof: Tomato bushy stunt virus: a resilient model system to study virus-plant interactions. In: Molecular Plant Pathology. 6. Jahrgang, Nr. 5, 1. September 2005, S. 491–502, doi:10.1111/j.1364-3703.2005.00301.x, PMID 20565674 (englisch).
- ↑ a b c d e G.P. Martelli, M. Russo, L. Rubino: Tomato bushy stunt virus. In: Descriptions of Plant Viruses. Association of Applied Biologists, Dezember 2001, abgerufen am 26. August 2019.
- ↑ Fanny Balique, Hervé Lecoq, Didier Raoult, Philippe Colson: Can Plant Viruses Cross the Kingdom Border and Be Pathogenic to Humans? In: Viruses. 7. Jahrgang, Nr. 4, 20. April 2015, S. 2074–2098, doi:10.3390/v7042074, PMID 25903834, PMC 4411691 (freier Volltext) – (englisch).
- ↑ Integrated Pest Management Program. In: UC Pest Management Guidelines. University of California Division of Agriculture and Natural Resources, Dezember 2013, abgerufen am 26. August 2019 (englisch).
- ↑ ICTV Taxonomy History for Tomato bushy stunt virus. In: International Committee on Taxonomy of Viruses. Juli 2015, abgerufen am 16. Dezember 2016 (englisch).
- ↑ B. D. Harrison, J. T. Finch, A. J. Gibbs, M. Hollings, R. J. Shepherd, V. Valenta, C. Wetter: Sixteen groups of plant viruses. In: Virology. 45. Jahrgang, Nr. 2, 1. August 1971, S. 356–363, doi:10.1016/0042-6822(71)90336-9 (englisch).
- ↑ NCBI Taxonomy Browser: Tomato bushy stunt virus (Species)
- ↑ Michael G. Rossmann: Structure of viruses: a short history. In: Quarterly Reviews of Biophysics. 46. Jahrgang, Nr. 2, 1. Mai 2013, ISSN 0033-5835, S. 133–180, doi:10.1017/S0033583513000012, PMID 23889891 (englisch, cambridge.org).
- ↑ S. C. Harrison, A. J. Olson, C. E. Schutt, F. K. Winkler, G. Bricogne: Tomato bushy stunt virus at 2.9 Å resolution. In: Nature. 276. Jahrgang, Nr. 5686, 23. November 1978, S. 368–373, doi:10.1038/276368a0, PMID 19711552 (englisch).
- ↑ Alexander Hellemans, Bryan H. Bunch: The timetables of science: a chronology of the most important people and events in the history of science. Simon and Schuster, 1988, ISBN 978-0-671-62130-8, S. 656 (englisch, google.com [abgerufen am 26. August 2019]).
- ↑ Patrick Q. Hearne, David A. Knorr, Bradley I. Hillman, Thomas J. Morris: The complete genome structure and synthesis of infectious RNA from clones of tomato bushy stunt virus. In: Virology. 177. Jahrgang, Nr. 1, 1. Juli 1990, S. 141–151, doi:10.1016/0042-6822(90)90468-7 (englisch).
- ↑ Edward K. Wagner, Martinez J. Hewlett, David C. Bloom, David Camerini: Basic Virology. John Wiley & Sons, 2007, ISBN 978-1-4051-4715-6, S. 268– (englisch, google.com [abgerufen am 26. August 2019]).
- ↑ a b Baodong Wu, Jörg Grigull, Moriam O. Ore, Sylvie Morin, K. Andrew White: Global Organization of a Positive-strand RNA Virus Genome. In: PLOS Pathogens. 9. Jahrgang, Nr. 5, 23. Mai 2013, ISSN 1553-7374, S. e1003363, doi:10.1371/journal.ppat.1003363, PMID 23717202, PMC 3662671 (freier Volltext) – (englisch).
- ↑ a b Herman B. Scholthof, Karen-Beth G. Scholthof, Marjolein Kikkert, A. O. Jackson: Tomato Bushy Stunt Virus Spread Is Regulated by Two Nested Genes That Function in Cell-to-Cell Movement and Host-Dependent Systemic Invasion. In: Virology. 213. Jahrgang, Nr. 2, 1995, S. 425–38, doi:10.1006/viro.1995.0015, PMID 7491767 (englisch).
- ↑ Richard W. Jones, A.O. Jackson, Thomas J. Morris: Defective-interfering RNAs and elevated temperatures inhibit replication of tomato bushy stunt virus in inoculated protoplasts. In: Virology. 176. Jahrgang, Nr. 2, 1990, S. 539–45, doi:10.1016/0042-6822(90)90024-L, PMID 2345965 (englisch).
- ↑ Tomato bushy stunt virus (TBSV), p22, movement protein (IPR005332) – Short name: TBSV_p22, auf: InterPro – Protein sequence analysis & classification
- ↑ kürzer als das (RNA-)Genom
- ↑ Karen-Beth G. Scholthof, Herman B. Scholthof, Andrew O. Jackson: The Effect of Defective Interfering RNAs on the Accumulation of Tomato Bushy Stunt Virus Proteins and Implications for Disease Attenuation. In: Virology. 211. Jahrgang, Nr. 1, 1. August 1995, S. 324–328, doi:10.1006/viro.1995.1410, PMID 7645230 (englisch).
- ↑ NCBI: Defective interfering RNA-4 of tomato bushy stunt virus (TBSV-P DI-4) und Defective interfering RNA-5 of tomato bushy stunt virus (TBSV-P DI-5)
- ↑ Selma Gago-Zachert, Jana Schuck, Claus Weinholdt, Marie Knoblich, Vitantonio Pantaleo, Ivo Grosse, Torsten Gursinsky, Sven-Erik Behrens: Highly efficacious antiviral protection of plants by small interfering RNAs identified in vitro;
Claus Weinholdt, Ivo Grosse, Jana Schuck, Sven-Erik Behrens, Vitantonio Pantaleo, Torsten Gursinsky: Highly efficacious antiviral protection of plants by small interfering RNAs identified in vitro. In: Nucleic Acids Research, 21. August 2019; doi:10.1093/nar/gkz678, ResearchGate:335320410 (englisch). - ↑ MLU: Plant protection: researchers develop new modular vaccination kit, Pressemitteilung Nummer 128/2019 der Martin-Luther-Universität Halle-Wittenberg vom 21. August 2019
- ↑ Annika Röcker: Ausgesuchte Impfstoffe gegen Pflanzenkiller, auf: Spektrum.de vom 22. August 2019
Auf dieser Seite verwendete Medien
Autor/Urheber: Opabinia regalis, Lizenz: CC BY-SA 4.0
The RNA silencing suppressor p19 from the tomato bushy stunt virus, rendered from PDB ID 1R9F. The protein forms a dimer whose two monomers are shown in blue and green. The dimer forms an interaction surface that binds double-stranded RNAs of 19 to 21 base pairs. Plants use RNA silencing as a defense mechanism against viruses; the p19 protein serves as a viral counter-defense that specifically binds siRNAs and suppresses the silencing machinery.
Keqiong Ye, Lucy Malinina & Dinshaw J. Patel. Recognition of small interfering RNA by a viral suppressor of RNA silencing. Nature 426, 874-878 (18 December 2003). DOI: 10.1038/nature02213.
http://www.nature.com/nature/journal/v426/n6968/full/nature02213.htmlAutor/Urheber: ViralZone, SIB Swiss Institute of Bioinformatics: Tombusvirus, Lizenz: CC BY 4.0
Schemazeichnungen eines Virusteilchens der Familie Tombusviridae (Querschnitt und Seitenansicht)
Autor/Urheber: Chandres, Lizenz: CC BY-SA 3.0
Nicotiana benthamiana plants
Autor/Urheber: Opabinia regalis, Lizenz: CC BY-SA 4.0
The assembled capsid of tomato bushy stunt virus, the type species of the tombusvirus family. The capsid is composed of 180 individual monomers of the TBSV capsid protein (p41) arranged in a T=3 icosahedron. The three symmetrically distinct p41 monomers are colored in orange, blue, and green to highlight the quasi-equivalent arrangement of subunits.
From PDB ID 2TBV.
Hopper P, Harrison SC, Sauer RT. Structure of tomato bushy stunt virus. V. Coat protein sequence determination and its structural implications. J Mol Biol. 1984 Aug 25;177(4):701-13. PMID 6481803.Autor/Urheber: ViralZone, SIB Swiss Institute of Bioinformatics: Tombusvirus, Lizenz: CC BY 4.0
Genom der Spezies Tombusvirus




