Tertiärstruktur
Unter Tertiärstruktur versteht man in der Biochemie den übergeordneten räumlichen Aufbau von Proteinen, Nukleinsäuren oder anderen Makromolekülen, die aus einer Einzelnen oder mehreren Ketten bestehen.[1][2] Sie ist aus mehreren Elementen der Sekundärstruktur zusammengesetzt und ist, genau wie die Sekundärstruktur, bereits in der Primärstruktur codiert.
Der Tertiärstruktur übergeordnet ist die Quartärstruktur. Die hierarchische Einteilung in Primärstruktur, Sekundärstruktur, Tertiärstruktur und Quartärstruktur wurde 1952 durch Kaj Ulrik Linderstrøm-Lang vorgeschlagen.
Proteine
Insbesondere bei Proteinen ist die dreidimensionale Struktur charakteristisch und für die biologische Funktion unbedingt notwendig. Während beziehungsweise nach der Herstellung des Proteins durch Translation einer mRNA wird das Protein durch Proteinfaltung in die biologisch wirksame Form überführt. Dieser Vorgang wird u. a. durch Chaperone unterstützt.
Bei einem globulären Protein wird die energetisch treibende Kraft für die Faltung der einzelnen Sekundärstrukturelemente durch die Kauzmann-Regel beschrieben: die hydrophoben Bereiche sind im Inneren, während die hydrophilen und/oder geladenen Bereiche dem wässrigen Milieu zugewandt sind (siehe auch hydrophober Effekt). Die Sekundärstrukturen lagern sich zu definierten Elementen zusammen. Bekannte Proteinfaltungsklassen sind zum Beispiel das 3- oder 4-Helix-Bündel, das β-Fass, das β-Sandwich oder das TIM-Fass.
In die Stabilisierung von Tertiärstrukturen sind Disulfidbrücken (stärkste Bindung), Ionenbindungen, Wasserstoffbrücken und hydrophobe Wechselwirkungen (schwächste Bindung) involviert.
Die nächste übergeordnete Ebene ist die Quartärstruktur.
Nukleinsäuren
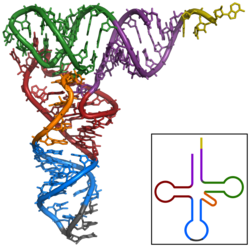
Nukleinsäuren können ebenfalls komplexe räumliche Strukturen einnehmen: tRNAs müssen für ihre Funktion in der korrekten Tertiärstruktur vorliegen.
Strukturaufklärung
Der Bereich der Biochemie, der sich mit der Aufklärung bzw. den Auswirkungen solcher Strukturen auseinandersetzt, heißt Strukturbiologie. Als Methoden der Strukturaufklärung dienen vorwiegend (Röntgen-)Kristallstrukturanalyse und mehrdimensionale NMR. Die Bioinformatik entwickelt Methoden bzw. Algorithmen zur Vorhersage der dreidimensionalen Struktur des Proteins aus dessen Aminosäuresequenz (Primärstruktur).
Einzelnachweise
- ↑ David L. Nelson: Lehninger Biochemie mit 131 Tabellen. 4., vollst. überarb. und erw. Auflage. Springer, Berlin 2009, S. 122.
- ↑ Eintrag zu tertiary structure. In: IUPAC (Hrsg.): Compendium of Chemical Terminology. The “Gold Book”. doi:10.1351/goldbook.T06282.
Auf dieser Seite verwendete Medien
Autor/Urheber: Holger87, Lizenz: CC BY-SA 3.0
Faltung des Proteins 1EFN mit Fokus auf die Tertiärstruktur
Autor/Urheber: Yikrazuul, Lizenz: CC BY-SA 3.0
Struktur der tRNAPhe aus Hefe. Dem Bild liegen die Daten aus PDB: 1ehz zu Grunde, es wurde mittels PyMOL gerendert.
- violett: Akzeptorstamm
- weinrot: D-Schleife
- blau: Anticodonschleife
- orange: variable Schleife
- grün: TPsiC-Schleife
- gelb: CCA-3' des Akzeptorstammes
- grau: Anticodon
Autor/Urheber: Yikrazuul, Lizenz: CC BY-SA 3.0
Struktur eines Pseudoknots aus pdb-Datei 1YMO. (A) sticks (B) Rückgrat