Tectiviridae
| Tectiviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 Röntgenkristallographie-basierte Struktur | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Tectiviridae | ||||||||||||||
| Links | ||||||||||||||
|
Die Virusfamilie Tectiviridae (lat. tectus: bedeckt) umfasst nur drei Gattungen Alphatectivirus, Betatectivirus und Gammatectivirus – die frühere Gattung Tectivirus wurde vom International Committee on Taxonomy of Viruses (ICTV) inzwischen aufgespalten.[2] Die Tectiviridae gehören zum Phylum Preplasmiviricota innerhalb des Realms Varidnaviria. Ihre Wirte sind Bakterien, was sie als Bakteriophagen klassifiziert.
Morphologie

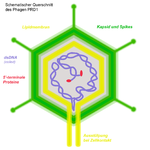
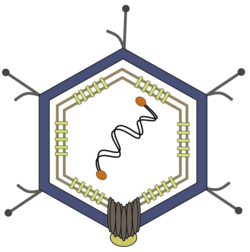
Dei den Tectividiae besitzen die Viruspartikel (Virionen) ein unbehülltes ikosaedrisches Kapsid mit einem Durchmesser von 66 nm. Dieses Kapsid besteht aus 240 Kapsomeren; die jeweils aus Trimeren des Kapsidproteins P3 bestehen. Das Kapsid besitzt an den Ikosaeder-Ecken jeweils einen fiberartigen Fortsatz (spike, Peplomer) von etwa 20 nm Länge, das aus zwei Proteinen (P2 und P5) aufgebaut und an der Basis an einem Pentonprotein verankert ist.
Innerhalb des Kapsids befindet sich ein Lipidmembran-Bläschen, das aus Virusproteinen und der Lipidmembran des Wirtsbakteriums besteht. Zur Ausschleusung des DNA-Genoms wird aus dem inneren Membranbläschen eine schwanzartige Röhre von ca. 60 × 10 nm durch das Kapsid ausgestülpt (daher die Namensgebung Tecti- für die Eigenschaft dieses „verdeckten“ Injektionsapparates). Diese ungewöhnliche Struktur eines Membranbläschens innerhalb eines ikosaedrischen Kapsids teilen sich die Tevtiviridae mit der Familie Corticoviridae.[3][4]
Genom
Das Genom der Tectiviridae ist unsegmentiert (monopartit) und besteht aus einem linearen, doppelsträngigen DNA-Molekül von etwa 15 kbp (Kilobasenpaaren) Länge. Dieses ist in verdrillter (coiled) Form innerhalb des Membranbläschens verpackt und die 5'-Enden beider Teilstränge sind kovalent mit Proteinen verknüpft. Der Prototyp der Familie, der Enterobacteria-Phage PRD1, kodiert für 25 Virusproteine.
Replikationszyklus


Die Replikation der Tectiviridae geschieht in folgenden Schritten:[3]
- Infektion: Der Phage heftet sich über sein Spike-Protein P2 an die Zielzelle. Daraufhin wird ein Proteinkomplex aus P2, P5, P31 und einem Teil des Kapsids (P3) freigesetzt, der eine Öffnung im Kapsid erzeugt.
- Die innere Kapsidmembran verwandelt sich in eine röhrenförmige Struktur (Schwanzstruktur), die durch das Loch im Kapsid ragt und die äußere Membran und Peptidoglykanschicht des Wirts durchdringt, um mit der Plasmamembran des Wirts zu verschmelzen und die virale DNA ins Zytoplasma des Wirts freizusetzen.[Anm. 1]
- Transkription und Translation der ersten („frühen“) Gene.
- Replikation der genomischen dsDNA.
- Transkription und Translation der restlichen („späten“) Gene.
- Assemblierung: Zusammenbau der noch leeren Kapsidhüllen der Nachkommenschaft.
- Die neu erzeugte genomische DNA wird in Kapsidhüllen verpackt.
- Die nun reifen Virionen werden durch Lyse aus der Zelle freigesetzt.
Wirte
Die Wirte der Tectiviridae sind mesophile Bakterien. Lediglich die Spezies Thermus Phage p37-14 befällt thermophile Bakterien und ist in vulkanischen Quellen und Geysieren zu finden.
Systematik
Besonders der Enterobacteria-Phage PRD1 zeigt äußerst viele Ähnlichkeiten zu Mitgliedern der Adenoviridae bezüglich der Kapsidstruktur (trimere hexagonale Kapsomere, Pentone, Fibern) und der Genomorganisation (lineare dsDNA, sogenannte Inverted Terminal Repeats, terminale Proteine, DNA-Polymerase Typ B). Diese Ähnlichkeiten deuten auf eine gemeinsame Herkunft, d. h. sie beruhen auf Homologie[6] Beide Familien wurden daher vom ICTV im März 2020 in dieselbe Klasse Tectiliviricetes gestellt.[7]
Nach ICTV Master Species List #38 vom März 2023:[8]
- Familie Tectiviridae

- Genus Betatectivirus[15][16]
- Spezies Bacillus-Virus AP50 (wiss. Betatectivirus AP50), mit Bacillus-Phage AP50
- Spezies Bacillus-Virus Bam35 (wiss. Betatectivirus Bam35), mit Bacillus-Phage Bam35 (Subtypen:[17] Bam35c,[12] pGIL01, pGIL02)
- Spezies Bacillus-Virus GIL16 (wiss. Betatectivirus GIL16), mit Bacillus-thuringiensis-Phage GIL16c alias Bacillus-Phage ΦNS11[13]
- Spezies Betatectivirus sato
- Spezies Betatectivirus sole
- Spezies Bacillus-Virus Wip1 (wiss. Betatectivirus Wip1)
- Genus Betatectivirus[15][16]
- Genus Gammatectivirus[18]
- Spezies Gluconobacter-Virus GC1 (wiss. Gammatectivirus GC1)
- Genus Gammatectivirus[18]


- Genus Deltatectivirus[19]
- Genus Epsilontectivirus
- vorgeschlagene Mitglieder der Familie ohne Gattungszuweisung:
- frühere Mitglieder:
Anmerkungen
- ↑ Vergleichbare Mechanismen findet man bei den Caudoviricetes und der Gattung Chlorovirus.
Literatur
- C. M. Fauquet, M. A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses, London, San Diego, 2004
- David M. Knipe, Peter M. Howley et al. (Hrsg.): Fields' Virology, 4. Auflage, Philadelphia 2001
Weblinks
- NCBI Taxonomy Browser: Tectiviridae (species).
Einzelnachweise
- ↑ a b c d e ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ↑ ICTV: Master Species List 2018b.v2 #34, März 2019
- ↑ a b c SIB: Tectiviridae, auf: Expasy ViralZone
- ↑ a b c ICTV:dsDNA Viruses – Textiviridae ( des vom 24. April 2019 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis., ICTV 9th Report (2011)
- ↑ Sari Mäntynen, Lotta-Riina Sundberg, Hanna M. Oksanen, Minna M. Poranen: Half a Century of Research on Membrane-Containing Bacteriophages: Bringing New Concepts to Modern Virology.Iin: MDPI: Viruses, Band 11, Nr. 1, Section Bacterial Viruses, 2019, S. 76; doi:10.3390/v11010076, PDF (englisch).
- ↑ Patrick Forterre: Evolution: Die wahre Natur der Viren, in: Spektrum August 2017, S. 37 (Online-Artikel vom 19. Juli 2017).
- ↑ a b ICTV: ICTV Taxonomy history: Pseudomonas virus PRD1, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ ICTV: Taxonomy Browser, abgerufen am 9. November 2019
- ↑ SIB: Alphatectivirus, auf: ViralZone
- ↑ NCBI: Alphatectivirus (genus)
- ↑ NCBI: Genomsequenz und Proteine des Phagen PRD1
- ↑ a b c Natalya Yutin, Disa Bäckström, Thijs J. G. Ettema, Mart Krupovic, Eugene V. Koonin: Vast diversity of prokaryotic virus genomes encoding double jelly-roll major capsid proteins uncovered by genomic and metagenomic sequence analysis, in: Virol Jv., Band 15; 10. April 2018; doi:10.1186/s12985-018-0974-y, PMC 5894146 (freier Volltext), PMID 29636073 (englisch) .
- ↑ a b Céline Verheust, Nadine Fornelos, Jacques Mahillon: GIL16, a New Gram-Positive Tectiviral Phage Related to the Bacillus thuringiensis GIL01 and the Bacillus cereus pBClin15 Elements, in: J Bacteriol. 187(6), März 2005, S. 1966–1973, doi:10.1128/JB.187.6.1966-1973.2005, PMC 1064052 (freier Volltext) (englisch).
- ↑ NCBI: Enterobacteria phage PR772 (no rank)
- ↑ SIB: Betatectivirus, auf: ViralZone
- ↑ NCBI: Betatectivirus (genus)
- ↑ NCBI: Bacillus virus Bam35 (species)
- ↑ NCBI: Gammatectivirus (genus)
- ↑ a b c d e f Steven M. Caruso, Tagide N. deCarvalho, Anthony Huynh, George Morcos, Nansen Kuo, Shabnam Parsa, Ivan Erill: A Novel Genus of Actinobacterial Tectiviridae. In: MDPI: Viruses, Band 11, Nr. 12, 7. Dezember 2019; doi:10.3390/v11121134, ResearchGate (englisch).
- ↑ NCBI: Rhodococcus phage Toil (species)
- ↑ NCBI Taxonomy Browser: unclassified Tectiviridae (list).
- ↑ Proposal 2017.013B
Auf dieser Seite verwendete Medien
Autor/Urheber: International Committee on Taxonomy of Viruses (ICTV), Lizenz: CC BY-SA 4.0
Röntgenkristallographie-basierte Struktur des Virions von Enterobacterio-Phage PRD1 (pseudo T=25), gesehen entlang der zweifachen Symmetrieachse.
Autor/Urheber: International Committee on Taxonomy of Viruses (ICTV), Lizenz: CC BY-SA 4.0
Diagram of the life cycle of Enterobacteria phage PRD1
Autor/Urheber: Steven M. Caruso, Tagide N. deCarvalho, Anthony Huynh, George Morcos, Nansen Kuo, Shabnam Parsa, Ivan Erill, Lizenz: CC BY-SA 4.0
Representative TEM images of CsCl-purified Streptomyces phage Forthebois from crude lysate; (d) Forthebois with nanotube from crude lysate. Black arrowheads indicate lipid membrane. White arrowheads indicate collar-like structure. Scale bar = 100 nm for both panels.
Autor/Urheber: Gleiberg, Lizenz: CC BY-SA 2.0 de
Schematischer Querschnitt durch ein Viruspartikel von Enterobacteria-Phage PRD1 (Familie Tectiviridae) zum Beginn des Eintritts in die Wirtszelle.
Autor/Urheber: ViralZone, SIB Swiss Institute of Bioinformatics: https://viralzone.expasy.org - see also permission note at File:T4likevirus virion.jpg, Lizenz: CC BY 4.0
Schemazeichnung: Virion der Familie Tectiviridae (Querschnitt und Aufsicht)
Autor/Urheber: Sari Mäntynen, Lotta-Riina Sundberg, Hanna M. Oksanen, Minna M. Poranen, Lizenz: CC BY-SA 4.0
Entry mechanisms of membrane-containing phages: Enterobacteria phage PRD1. Upon attachment to the host receptor, the internal membrane vesicle of PRD1 transforms into a proteo-lipidic tube, which traverses the cell envelope and provides a conduit for transferring the linear dsDNA genome into the cytoplasm. CM, cytoplasmic membrane; PG, peptidoglycan layer; OM, outer membrane of the envelope in Gram-negative host bacterium.
Autor/Urheber: International Committee on Taxonomy of Viruses (ICTV), Lizenz: CC BY-SA 4.0
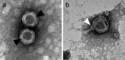
Negative stain electron micrograph of Enterobacteria phage PRD1 particles. The inset shows a particle with a protruding membranous tail-like structure. The bar represents 100 nm.
Autor/Urheber: International Committee on Taxonomy of Viruses (ICTV), Lizenz: CC BY-SA 4.0
Schematic picture of an Enterobacteria phage PRD1 virion.
Autor/Urheber: Steven M. Caruso, Tagide N. deCarvalho, Anthony Huynh, George Morcos, Nansen Kuo, Shabnam Parsa, Ivan Erill, Lizenz: CC BY-SA 4.0
Representative TEM images of (a) CsCl-purified Streptomyces phage WheeHeim; (b) WheeHeim with nanotube from crude lysate. Scale bar = 100 nm for both panels.
![Details eines Virions von Enterobacteria-Phage PRD1[4]](http://upload.wikimedia.org/wikipedia/commons/thumb/e/ed/F30-01c-9780123846846.png/150px-F30-01c-9780123846846.png)