Squalen-Monooxygenase
| Squalen-Monooxygenase | ||
|---|---|---|
| Eigenschaften des menschlichen Proteins | ||
| Masse/Länge Primärstruktur | 574 Aminosäuren | |
| Kofaktor | FAD | |
| Bezeichner | ||
| Gen-Name | SQLE | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 1.14.99.7, Monooxygenase | |
| Reaktionsart | Addition eines Sauerstoffatoms | |
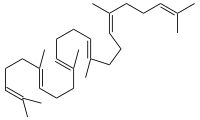
| Substrat | Squalen + red.Acc./H2 + O2 | |
| Produkte | Squalenepoxid + ox.Acc. + H2O | |
| Vorkommen | ||
| Homologie-Familie | Hovergen | |
| Übergeordnetes Taxon | Eukaryoten, wenige Bakterien[1] | |
Die Squalen-Monooxygenase (Squalenepoxidase, SE) ist das Enzym, das die Oxidation von Squalen zu S-Squalen-2,3-epoxid katalysiert. Diese Reaktion findet in Eukaryoten und manchen Bakterien statt; in Tieren ist sie Teil der Cholesterinbiosynthese. Im Mensch ist SE in den Mikrosomen lokalisiert.[1][2][3] Die "-fin" Antimykotika, wie Terbinafinoder Amorolfin, hemmen die Squalenepoxidase und finden Anwendung z. B. bei der Bekämpfung von Nagelpilz.
Katalysierte Reaktion
Squalen wird zu (S)-Squalen-2,3-epoxid oxidiert, der Acceptor wird dehydriert.
Weblinks
- Jassal / reactome: Squalene is oxidized to its epoxide
Einzelnachweise
- ↑ a b InterPro-Eintrag
- ↑ Lamb DC, Jackson CJ, Warrilow AG, Manning NJ, Kelly DE, Kelly SL: Lanosterol biosynthesis in the prokaryote Methylococcus capsulatus: insight into the evolution of sterol biosynthesis. In: Mol. Biol. Evol. 24. Jahrgang, Nr. 8, August 2007, S. 1714–21, doi:10.1093/molbev/msm090, PMID 17567593.
- ↑ UniProt Q14534
Auf dieser Seite verwendete Medien
Struktur von (S)-Squalen-2,3-epoxid
Struktur von Squalen