Peplomer
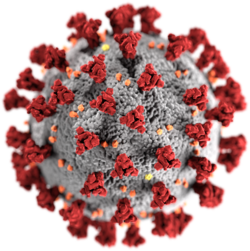
Ein Peplomer (von altgriechisch πέπλοςpeplos, deutsch ‚Gewand‘, ‚Decke‘ und altgriechisch μέροςmeros, deutsch ‚Teil‘), S-Glykoprotein (S für englisch spikes) oder Spike-Protein[1] ist eine nach außen ragende Proteinstruktur einer Virushülle. Spike-Proteine werden auf einer elektronenmikroskopischen Abbildung sichtbar. Sie haben eine Funktion bei der Bindung an die Wirtszelle.
Begriff
Heute wird der Begriff Peplomer nur noch bei wenigen Virengruppen verwendet, bei denen die Hüllproteine besonders große und charakteristische Strukturen ausbilden, z. B. bei den Orthomyxoviridae. Beim Virion von SARS-CoV-2 ist statt Peplomer der Begriff Spikeprotein (englisch spike ‚Dorn‘, ‚Stachel‘) üblich: Dieser lässt jedoch eine spitze Struktur vermuten, was bei den genannten Hüllstrukturen nicht gegeben ist; sie sind außen rund, abgeflacht oder knopfförmig. Beide Begriffe, Peplomer und Spikes, beschreiben jedoch nur eine morphologisch sichtbare Struktur und sind nicht identisch mit den Begriffen Membranprotein oder Hüllprotein; viele Viren bilden keine prominenten Strukturen aus.
Aufbau
Peplomere sind aus viralen Membranproteinen aufgebaut, die mit einer Proteindomäne in der Lipidmembran verankert sind (Transmembranäre Domäne). Eine weitere Domäne ragt nach außen und ist meistens durch die Anbindung verschiedener Zuckerreste glykosyliert. Diese äußere Domäne der Peplomere vermittelt die Bindung an die Oberflächenrezeptoren der Zielzelle und damit den Eintritt des Virus in die Zelle. Ebenfalls definieren die äußeren Epitope der Peplomere die serologischen Eigenschaften des Virus und die Interaktion mit Antikörpern des Wirtes. Ein Peplomer kann aus einzelnen oder zu Dimeren oder Trimeren zusammengelagerten Membranproteinen zusammengesetzt sein, wobei die Untereinheiten identisch (Homomer) oder verschieden (Heteromer) sein können. Die Zusammensetzung eines Peplomers kann formalisiert dargestellt werden, beispielsweise ein einfaches Homodimer aus zwei verschiedenen Hüllproteinen E1 und E2 schreibt man als [E1, E2], treten diese Dimer wiederum zu größeren Gruppen zusammen, z. B. Trimeren, so ist dies formalisiert: [E1, E2]3. Eine Peplomer mit der Formel [E1, E2, E3]2 stünde für zwei zusammengelagerte Trimere aus drei verschiedenen Hüllproteinen, die zusammen ein Hexamer bilden.
Literatur
- F. Fenner et al.: The Biology of Animal Viruses. 2. Auflage. New York/London 1968, ISBN 0-12-253040-3, S. 5 f. (englisch).
- D. J. Garwes et al.: Identification of epitopes of immunological importance on the peplomer of porcine transmissible gastroenteritis virus. In: Adv Exp Med Biol. Band 218, 1987, S. 509–515, PMID 2449047 (englisch).
- H. G. Niesters et al.: The peplomer protein sequence of the M41 strain of coronavirus IBV and its comparison with Beaudette strains. In: Virus Res. Band 5, Nr. 2–3, 1986, S. 253–263, PMID 2429473 (englisch).
Einzelnachweise
- ↑ SARS-CoV-2: Virologische Basisdaten sowie Virusvarianten. (Stand: 11. Dezember 2020). In: Website des Robert Koch-Instituts: [1]. Robert Koch-Institut, 25. Januar 2021, abgerufen am 7. Februar 2021 (Siehe z. B. Absatz 2).
Auf dieser Seite verwendete Medien
Diese Abbildung, die in den Zentren für die Kontrolle und Prävention von Krankheiten (CDC) erstellt wurde, zeigt die ultrastrukturelle Morphologie von Coronaviren. Beachten Sie die Spitzen, die äußere Oberfläche des Virus schmücken und bei elektronenmikroskopischer Betrachtung das Aussehen einer das Virion umgebenden Korona vermitteln. Ein neuartiges Coronavirus namens schweres akutes respiratorisches Syndrom Coronavirus 2 (SARS-CoV-2) wurde als Ursache für einen Ausbruch einer Atemwegserkrankung identifiziert, die erstmals 2019 in Wuhan, China, entdeckt wurde. Die durch dieses Virus verursachte Krankheit wurde als Coronavirus-Krankheit bezeichnet 2019 (COVID-19).
