Sorbitdehydrogenase
| Sorbitdehydrogenase | ||
|---|---|---|
 | ||
| Bändermodell des Tetramer, NAD und Zink als Kalotten, nach PDB 1PL8 | ||
| Eigenschaften des menschlichen Proteins | ||
| Masse/Länge Primärstruktur | 356 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Homotetramer | |
| Kofaktor | Zink, NAD | |
| Bezeichner | ||
| Gen-Name | SORD | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 1.1.1.14, Oxidoreduktase | |
| Reaktionsart | Oxidation | |
| Substrat | Sorbit + NAD+ | |
| Produkte | Fructose + NADH | |
| Vorkommen | ||
| Übergeordnetes Taxon | Lebewesen | |
| Orthologe | ||
| Mensch | Hausmaus | |
| Entrez | 6652 | 20322 |
| Ensembl | ENSG00000140263 | ENSMUSG00000027227 |
| UniProt | Q00796 | Q64442 |
| Refseq (mRNA) | NM_003104 | NM_146126 |
| Refseq (Protein) | NP_003095 | NP_666238 |
| Genlocus | Chr 15: 45.02 – 45.08 Mb | Chr 2: 122.23 – 122.27 Mb |
| PubMed-Suche | 6652 | 20322 |
Sorbitdehydrogenase (Sdh) (Gen: SORD) ist der Name für Enzyme, die Sorbit zu Fructose umwandeln. Dies ist der zweite Schritt im Polyolweg, der von Zellen benutzt wird, um ohne ATP-Verbrauch Fructose aus Glucose herzustellen. Jedes Lebewesen benutzt Sdh. In Säugetieren ist das Enzym in allen Gewebetypen aktiv.[1][2]
Es gibt Hinweise darauf, dass Polymorphismen im SORD-Gen mit Retikulopathie bei Diabetes mellitus Typ 2 assoziiert sind. Sdh-Hemmer (oder auch Aldosereduktase-Hemmer) können das Auftreten neurologischer und ophthalmologischer Probleme bei Diabetes verhindern und sind daher interessant als potenzielle Arzneistoffe.[3][4][5] Darüber hinaus können SORD Mutationen auch eine Neuropathie vom Charcot-Marie-Tooth Typ 2 auslösen[6]
Struktur und Funktion
Sdh bildet ein komplexes Tetramer, das durch ein Netz von Wasserstoffbrücken seine spezielle Form erhält. Dieser Mechanismus hat sich bei allen Säugetieren erhalten und ist essenziell für die Funktion des Enzyms.[7]
Die katalysierte Reaktion:
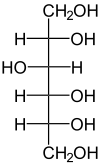
D-Sorbit wird zu D-Fructose dehydriert und umgekehrt.
Einzelnachweise
- ↑ El-Kabbani O, Darmanin C, Chung RP: Sorbitol dehydrogenase: structure, function and ligand design. In: Curr. Med. Chem.. 11, Nr. 4, Februar 2004, S. 465–76. doi:10.2174/0929867043455927. PMID 14965227.
- ↑ UniProt Q00796
- ↑ Szaflik JP, Majsterek I, Kowalski M, et al: Association between sorbitol dehydrogenase gene polymorphisms and type 2 diabetic retinopathy. In: Exp. Eye Res.. 86, Nr. 4, April 2008, S. 647–52. doi:10.1016/j.exer.2008.01.009. PMID 18289528.
- ↑ Schmidt RE, Dorsey DA, Beaudet LN, et al: A potent sorbitol dehydrogenase inhibitor exacerbates sympathetic autonomic neuropathy in rats with streptozotocin-induced diabetes. In: Exp. Neurol.. 192, Nr. 2, April 2005, S. 407–19. doi:10.1016/j.expneurol.2004.12.018. PMID 15755558.
- ↑ Kador PF, Inoue J, Blessing K: Anticataract activity of analogs of a sorbitol dehydrogenase inhibitor. In: J Ocul Pharmacol Ther. 20, Nr. 4, August 2004, S. 333–44. doi:10.1089/1080768041725281. PMID 15321028.
- ↑ Andrea Cortese, Yi Zhu, Adriana P. Rebelo, Sara Negri, Steve Courel: Biallelic mutations in SORD cause a common and potentially treatable hereditary neuropathy with implications for diabetes. In: Nature Genetics. Band 52, Nr. 5, Mai 2020, ISSN 1546-1718, S. 473–481, doi:10.1038/s41588-020-0615-4 (nature.com [abgerufen am 2. Dezember 2020]).
- ↑ Hellgren M, Kaiser C, de Haij S, Norberg A, Höög JO: A hydrogen-bonding network in mammalian sorbitol dehydrogenase stabilizes the tetrameric state and is essential for the catalytic power. In: Cell. Mol. Life Sci.. 64, Nr. 23, Dezember 2007, S. 3129–38. doi:10.1007/s00018-007-7318-1. PMID 17952367.
Weblinks
Auf dieser Seite verwendete Medien
Struktur von D-Sorbitol
ribbon model of sorbitol dehydrogenase (Sdh) tetramer with NAD and zinc as balls, from PDB 1PL8. Ref.: T.A.Pauly et al. (2003). X-ray crystallographic and kinetic studies of human sorbitol dehydrogenase.. Structure, 11, 1071-1085. PMID 12962626 [DOI: 10.1016/S0969-2126(03)00167-9]