Serratia marcescens
| Serratia marcescens | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
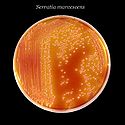
 Kolonien von Serratia marcescens auf einem Agargel in einer Petrischale | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Serratia marcescens | ||||||||||||
| Bizio 1823 |

Serratia marcescens ist ein Bakterium, das zur Gattung Serratia in der Familie der Enterobacteriaceae (Enterobakterien) gehört.
Merkmale
Zur Art Serratia marcescens gehören Gram-negative, fakultativ anaerobe, sich aktiv mit peritrich angeordneten Geißeln bewegende, stäbchenförmige Bakterien. Bis zur Entdeckung einer Unterart 2003 ging man davon aus, dass Serratia marcescens – wie alle gramnegativen Bakterien – keine Endosporen bildet. Dies wurde durch Serratia marcescens subsp. sakuensis widerlegt, die Endosporen ließen sich im Elektronenmikroskop erkennen, sie überleben eine gängige Hitzebehandlung, und die für die Sporenhülle typische Dipicolinsäure ist nachweisbar.[1] Das International Committee on Systematics of Prokaryotes konnte bislang jedoch noch keine Sporen in dem Isolat feststellen und so auch nicht die These bestätigen, dass ein Gentransfer mit Bacillus spp. im Abwasser stattgefunden habe könnte.[2]
Serratia marcescens produziert die hydrolytischen Enzyme DNase, Chitinase und Lipase und kann Gelatine hydrolysieren. Sie bildet die Restriktionsendonuklease SmaI, die in der Molekularbiologie eingesetzt wird.
Sie kommen ubiquitär im Boden, Wasser, auf Tieren und Pflanzen vor und sind in der Regel harmlose Saprobionten (Destruenten organischer Stoffe). Die Bakterien können problemlos auf gängigen Nährmedien kultiviert werden. Sie bilden teilweise das rote Pyrrol-Pigment Prodigiosin (von lateinisch prodigium = Wunderzeichen, siehe unter Historisches), wodurch die Kolonien rot gefärbt sind (siehe Bild). Dies kommt bei aus der Umwelt isolierten Stämmen häufiger vor als bei klinischen Isolaten von Patienten.[3]
Das Genom von Serratia marcescens wurde vom Sanger Institute (Cambridge, Großbritannien) vollständig sequenziert. Es besteht aus einem einzigen in sich geschlossenen DNA-Strang (Bakterienchromosom) und hat eine Größe von 5,1 MBp.
Systematik
Serratia marcescens ist die Typusart der Gattung Serratia. Diese steht in der Familie der Enterobacteriaceae, einer großen Gruppe gramnegativer Bakterien. Die Art wurde 1823 von Bartolomeo Bizio als Bacillus marcescens erstbeschrieben. Der vollständige Name des Synonyms lautet Bacillus marcescens (Bizio 1823) Trevisan in de Toni and Trevisan 1889.[4] Weitere Synonyme werden in den einschlägigen Listen nicht genannt.[4][5] Die zuvor taxonomisch nicht genau bestimmten Isolate „Enterobacteriaceae Bakterium KO4“ und „Pantoea sp. NAB7“ sind der Art S. marcescens zugeordnet worden.[5]
2003 wurde die Art in zwei Unterarten (Subspezies) aufgeteilt:[4]
- Serratia marcescens subsp. marcescens (Bizio 1823)Ajithkumar et al. 2003
- Serratia marcescens subsp. sakuensisAjithkumar et al. 2003
Diese Subspezies wurde im Belebtschlamm einer Abwasserbehandlungsanlage gefunden.[1]
Humanmedizinische Bedeutung
Serratia marcescens ist ein fakultativer Krankheitserreger (opportunistischer Erreger). Sie kann bei immungeschwächten Personen folgende Krankheiten verursachen: Harnwegsentzündungen, Sepsis, Pneumonie, Endokarditis, Meningitis, Osteomyelitis.
Die beiden Subspezies von Serratia marcescens werden durch die Biostoffverordnung in Verbindung mit der TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466 der Risikogruppe 2 zugeordnet.[6] Sie gelten folglich als Bakterien, die „eine Krankheit beim Menschen hervorrufen können und eine Gefahr für Beschäftigte darstellen könnten; eine Verbreitung in der Bevölkerung ist unwahrscheinlich; eine wirksame Vorbeugung oder Behandlung ist normalerweise möglich“.(§ 3 Biostoffverordnung). Weiterhin ist für S. marcescens subsp. marcescens angegeben, dass sie pathogen für Menschen und Wirbeltiere ist, dass es aber normalerweise zu keiner Übertragung zwischen den Wirtsgruppen kommt, es sich also nicht um einen Zoonoseerreger handelt.[6]
Früher wurde das Bakterium als vollständig apathogen (nicht krank machend) betrachtet, erst in den letzten Jahrzehnten wurde die zunehmende Bedeutung als Erreger nosokomialer Krankheiten erkannt. Zahlreiche Ausbrüche innerhalb von Gesundheitseinrichtungen wurden beschrieben, sowohl bei Erwachsenen als auch auf Neugeborenen- und Kinderintensivstationen.[2]
Ausbreitung
Die Bakterien kommen überall im Boden, Wasser, auf Tieren und Pflanzen vor. Die Infektion kann daher aus der Umgebung, aber auch von Mensch zu Mensch durch direkten Kontakt, Tröpfcheninfektion oder medizinisches Personal erfolgen. Bei gesunden, immunkompetenten Menschen führt der Kontakt mit dem Erreger üblicherweise nicht zur Entstehung einer Krankheit.
Häufigkeit der Erkrankungen
Serratia marcescens ist ein seltener Krankheitserreger. Harnwegsentzündungen werden in etwa 2 % der Fälle durch dieses Bakterium verursacht. Bei Pneumonie und Sepsis bewegt sich der Anteil um 1 %. Diese Zahlen gelten für ambulant erworbene Infektionen, bei nosokomialen Infektionen liegt die Rate tendenziell etwas höher.
Diagnostik
Die Diagnose erfolgt durch Kultivierung des Erregers aus Blut- und Urinkulturen, Bronchialsekret oder bronchoalveolärer Lavage. Ein gut geeignetes Nährmedium ist z. B. MacConkey-Agar. Nach Anlegen einer Reinkultur kann die Spezies am einfachsten mit biochemischen Methoden („Bunte Reihe“) bestimmt werden.
Therapie
Die Behandlung einer durch Serratia marcescens verursachten Krankheit sollte, wann immer möglich, nach Resistenzprüfung (Antibiogramm) erfolgen. Die „kalkulierte“ Therapie kann z. B. mit einem Fluorchinolon oder einem Carbapenem begonnen werden, ggf. in Kombination mit einem Aminoglykosid, z. B. Gentamicin. Aufgrund der chromosomal codierten β-Laktamase vom Typ AmpC besitzen Bakterien der Art Serratia marcencens eine natürliche Resistenz gegenüber einer Reihe von β-Laktam-Antibiotika (Amoxicillin ± Clavulansäure, Ampicillin ± Sulbactam, Cephalosporine der ersten und zweiten Generation). Darüber hinaus ist auf dem Chromosom von S. marcescens ein Aminoglykosid-spaltendes Enzym vom Typ AAC(6')-Ic codiert, das die Wirksamkeit aller Aminoglykoside außer Gentamicin, Streptomycin und Arbekacin beeinträchtigen kann. Außerdem bestehen natürliche Resistenzen gegenüber Colistin und Nitrofurantoin.[7]
Problematisch für die Therapie ist auch die Fähigkeit von S. marcescens, nach Erwerb entsprechender Plasmide extended-spectrum-β-Lactamasen (ESBL) zu produzieren.[2] Damit können die Bakterien Antibiotika vom β-Lactam-Typ (z. B. alle Penicilline und Cephalosporine) unwirksam machen.
Veterinärmedizinische Bedeutung
Nachgewiesen ist, dass Serratia marcescens bei Steinkorallen der Art Acropora palmata die White pox Krankheit auslöst.[8] Gentests und Laborversuche ergaben, dass dieses für Korallen tödliche Bakterium nicht von anderen Tieren stammt, sondern aus Abwässern des Menschen.[9]
Von S. marcescens und weiteren Arten der Gattung wird seit den 1960er-Jahren angenommen, dass sie für verschiedene Insekten pathogen sind, allerdings bei nur moderater Virulenz. So wurden u. a. bestimmte Bakterienstämme, die als S. marcescens Biotyp A4b bezeichnet werden, mehrfach bei erkrankten Larven der Bienenart Apis mellifera (Westliche Honigbiene) gefunden. Krankheitssymptome treten erst auf, wenn die Bakterien in das Hämocoel (eine sekundäre Leibeshöhle, vergleiche auch Zölom) gelangen, was schließlich zu einer tödlich verlaufenden Septikämie („Blutvergiftung“) führt.[10][11] Es wird vermutet, dass die Bakterien durch die Aufnahme pflanzlicher Nahrung in den Verdauungstrakt und bei spontan auftretenden Rissen in der Darmwand in das Hämocoel gelangen. Bei Bienen erfolgt die Infektion möglicherweise über die Tracheen, und weil die Krankheit oft bei Völkern auftritt, die von Milben befallen sind, könnten die Parasiten als Überträger fungieren.[11] Die Hämolymphe der Insekten ist normalerweise bakterizid für Nicht-Pathogene, kann aber die Vermehrung von Serratia und einigen anderen Krankheitserregern nicht verhindern. Bakterielle Enzyme (Lecithinase, Proteinase und Chitinase) werden hierbei als Virulenzfaktoren angesehen. Für einige Insektentaxa (u. a. Heuschrecken und Baumwollkapselkäfer) sind sogar experimentell LD50-Werte in Bezug auf die Zellzahlen, die in das Hämocoel eingebracht werden müssen, bestimmt worden. Wenn die Bakterien mit der Nahrung aufgenommen werden, ist der LD50-Wert jedoch höher.[10]
Der wissenschaftliche Mitarbeiter des US-Landwirtschaftsministeriums Carlton Earl Burnside isolierte im Jahre 1928 aus Bienen mit Septikämie ein Bakterium, das er als „Bacillus apisepticus“ erstbeschrieb. 1959 wurde dieses Bakterium von den Kanadiern Landerkin und Katznelson in die Gattung Pseudomonas gestellt,[11] und bis heute wird es in der Imker-Fachliteratur meist als „Pseudomonas apisepticus (Burnside, 1928)“ aufgeführt. Der Tscheche Oleg Lysenko stellte hingegen 1963 fest, dass „P. apisepticus“ identisch mit S. marcescens sei. 1979 wurde ein Stamm von „P. apisepticus“ aus dem Microbial Research Institute in Ottawa als S. marcescens Biotyp A4a bestimmt.[12] Bereits zum Ende der 1950er Jahre identifizierten Lysenko und der Engländer J. P. Stevenson Bakterien, die bei Seidenspinnerraupen bzw. Wüstenheuschrecken eine Septikämie verursachen, als eine nicht-pigmentierte Form von S. marcescens.[13] Die Erstbeschreibung der verschiedenen unpigmentierten insektenpathogenen Bakterienformen, mit Ausnahme von S. marcescens selbst, entspricht jedoch nicht den heutigen Anforderungen des Internationalen Codes der Nomenklatur von Bakterien. Unter anderem existiert heute kein Originalmaterial mehr, anhand dessen sich feststellen ließe, ob es sich bei den in den 1950ern und 60ern als S. marcescens identifizierten, ursprünglich anderen Arten zugewiesenen Stämmen tatsächlich um die gleichen Formen handelte, die einst unter dem Namen ebenjener anderer Arten beschrieben worden waren. Deshalb werden heute weder „Bacillus apisepticus“ bzw. „Pseudomonas apisepticus“ noch die mit den Arbeiten Lysenkos und Stevensons verbundenen Namen „Bacillus noctuarum“ bzw. „Pseudomonas noctuarum“ sowie „Aeromonas margarita“ offiziell als Synonyme von Serratia marcescens geführt (vergleiche Abschnitt Systematik).
Anwendung
Das Enzym Sma1 stammt aus Serratia und wird in der Biochemie und Molekularbiologie zum Schneiden von doppelsträngiger DNA an einer bestimmten Sequenz verwendet.
Historisches
Serratia marcescens wurde 1819 auf verdorbener Polenta von dem Pharmazeuten Bartolomeo Bizio aus Padua entdeckt. Er hielt die Kolonien des Bakteriums auf der Polenta für einen Pilz und benannte ihn Serratia nach seinem Physiklehrer, dem Physiker und Dampfschiffkonstrukteur Serafino Serrati aus Florenz, und wegen seiner weichen Konsistenz marcescens (lateinisch marcescere = erschlaffen). Christian Gottfried Ehrenberg benannte ohne Kenntnis von Bizios Entdeckung das Bakterium 1848 Monas prodigiosa.[14]
Man schreibt Serratia marcescens das „Wunder von Bolsena“ (1263) und andere Blutwunder zu, die seit dem 13. Jahrhundert bis in die Reformationszeit hinein sehr populär waren. Auf den seit dem 12. Jahrhundert für die lateinische Messfeier verwendeten ungesäuerten Hostien kann Serratia marcescens gut gedeihen und rote Verfärbungen bewirken, was den falschen Eindruck eines Hostienwunders erweckt. Der Alternativname Bacterium prodigiosum und die Bezeichnung des von ihm gebildeten Farbstoffs Prodigiosin gehen auf den Zusammenhang mit diesen scheinbaren Wundern zurück: lateinisch prodigium, „Wunderzeichen“.[15]
Im September 1950 versprühten zwei US-U-Boote an der Küste von San Francisco Serratia marcescens, um herauszufinden, wie viele Bewohner sich damit infizieren würden und wie es sich verbreitet. Das Bakterium ist für gesunde Menschen ungefährlich, greift jedoch immungeschwächte Personen an. In Krankenhäusern kam es zu Todesfällen, die auf Infektion mit den versprühten Erregern zurückgeführt werden konnten.[16] Derartige Feldversuche wurden in der Zeit des Kalten Krieges im Rahmen der Entwicklung von Biowaffen durchgeführt.
2007 benutzen Wissenschaftler Bakterien dieser Art, um kleine Lasten durch eine Flüssigkeit zu bewegen. Ziel dieser Forschung ist es, Transportmechanismen für Mikromaschinen zu entwickeln.[17]
Siehe auch
- Filtermembran: S. marcescens wird zur Prüfung von Filtermembranen der Porengröße 0,45 µm herangezogen.
- Blutwunder
Quellen
Literatur
- Francine Grimont, Patrick A. D. Grimont: The Genus Serratia (Chapter 3.3.11). In: Martin Dworkin, Stanley Falkow, Eugene Rosenberg, Karl-Heinz Schleifer, Erko Stackebrandt (Hrsg.): The Prokaryotes. A Handbook on the Biology of Bacteria. 3. Auflage. Volume 6: Proteobacteria: Gamma Subclass. Springer-Verlag, New York 2006, ISBN 0-387-25496-X, S. 219–244, doi:10.1007/0-387-30746-x_11.
- Stefan Winkle: Das Blutwunder als mikrobiologisches und massenpsychologisches Phänomen. Beitrag zur Geschichte des Bacterium prodigiosum (Serratia marcescens) und zur Phänomenologie der Intoleranz. In: Laboratoriumsmedizin 7 (1983), Heft 9, S. 143–149.
Einzelnachweise
- ↑ a b B. Ajithkumar, V. P. Ajithkumar u. a.: Spore-forming Serratia marcescens subsp. sakuensis subsp. nov., isolated from a domestic wastewater treatment tank. In: International Journal of Systematic and Evolutionary Microbiology. Band 53, Nr. 1, Januar 2003, S. 253–258, ISSN 1466-5026. doi:10.1099/ijs.0.02158-0. PMID 12656181.
- ↑ a b c Steven D. Mahlen: Serratia infections: from military experiments to current practice. In: Clinical Microbiology Reviews. Band 24, Nr. 4. American Society for Microbiology, 2011, ISSN 0893-8512, S. 755–791, PMID 21976608 (asm.org [abgerufen am 17. Februar 2013]).
- ↑ H. M. Aucken, T. L. Pitt: Antibiotic resistance and putative virulence factors of Serratia marcescens with respect to O and K serotypes. In: Journal of Medical Microbiology. Band 47, Nr. 12. Society for General Microbiology, 1998, ISSN 0022-2615, S. 1105–1113, doi:10.1099/00222615-47-12-1105, PMID 9856647 (englisch, sgmjournals.org [PDF; abgerufen am 17. Februar 2013]).
- ↑ a b c Jean Euzéby, Aidan C. Parte: Genus Serratia. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Abgerufen am 6. August 2014.
- ↑ a b Serratia marcescens. In: Taxonomy Browser auf der Website des National Center for Biotechnology Information (NCBI). Abgerufen am 6. August 2014.
- ↑ a b TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen. In: Webseite der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. April 2012, S. 200, abgerufen am 30. April 2014.
- ↑ R. Leclercq et al.: EUCAST expert rules in antimicrobial susceptibility testing. In: Clinical Microbiology and Infection. Band 19, Nr. 2. Wiley-Blackwell, 2013, ISSN 1469-0691, S. 141–160, doi:10.1111/j.1469-0691.2011.03703.x, PMID 22117544.
- ↑ Kathryn L. Patterson et al.: The etiology of white pox, a lethal disease of the Caribbean elkhorn coral, Acropora palmata. In: Proc Natl Acad Sci U S A, Band 99, Nr. 13, 25. Juni 2002, S. 8725–30, PMID 12077296
- ↑ Kathryn Patterson Sutherland et al.: Human Pathogen Shown to Cause Disease in the Threatened Eklhorn Coral Acropora palmata. In: PLoS ONE, Band 6, Nr. 8, e23468, doi:10.1371/journal.pone.0023468
- ↑ a b Francine Grimont, Patrick A. D. Grimont: The Genus Serratia. In: The Prokaryotes. A Handbook on the Biology of Bacteria, Volume 6. 2006, S. 222–223.
- ↑ a b c Aloysius Krieg: Diseases caused by bacteria and other prokaryotes. In: James R. Fuxa, Yoshinori Tanada (Hrsg.): Epizootiology of Insect Diseases. Wiley-Interscience, New York 1987, S. 323–355 (S. 328 f.), ISBN 0-471-87812-X.
- ↑ Patrick A. D. Grimont, Francine Grimont, Oleg Lysenko: Species and Biotype Identification of Serratia Strains Associated with Insects. In: Current Microbiology. Band 2, Nr. 3. Springer-Verlag, New York 1979, S. 139–142, doi:10.1007/BF02605870 (englisch).
- ↑ G. E. Bucher: Nonsporulating Bacterial Pathogens. In: Edward Steinhaus (Hrsg.): Insect Pathology – An Advanced Treatise. Volume 2. Academic Press, New York/London 1963, S. 117–147 (S. 127), doi:10.1016/B978-0-12-395603-3.50008-2, ISBN 978-0-12-395603-3.
- ↑ Stefan Winkle: Das Blutwunder als mikrobiologisches und massenpsychologisches Phänomen. In: Laboratoriumsmedizin 7 (1983), S. 147f.
- ↑ Stefan Winkle: Das Blutwunder als mikrobiologisches und massenpsychologisches Phänomen. In: Laboratoriumsmedizin 7 (1983), S. 143f.
- ↑ Bakterienkrieg vor einem Bundesgericht, HAZ (Hannoversche Allgemeine Zeitung), 31. Januar 1981, zitiert in Jakob Segal, Lilli Segal: Aids - die Spur führt ins Pentagon. In: Jakob Segal, Lilli Segal, Manuel Kiper (Hrsg.): Biokrieg. Verlag Neuer Weg, 2. ergänzte Auflage, Oktober 1990, ISBN 3-88021-199-X, S. 140.
- ↑ Edward Steager, Chang-Beom Kim, Jigarkumar Patel, Socheth Bith, Chandan Naik: Control of microfabricated structures powered by flagellated bacteria using phototaxis. In: Applied Physics Letters. Band 90, Nr. 26, 25. Juni 2007, S. 263901, doi:10.1063/1.2752721.
Weblinks
- Basilio J Anía u. a.: Serratia (in englischer Sprache). In: Medscape. Abgerufen am 7. August 2014.
Auf dieser Seite verwendete Medien
Autor/Urheber: de:Benutzer:Brudersohn, Lizenz: CC BY-SA 3.0
Kolonien auf Agargel-Nährboden