Salmonellen
| Salmonellen | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
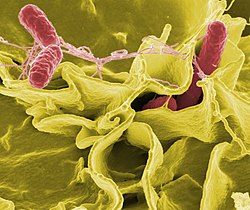
 Sekundärelektronenmikroskopaufnahme von Salmonellen (rot eingefärbt) | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Salmonella | ||||||||||||
| Lignières 1900 | ||||||||||||
| Arten | ||||||||||||
Nach der derzeit gültigen Nomenklatur
Salmonella enterica wird in die unten aufgeführten sechs Subspezies unterteilt. Innerhalb der Subspezies ordnet man individuelle Isolate einem der mehr als 2600 Serovare zu. |
Salmonellen (Salmonella) sind eine Gattung stäbchenförmiger Bakterien aus der Familie der Enterobakterien (Enterobacteriaceae), die bei Menschen und vielen Tieren Krankheiten verursachen können. Joseph Lignières benannte die von ihm beschriebene Gattung im Jahr 1900 nach dem US-amerikanischen Tierarzt Daniel Elmer Salmon.
Beschreibung
Salmonellen sind stäbchenförmige gramnegative Bakterien mit einem Durchmesser zwischen etwa 0,7 bis 1,5 µm und einer Länge von etwa 2 bis 5 µm. Sie sind vorwiegend aktiv beweglich, peritrich begeißelt, fakultativ anaerob, chemoorganotroph mit oxidativem und fermentativem Energiestoffwechsel und nicht sporenbildend. Sie werden in der Gattung Salmonella zusammengefasst und der Familie der Enterobacteriaceae zugeordnet. Sie sind eng verwandt mit der Gattung Escherichia. Sie kommen weltweit in wechsel- und gleichwarmen Tieren, in Menschen und in Habitaten außerhalb von Lebewesen vor.
Salmonellen als Krankheitserreger
Salmonellosen (Erkrankungen durch Salmonellen) gehören zu den Zoonosen, Menschen wie Tiere sind von Infektionen betroffen. Für Menschen ist die Übertragung über Lebensmittel eine häufige Ursache für eine Erkrankung an der Salmonellose.[1] Salmonellen kommen unter anderem besonders auf Eiern und Geflügelfleisch vor. Im Gegensatz zu beispielsweise den 1980er Jahren ist Schweinefleisch in den 2020er Jahren vergleichsweise selten mit Salmonellen belastet.
Man unterscheidet zwischen den Enteritis- und den Typhus/Paratyphus-Salmonellen, wobei letztere aufgrund spezieller Virulenzfaktoren und eines Kapselproteins (Virulenz-Antigen) schwerere Erkrankungen verursachen. Auslöser sind Salmonella enterica subsp. enterica Serovar Typhi, kurz Salmonella Typhi und Salmonella Paratyphi. Salmonella Typhi kommt bei Tieren nicht vor und ist an Menschen angepasst.
Enteritis-Salmonellen, beispielsweise Salmonella enterica subsp. enterica Serovar Enteritidis (Kurzbezeichnung Salmonella Enteritidis) und Salmonella Typhimurium, verursachen beim Menschen meist spontan ausheilende Durchfallerkrankungen, die in der Regel nicht antibiotisch behandelt werden müssen. Allerdings können bei Risikogruppen wie Säuglingen, Kleinkindern, alten Menschen, HIV-Patienten und immungeschwächten Patienten schwere Erkrankungen (Allgemeininfektionen) hervorgerufen werden.
| ausgewählte Fallzahlen | |
|---|---|
| Jahr | Fälle |
| 1990 | 200.000 |
| 2005 | 52.245[2] |
| 2020 | 8.743[3] |
In Deutschland gehören Salmonellosen zu den sogenannten meldepflichtigen Erkrankungen (§ 6 bzw. § 7) des Infektionsschutzgesetzes. Die amtlichen Meldungen sind seit 1990 von etwa 200.000 auf rund 55.000 Fälle im Jahr 2005 zurückgegangen. Für das Jahr 2014 wurden in Deutschland nur noch 16.220 Fälle gemeldet. 17 Fälle wurden als krankheitsbedingt verstorben übermittelt. In der Schweiz melden die Labore jährlich zwischen 1200 und 1500 Nachweise von Salmonellen.[4]
Bis zu 5 % der Erkrankten werden zu sogenannten Dauerausscheidern, nach der Genesung scheiden sie weiterhin Salmonellen (via Galle oder Dünndarm) mit dem Stuhl aus. Nach § 42 Abs. 1 IfSG zieht das ein dauerhaftes Tätigkeits- und Beschäftigungsverbot in der Herstellung bestimmter Lebensmittel sowie in Küchen von Gaststätten und sonstigen Einrichtungen der Gemeinschaftsverpflegung nach sich.[5]
Salmonellen sind außerhalb des menschlichen bzw. tierischen Körpers wochenlang lebensfähig. In einigen Fällen können sie sich bereits ab einer Temperatur von etwa 6 °C vermehren. Sie sind dazu dann bis zu einer Temperatur von etwa 47 °C lebensfähig. Mit einer Erhitzung auf 70 °C für 10 min. wird eine sichere Abtötung erreicht. Durch Einfrieren werden die Bakterien hingegen nicht abgetötet.[1]
Hände können mit alkoholischen Desinfektionsmitteln desinfiziert werden. Bei behördlich angeordneten Desinfektionsmaßnahmen ist die Liste der vom Robert Koch-Institut geprüften und anerkannten Desinfektionsmittel und -verfahren (doi:10.1007/s00103-017-2634-6) heranzuziehen.[1] In getrocknetem Kot sind Salmonellen über 2,5 Jahre lang nachweisbar. In sauren Lösungen sterben sie rasch ab, weswegen die Einnahme von Medikamenten, welche die Produktion der Magensäure hemmen, das Risiko einer Infektion mit Salmonellen erhöht.[6] Sonnenlicht (UV-Strahlung) beschleunigt das Absterben der Erreger.
In Dänemark gab es 2021 einen Salmonellose-Ausbruch, welcher auf Flohsamenschalen zurückzuführen ist. Insgesamt gab es 33 bestätigte Fälle und drei Todesfälle.[7]
Geschichte
- 1874 beschrieb der polnische Pathologe Tadeusz Browicz zum ersten Mal ein Bakterium als Ursache von Typhus.
- 1880 wurde der Erreger des Typhus abdominalis beim Menschen von Karl Joseph Eberth und Robert Koch entdeckt.
- 1881 gelang Georg Gaffky die Züchtung des Erregers in Reinkultur (Publiziert 1884).
- 1888 August Gärtner entdeckt den Verursacher der „Fleischvergiftung“ und damit einen Erreger der Salmonellen-Enteritis (Bacillus enteritidis Gärtner = Salmonella enteritidis).[8]
- 1889 fand Daniel Elmer Salmon, nach dem die Gattung Salmonella benannt wurde, die „Schweinecholera“-Bakterien.
- Zwischen 1888 und 1892 entdeckte Friedrich Loeffler im Rahmen einer Mäusetyphus-Epidemie bei Versuchsmäusen in Greifswald[9] den auch für den Menschen gefährlichen Enteritis-Erreger Salmonella typhi-murium (Salmonella Serovar Typhimurium).
Systematik und Nomenklatur



Na2S2O3 wird zu H2S verstoffwechselt, welches mit Eisen(III)-Ionen im Nährmedium zu schwarzem Eisensulfid reagiert
Die Systematik und Nomenklatur der Salmonella-Arten ist sehr komplex. In der Anfangsphase wurden Salmonella-Arten nach klinischen Gesichtspunkten gebildet und benannt, beispielsweise Salmonella typhi-murium (Mäusetyphus), S. abortus-ovis (Abort des Schafs), S. cholerae-suis (Cholera des Schweins).
Kauffmann definierte 1941 aufgrund serologischer Befunde jedes neu entdeckte Serovar als neue Art.[10]
Als erkannt wurde, dass die Wirtsspezifität mancher Arten nicht existiert – S. typhimurium und S. choleraesuis sind auch für den Menschen pathogen – wurden neue Serovare als eigenständige Salmonella-Arten angesehen und nach dem Ort bezeichnet, an dem der erste Stamm der neuen Art isoliert wurde.
1966 wurde beim Neunten Internationalen Mikrobiologischen Kongress in Moskau beschlossen, den Bindestrich in den Artnamen zu entfernen (also z. B. S. typhimurium).
Aufgrund molekularbiologischer Erkenntnisse (insbesondere DNA/DNA-Hybridisierung) schlugen Le Minor und Popoff vor, die Gattung Salmonella als nur aus einer einzigen Art bestehend anzusehen, nämlich aus S. enterica – diesem Vorschlag wurde jedoch nicht stattgegeben.[11][12] Derzeit (Stand 2023) gilt, dass es zwei Arten gibt: Salmonella enterica und Salmonella bongori. Zu 99,5 % gehören Salmonellen, die aus Salmonellose-erkrankten Menschen und warmblütigen Tieren isoliert wurden, der Gruppe I an Salmonella enterica ssp. enterica. Vertreter der Subspezies salamae (Gruppe II) und houtenae (Gruppe IV) wurden aus Reptilien isoliert; am seltensten sind Subspezies der Gruppe VI (Subspezies indica). S. enterica ssp. bongori wurde von Le Minor und Popoff (1987)[11] als Gruppe V klassifiziert, aber Reeves u. a. (1989)[13] schlugen vor, die Gruppe V als eigene Art S. bongori. zu bezeichnen.
Medizinisch relevante Salmonellen gehören der
- Gruppe I (S. enterica ssp. enterica) sowie der
- Gruppe IIIa (S. enterica ssp. arizonae) und der
- Gruppe IIIb (S. enterica ssp. diarizonae) an.
Diese sechs Salmonellengruppen können mit biochemischen Tests wie folgt klassifiziert werden:[14]
| Gruppe (nach Subspezies) | Gruppe (nach Nummer) | Malonat | ONPG | Dulcit | Salicin | Galakturonsäure | Gelatine |
|---|---|---|---|---|---|---|---|
| S. enterica ssp. enterica | I | negativ | negativ | positiv | negativ | negativ | negativ |
| S. enterica ssp. salamae | II | positiv | negativ | positiv | negativ | positiv | positiv |
| S. enterica ssp. arizonae, und S. enterica ssp. diarizonae | III | positiv | positiv | negativ | negativ | schwach | positiv |
| S. enterica ssp. houtenae | IV | negativ | negativ | negativ | positiv | positiv | positiv |
| S. enterica ssp. indica | VI | negativ | variabel | variabel | negativ | positiv | positiv |
| S. bongori | V | negativ | positiv | positiv | negativ | positiv | negativ |
Erläuterungen: Malonat: Verwertung von Malonat als Energie- und Kohlenstoffquelle (C-Quelle); ONPG: Hydrolyse von ONPG; Dulcit: Verwertung von Dulcit, einem Zuckeralkohol, als Energie- und C-Quelle; Salicin: Verwertung von Salicin, einem Glycosid, das in Weiden (Gattungsname: Salix) vorkommt, als Energie- und C-Quelle, Galakturonsäure: Verwertung von Galacturonsäure, dem Hauptbestandteil der Pektine, Gelatine: Hydrolyse von Gelatine.
Nach einer fast zwanzig Jahre andauernden Diskussion wurden 2005 wurden Vorschläge von Léon Le Minor und Michel Y. Popoff[11] und Michael W. Reeves[13] angenommen und zu einer reformierten Nomenklatur zusammengefasst.[15][16] Dieses formale, von mikrobiologischen Systematikern erstellte System führt zu sehr umfangreichen Namen. Vollständig würde der Erreger des Typhus zum Beispiel -Salmonella enterica subspezies enterica serovar Typhi- heißen, wobei ein Teil des Namens kursiv geschrieben würde und ein anderer nicht. Aus Praktikabilitätsgründen wird dieses Benennungsprinzip von beispielsweise Mikrobiologen und Infektiologen verkürzt angewandt zu beispielsweise Salmonella Typhi.
Es gibt nach dem Kauffmann-White-Schema insgesamt mehr als 2600 Salmonellen-Serovare, die sich aufgrund des Vorkommens von unterschiedlichen O- und H-Antigenen unterscheiden.[17][18] Die O-Antigene sind Bestandteil der Lipopolysaccharide (LPS) der Zellwand und die H-Antigene Bestandteil der Proteinbausteine der Geißeln (Flagellen), mit denen sich die Salmonellen fortbewegen können. Zusätzlich verfügen einige Arten über ein Antigen in der Schleimhülle („Kapsel“), sogenanntes Kapselantigen (= K-Antigen). Da durch das Kauffmann-White-Schema nur eine H-Phase nachweisbar ist und zur Typisierung aber auf jeden Fall beide H-Phasen benötigt werden, muss die zweite (andere) H-Phase durch eine Schwärmplatte nach Sven-Gard zur Ausbildung gebracht werden.
- O-Antigen (= somatisches Antigen). lokalisiert in der äußeren Membran, Lipopolysaccharide, thermostabil, Formaldehydunbeständig, sogenannte Oberflächen-Antigene
- H-Antigen (= Geißelantigen, H wie Hauch). thermolabil, Formaldehydbeständig.
- K-Antigen (= Kapselantigen, Hüllenantigen). besteht aus drei Fraktionen mit unterschiedlicher Wärmeempfindlichkeit und wird der Zellwand zugerechnet.
Epidemiologische Gruppen
Einordnung der Serovare nach der Anpassung an bestimmte Wirte:
- an den Menschen angepasste Serovare, die Typhus oder Paratyphus verursachen (Salmonella Typhi, Salmonella Paratyphi A, B und C),
- an bestimmte Tierarten angepasste Serovare, die tierspezifische Erkrankungen hervorrufen und für andere Tierarten und den Menschen nicht von Bedeutung sind Salmonella Dublin (Rind), Salmonella Abortusovis, Abortusequi (Schaf, Pferd)
- Serovare ohne spezielle Wirtsanpassung, die bei allen Tierspezies als Erreger von Enteritiden auftreten und beim Menschen Lebensmittelvergiftungen hervorrufen
- Serovare ohne spezielle Wirtsanpassung, die bei Mensch und Tier als Erreger von Salmonellosen auftreten und eine hohe Virulenz besitzen.
Infektionsmöglichkeiten
Infektionen mit Salmonellen sind möglich:
- durch Unsauberkeit im Lebensmittelbereich, insbesondere in Küchen
- durch die Ausscheidungen von Erkrankten, aber auch klinisch gesund erscheinenden infizierten Menschen (Dauerausscheider) und Tieren (gefährdet: andere Tiere und Pflegepersonal); vor allem auch von unerkannt infizierten Reptilien (Befallsrate bei 90 %), eine Gefahr vor allem für Kleinkinder[19]
- durch unhygienisch aufgetautes Geflügel (viele Bakterien befinden sich im Tauwasser) sowie
- durch rohe Eier, die von mit Salmonellen infiziertem Geflügel stammen (die Salmonellen befinden sich normalerweise nur auf der Eierschale, können bei verletzter Kutikula jedoch auch ins Innere gelangen).
Medizinisch bedeutsame Vertreter

- Salmonella enterica ssp. arizonae, bei Kaltblütern, Geflügel, Säugetieren
- Salmonella Choleraesuis, Darmkommensale des Schweines, der Erkrankungen in heranwachsenden Schweinen auslösen kann (Salmonellose des Schweins); pathogen für Menschen bei Resistenzschwäche;[20]
- Salmonella Enteritidis, Vorkommen im Darm von Rindern, Nagetieren, Enten (auch deren Eiern) und Menschen; Erreger des Kälberparatyphus und akuter Gastroenteritis des Menschen.
- Salmonella Paratyphi A, rein humanpathogen, Erreger des „Paratyphus A“ (Paratyphöse Gastroenteritis), Übertragung durch Kontakt und infektiöse Lebensmittel oder Wasser.
- Salmonella Paratyphi B: Von dieser Serovarietät existieren zwei Varianten, die sich hinsichtlich der Fermentation von d-Tartrat unterscheiden. Die d-Tartrat-negative Variante kommt praktisch nur bei Menschen vor, gilt als Erreger des „Paratyphus B“ und kommt in Mitteleuropa kaum vor. Die d-Tartrat-positive Variante, ist hingegen in Mitteleuropa vorherrschend. Sie hat ein tierisches Reservoir, gilt als Auslöser einer Salmonellenenteritis und wird auch als Salmonella Java bezeichnet.[21]
- Salmonella Typhi, Vorkommen in gemäßigten bis tropischen Zonen, humanpathogener Erreger des Typhus abdominalis, Übertragung durch Kontakt und infektiöse Lebensmittel, Wasser oder Fliegenkot. 2–5 % aller Erkrankten bleiben Dauerausscheider.[22]
- Salmonella Typhimurium, Erreger einer für Vögel meist tödlich verlaufenden, fieberhaften Darminfektion. Auch Säugetiere können durch kontaminierte Futtermittel erkranken. Häufiger Auslöser der Salmonellenenteritis („Lebensmittelvergiftung“) des Menschen.[23]
- Salmonella Dublin, einer der Erreger der anzeigepflichtigen Rindersalmonellose
- Salmonella Typhisuis, neben S. Choleraesuis einer der Verursacher der Salmonellose des Schweins
Meldepflicht
In Deutschland ist jeder direkte Nachweis von Salmonella Paratyphi oder Salmonella Typhi namentlich meldepflichtig nach § 7 des Infektionsschutzgesetzes. Zudem der direkte oder indirekte Nachweis sonstiger Salmonellen, soweit der Nachweis auf eine akute Infektion hinweist. Die Meldepflicht betrifft in erster Linie die Leitungen von Laboren (§ 8 IfSG).
In der Schweiz ist der positive und negative laboranalytische Befund zu Salmonellen (Salmonella spp.) für Laboratorien meldepflichtig und zwar nach dem Epidemiengesetz (EpG) in Verbindung mit der Epidemienverordnung und Anhang 3 der Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen.
Literatur
- Giulia Enders, Jill Enders (Illustrationen): Darm mit Charme. Alles über ein unterschätztes Organ. Ullstein, Berlin 2014, ISBN 978-3-550-08041-8.[24][25]
- Werner Köhler: Salmonellen. In: Werner E. Gerabek u. a. (Hrsg.): Enzyklopädie Medizingeschichte. De Gruyter, Berlin / New York 2005, ISBN 3-11-015714-4, S. 1282.
Weblinks
- Literatur von und über Salmonellen im Katalog der Deutschen Nationalbibliothek
- Bedeutung der Salmonellen als Krankheitserreger. Bundesinstitut für Risikobewertung
- Fragen und Antworten zum Schutz vor Infektionen mit Salmonellen. Bundesinstitut für Risikobewertung
Einzelnachweise
- ↑ a b c RKI-Ratgeber Salmonellose. Robert Koch-Institut, 1. April 2016, abgerufen am 4. Dezember 2021.
- ↑ Robert Koch-Institut: Infektionsepidemiologisches Jahrbuch meldepflichtiger Krankheiten für 2005. Berlin 2006, ISBN 3-89606-094-5.
- ↑ Robert Koch-Institut: Infektionsepidemiologisches Jahrbuch meldepflichtiger Krankheiten für 2020. Berlin 2021, ISBN 978-3-89606-311-3. doi:10.25646/8773
- ↑ Salmonellose – Erreger und Übertragung 17. Januar 2013, Schweizerische Eidgenossenschaft – Bundesamt für Gesundheit (BAG) Abruf 23. November 2016.
- ↑ https://www.rki.de/DE/Content/Infekt/EpidBull/Merkblaetter/Ratgeber_Salmonellose.html
- ↑ Hau-Hsin Wu, Yung-Tai Chen, Chia-Jen Shih, Yi-Tzu Lee, Shu-Chen Kuo, Te-Li Chen: Association Between Recent Use of Proton Pump Inhibitors and Nontyphoid Salmonellosis: A Nested Case-Control Study. In: Clinical Infectious Diseases. Band 59, Nr. 11, 1. Dezember 2014, ISSN 1537-6591, S. 1554–1558, doi:10.1093/cid/ciu628, PMID 25091310, PMC 4650768 (freier Volltext) – (oup.com [abgerufen am 30. Januar 2023]).
- ↑ Der Spiegel: Dänemark: Salmonellen in Naturheilmittel – drei Tote. Abgerufen am 15. April 2021.
- ↑ Werner Köhler: Infektionskrankheiten. In: Werner E. Gerabek, Bernhard D. Haage, Gundolf Keil, Wolfgang Wegner (Hrsg.): Enzyklopädie Medizingeschichte. De Gruyter, Berlin / New York 2005, ISBN 3-11-015714-4, S. 667–671; hier: S. 670.
- ↑ Vgl. Gundolf Keil: Robert Koch (1843–1910). Ein Essai. In: Medizinhistorische Mitteilungen. Zeitschrift für Wissenschaftsgeschichte und Fachprosaforschung. Band 36/37, 2017/2018 (2021), S. 73–109, hier: S. 87 f.
- ↑ F. Kauffmann: Die Bakteriologie der Salmonella-Gruppe. Munksgaard, Kopenhagen 1941.
- ↑ a b c L. Le Minor, M. Y. Popoff: Request for an opinion. Designation of Salmonella enterica. sp. nov., nom. rev., as the type and only species of the genus Salmonella. In: Int. J. Syst. Bacteriol. Band 37, 1987, S. 465–468.
- ↑ J. P. Euzéby: Revised Salmonella nomenclature: designation of Salmonella enterica (ex Kauffmann and Edwards 1952) Le Minor and Popoff 1987 sp. nov. nom. rev. as the neotype species of the genus Salmonella Lignieres 1900 (Approv Lists 1980), rejection of the name Salmonella choleraesuis (Smith 1894) Weldin 1927 (Approved Lists 1980), and conservation of the name Salmonella typhi (Schroeter 1886) Warren and Scott 1930 (Approved Lists 1980). Request for an Opinion. In: International Journal of Systematic and Evolutionary Microbiology. Band 49, Nr. 2, 1. April 1999, ISSN 1466-5026, S. 927–930, doi:10.1099/00207713-49-2-927 (microbiologyresearch.org [abgerufen am 30. Januar 2023]).
- ↑ a b M. W. Reeves, G. M. Evins, A. A. Heiba, B. D. Plikaytis, J. J. Farmer III: Clonal nature of Salmonella typhi and its genetic relatedness to other salmonellae as shown by multilocus enzyme electrophoresis and proposal of Salmonella bongori comb. nov. In: J. Clin. Microbiol. Band 27, 1989, S. 313–320. PMID 2915026.
- ↑ Referensmetodik för laboratoriediagnostik vid kliniskt mikrobiologiska laboratorier. ( vom 27. Mai 2006 im Internet Archive; PDF) Tabelle 7
- ↑ Judicial Commission of the International Committee on Systematics of Prokaryotes: The type species of the genus Salmonella Lignieres 1900 is Salmonella enterica (ex Kauffmann and Edwards 1952) Le Minor and Popoff 1987, with the type strain LT2T, and conservation of the epithet enterica in Salmonella enterica over all earlier epithets that may be applied to this species. Opinion 80. In: Int. J. Syst. Evol. Microbiol. Band 55, 2005, S. 519–520. PMID 15653929
- ↑ B. J. Tindall, P. A. Grimont, G. M. Garrity, J. P. Euzeby: Nomenclature and taxonomy of the genus Salmonella. In: Int. J. Syst. Evol. Microbiol. Band 55, 2005, S. 521–524. PMID 15653930.
- ↑ Cameron P. Thompson, Alexandra N. Doak, Naufa Amirani, Erin A. Schroeder, Justin Wright, Subhashinie Kariyawasam, Regina Lamendella, Nikki W. Shariat: High-Resolution Identification of Multiple Salmonella Serovars in a Single Sample by Using CRISPR-SeroSeq. In: Applied and Environmental Microbiology. Band 84, Nr. 21, 1. November 2018, ISSN 1098-5336, S. e01859–18, doi:10.1128/AEM.01859-18, PMID 30170999, PMC 6193385 (freier Volltext).
- ↑ Sangeeta Banerji, Sandra Simon, Andreas Tille, Angelika Fruth, Antje Flieger: Genome-based Salmonella serotyping as the new gold standard. In: Scientific Reports. Band 10, Nr. 1, 9. März 2020, ISSN 2045-2322, S. 4333, doi:10.1038/s41598-020-61254-1, PMID 32152449, PMC 7062728 (freier Volltext) – (nature.com [abgerufen am 30. Januar 2023]).
- ↑ Marieke Degen: Gefährliche Lieblinge, DLF – Forschung Aktuell, 4. April 2011 (8. April 2011)
- ↑ Po Lin Chen, Chi Jung Wu, Chia Ming Chang, Hsin Chun Lee, Nan Yao Lee, Hsin I. Shih, Ching Chi Lee, Nai Ying Ko, Li Rong Wang, Wen Chien Ko: Extraintestinal focal infections in adults with Salmonella enterica serotype Choleraesuis bacteremia. In: Journal of Microbiology, Immunology, and Infection = Wei Mian Yu Gan Ran Za Zhi. Band 40, Nr. 3, Juni 2007, ISSN 1684-1182, S. 240–247, PMID 17639165.
- ↑ Justin Denny, J. Threlfall, J. Takkinen, S. Löfdahl, T. Westrell, C. Varela, B. Adak, N. Boxal, S. Ethelberg, M. Torpdahl, M. Straetemans, W. van Pelt: Multinational Salmonella Paratyphi B variant Java (Salmonella Java) outbreak, August–December 2007. In: Eurosurveillance. Band 12, Nr. 51. European Centre for Disease Prevention and Control (ECDC), 2007, ISSN 1025-496X, PMID 18179762 (eurosurveillance.org [abgerufen am 19. Februar 2013]).
- ↑ Typhus abdominalis, Paratyphus. In: RKI-Ratgeber. Robert Koch-Institut, 15. Januar 2008, abgerufen am 30. Januar 2023.
- ↑ Fachinformation – Keime im Visier – Salmonella spp. LADR Lebensmittelanalytik
- ↑ Sophia Guttenberger: Das verkannte Tabu-Organ. Rezension. In: Spektrum der Wissenschaft. 16. Mai 2014.
- ↑ Pete Smith: Ein Schätzelchen, dieser Darm! Rezension. In: Ärzte Zeitung. 14. April 2014.
Auf dieser Seite verwendete Medien
Autor/Urheber: Nathan Reading from Halesowen, UK, Lizenz: CC BY 2.0
Salmonella species growing on XLD agar. Showing classical H2S production.
Autor/Urheber: Flickr user denn, Lizenz: CC BY-SA 2.0
A microbiological culture of Salmonella sp. bacteria on DC agar culture medium
Autor/Urheber: Nathan Reading from Halesowen, UK, Lizenz: CC BY 2.0
Salmonella species growing on X.L.D. agar.
Showing classical H2S productionColor-enhanced scanning electron micrograph showing Salmonella Typhimurium (red) invading cultured human cells Credit: Rocky Mountain Laboratories, NIAID, NIH All the images, except specified ones from the World Health Organization (WHO), are in the public domain. For the public domain images, there is no copyright, no permission required, and no charge for their use.
Autor/Urheber: Photo: Volker Brinkmann, Max Planck Institute for Infection Biology, Berlin, Germany, Lizenz: CC BY 2.5
Salmonella typhimurium