Rotavirus
| Rotavirus | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
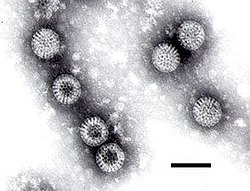 Rotaviren in einer TEM-Aufnahme (Längenmaßstab 100 nm) | ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| Rotavirus | ||||||||||||||||
| Links | ||||||||||||||||
|


Die Gattung Rotavirus umfasst Viren der Familie Sedoreoviridae in der Ordnung Reovirales mit einem Genom aus elf Segmenten einer doppelsträngigen RNA (dsRNA). Rotaviren besitzen sehr komplexe Viruspartikel (Virionen), die aus zwei konzentrischen Kapsiden und einer inneren Corestruktur bestehen; dies wird auch als dreischichtige Kapsidstruktur bezeichnet. Zwischen äußerem und innerem Kapsid sind in TEM-Aufnahmen Proteinbrücken und Kanäle zu erkennen, die in der Negativkontrastierung radähnlich erscheinen. Von diesem Aussehen ist auch der Name der Gattung (von lateinisch rota „Rad“) abgeleitet.[3] Rotaviren wurden erstmals in den 1950er Jahren als Erreger einer Diarrhoe bei der Maus identifiziert, seither fand man sie bei mehreren Säugetieren und Vögeln. Für den Menschen sind Subtypen aus drei Rotavirusspezies bekannt, die man als Humane Rotaviren zusammenfasst.
Genom
Genomkarte der Gattung Rotavirus.
Systematik

Einen ähnlichen Aufbau mit zwei konzentrischen Schalen haben auch die Viren der Gattungen Coltivirus und Orbivirus (letztere beispielsweise mit Spezies Blauzungenvirus), beide in derselben Familie Sedoreoviridae (früher: Unterfamilie Sedoreovirinae).[4]
Die Gattung Rotavirus wird in neun Spezies (A-J) eingeteilt.[5] Spezies E wurde noch nicht als eigenständige Spezies bestätigt. Als eines der wichtigsten Definitionskriterien einer Rotavirus-Spezies gilt, dass nur zwischen den Subtypen ein Reassortment stattfinden kann, nicht jedoch zwischen einzelnen Spezies. Sie bilden somit einen einheitlichen Genpool, der mittels Antigenshift und Antigendrift neue Subtypen bilden kann. Bei Sequenzvergleichen des Coreproteins VP2 erscheinen die Spezies Rotavirus A und C als eng verwandt, während Rotavirus B deutliche Sequenzunterschiede aufweist.
- Gattung Rotavirus
- Spezies Rotavirus A
- Subtyp Simianes Rotavirus A
- Subtyp Aviäres Rotavirus A
- Subtyp Bovines Rotavirus A
- Subtyp Porcines Rotavirus A
- Subtyp Caprines Rotavirus A
- Subtyp Equines Rotavirus A
- Subtyp Humanes Rotavirus A
- Subtyp Lapines Rotavirus A
- Subtyp Kaninchen-Rotavirus
- Vorläufige Subtypen der Spezies:
- Subtyp Rotavirus der Lämmer
- Subtyp Canines Rotavirus
- Subtyp Rhesus-Rotavirus
- Spezies Rotavirus B
- Subtyp (Serogruppe) Bovines Rotavirus B
- Subtyp Humanes Rotavirus B
- Spezies Rotavirus C
- Subtyp Humanes Rotavirus C
- Spezies Rotavirus D
- Subtyp Hühner-Rotavirus D
- Spezies Rotavirus E *
- Subtyp Porcines Rotavirus E
- Spezies Rotavirus F
- Subtyp Hühner-Rotavirus F
- Spezies Rotavirus G
- Subtyp Hühner-Rotavirus G
- Spezies Rotavirus H
- Spezies Rotavirus I
- Spezies Rotavirus J
Quellen
- R. F. Ramig et al.: Genus Rotavirus. In: C. M. Fauquet, M. A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses. London, San Diego 2005, ISBN 0-12-249951-4, S. 484–496
- Robert D. Shaw und Harry B. Greenberg: Rotaviruses (Reoviridae). In: Allan Granoff, Robert G. Webster (Hrsg.): Encyclopedia of Virology. San Diego, 1999 (Band 1–3), ISBN 0-12-227030-4, Band 3, S. 1576–1592
- J. Matthijnssens et al.: Full genomic analysis of human rotavirus strain B4106 and lapine rotavirus strain 30/96 provides evidence for interspecies transmission. J. Virol. (2006) 80(8): S. 3801–3810, PMID 16571797
- J. A. Lawton et al.: Three-dimensional structural analysis of recombinant rotavirus-like particles with intact and amino-terminal-deleted VP2: implications for the architecture of the VP2 capsid layer. J. Virol. (1997) 71(10): S. 7353–7360, PMID 9311813, PMC 192080 (freier Volltext)
Weblinks
- Gattung Rotavirus (NCBI)
- Rotavirus in der Datenbank des ICTV
- Early Steps of Viral Assembly Revealed by Genetically Engineered Slow-Growing Mutant Rotavirus, auf: SciTechDaily vom 25. Juni 2020, Quelle: Baylor College of Medicine
- S. Lopez, C. F. Arias: Early Steps in Rotavirus Cell Entry, in: Curr Top Microbiol Immunol 309, 2006, S. 39–66, doi:10.1007/3-540-30773-7_2, PMID 16909896.
Einzelnachweise
- ↑ a b c d e ICTV: Bluetongue virus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ ICTV Master Species List 2018b v1 MSL #34, Feb. 2019
- ↑ Cornelia Henke-Gendo: Virale Gastroenteritiserreger. In: Sebastian Suerbaum, Gerd-Dieter Burchard, Stefan H. E.Kaufmann, Thomas F. Schulz (Hrsg.): Medizinische Mikrobiologie und Infektiologie. Springer-Verlag, 2016, ISBN 978-3-662-48678-8, S. 513 ff., doi:10.1007/978-3-662-48678-8_65.
- ↑ Houssam Attoui et al.: Coltiviruses and seadornaviruses in North America, Europe, and Asia. In: Emerging Infectious Diseases. Band 11, Nr. 11, November 2005, S. 1673–1679, doi:10.3201/eid1111.050868, PMID 16318717, PMC 3367365 (freier Volltext) – (englisch).
- ↑ ICTV Master Species List 2022_MSL38.v1. In: ICTV. Abgerufen am 27. September 2023 (englisch).
Auf dieser Seite verwendete Medien
Autor/Urheber: Javier M. Rodríguez, Francisco J. Chichón, Esther Martín-Forero, Fernando González-Camacho, José L. Carrascosa, José R. Castón, and Daniel Luque, Lizenz: CC BY 4.0
Single particle three-dimensional structure of Rotavirus strain SA11. (A) Surface-shaded representation of the outer surfaces of NTR particles, viewed along an icosahedral 2-fold axis. The surfaces are radially color-coded to represent VP4 or VP8*/VP5* spikes (red), VP7 (yellow), and VP6 (blue). The density is contoured at 1σ above the mean. The bar represents 100 Å. (B) Transverse sections, 2.8 Å thick, taken from the maps of NTR- TLP, parallel but displaced 34 Å from the central section, viewed along a 2-fold axis (darker, denser). Arrows indicate spikes in the surface-shaded representations in A and the corresponding densities in B. For the NTR map, the relative density of the spike contained ∼50% of the shell density (C) Close up view of the NTR (C) spike represented as in A.
Note the wheel-like appearance of some of the rotavirus particles. The observance of such particles gave the virus its name ('rota' being the Latin word meaning wheel). Bar = 100 nanometers.
Source: Cell culture.
Method: Negative-stain Transmission Electron MicroscopyAutor/Urheber: ViralZone, SIB Swiss Institute of Bioinformatics: https://viralzone.expasy.org, Lizenz: CC BY 4.0
Schemazeichnng eines Virions der Gattung Rotavirus, Querschnitt und Seitenansicht, angeschnitten.
Autor/Urheber: ViralZone, SIB Swiss Institute of Bioinformatics: https://viralzone.expasy.org, Lizenz: CC BY 4.0
Genomkarte der Gattung Rotavirus





