Plastiden-DNA
Fachsprachlich wird als Chloroplasten-DNA bzw. Plastiden-DNA, kurz ctDNA, cpDNA[1] oder ptDNA,[2] die doppelsträngige, zumeist zirkuläre DNA im Inneren (Stroma) von Chloroplasten (oder allgemeiner Plastiden) bezeichnet. Das Chloroplasten/Plastiden-Genom wird als Plastom bezeichnet. Bis auf wenige Ausnahmen enthalten Chloroplasten und andere Plastiden solche eigene DNA.[3]
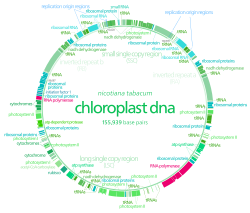
Der erste Nachweis von Plastiden-DNA erfolgte 1962.[4] 1986 wurde erstmals ein Plastom sequenziert, als zwei japanische Forscherteams die Chloroplasten-DNA von Marchantia polymorpha (Brunnenlebermoos, Lebermoose) und Nicotiana tabacum (Tabak, Nachtschattengewächse) sequenzierten.[5] Seitdem wurden Hunderte von Chloroplasten-DNAs aus verschiedenen Spezies sequenziert. Meist handelt es sich dabei jedoch um Chloroplastida, d. h. Landpflanzen oder Grünalgen. Glaucophyten (Glaucophyta), Rotalgen (Rhodophyta syn. Rhodophyceae) und andere Algengruppen sind stark unterrepräsentiert.[6]
Molekulare Struktur

Bei den Chloroplastida (mit den Grünalgen und Landpflanzen) haben die Chloroplasten ihr gesamtes Genom gewöhnlich in einem einzigen großen DNA-Ring zusammengefasst,[6] der normalerweise 120.000–170.000 bp (Basenpaare) lang ist.[4][8][9][10] Die Konturlänge liegt bei etwa 30 bis 60 µm, die Masse bei etwa 80 bis 130 MDa (Millionen Dalton).[11]
Obwohl die Chloroplasten-DNA normalerweise als zirkuläres Molekül angenommen wird, gibt es Hinweise darauf, dass sie – vor allem bei anderen Gruppen wie den erwähnten Glaucophyten oder Rotalgen – häufig eine lineare Form haben könnte.[6][12]
Gegenläufige Kopien (Inverted Repeats)
In den meisten photosynthetischen Organismen hat das Chloroplasten-Genom eine vierteilige Struktur. Hierbei handelt es sich um zwei gegenläufige Kopien (englisch inverted repeats), die einen langen Einzelkopie-Abschnitt (englisch long single copy section, LSC) von einem kurzen Einzelkopie-Abschnitt (englisch short single copy section, SSC) trennen.[9][6]
Die Größen der drei unterschiedlichen DNA-Regionen kann von Spezies zu Spezies variieren bis hin zum vollständigen Verlust der kopierten Regionen. Die Inverted Repeats reichen in der Länge von 4 bis 25 kbp, mit jeweils nur vier bis zu über 150 Genen.[6] Inverted Repeats in Pflanzen befinden sich eher am oberen Ende dieser Skala und sind jeweils 20 bis 25 kbp lang.[9][13] Unter den Landpflanzen sind die Inverted Repeats jedoch hoch konserviert und akkumulieren nur wenige Mutationen.[9][13] Ähnliche Inverted Repeats wie bei Grünalgen und Landpflanzen gibt es im Genom von Cyanobakterien und den beiden anderen Chloroplastenlinien (Glaucophyten und Rotalgen, s. u.), was darauf hindeutet, dass die Inverted Repeats älter sind als die Chloroplasten,[6] auch wenn einige Chloroplastenlinien diese Merkmale ihrer DNA seitdem verloren haben[13][14] oder diese umgedreht haben (zurück zu direkten Wiederholungen).[6] Möglicherweise tragen die Inverted Repeats dazu bei, den Rest des Chloroplasten-Genoms zu stabilisieren: Chloroplasten-DNAs, die einige der Inverted Repeats verloren haben, neigen dazu sich umzuordnen (englisch DNA rearrangement).[14]
Nucleoide
Neue Chloroplasten können bis zu 100 Kopien ihrer DNA enthalten,[4] auch wenn ihre Anzahl mit dem Alter der Chloroplasten auf etwa 15 bis 20 abnimmt.[15] Sie sind normalerweise wie bei Prokaryoten in Nucleoide (Kernäquivalente, hier: Chloroplasten-Nucleoide oder kurz cp-Nucleoide) verpackt, die mehrere identische Chloroplasten-DNA-Ringe enthalten können. In jedem Chloroplasten finden sich zudem etliche solche Nucleoide.[11] Bei primitiven Rotalgen befinden sich die cp-Nucleoide im Zentrum des Chloroplasten, während bei grünen Pflanzen und Grünalgen die cp-Nucleoide im gesamten Stroma verteilt sind.[16]
Die DNA von Chloroplasten, wie auch von Bakterien und Mitochondrien ist zwar nicht mit echten Histonen assoziiert (diese gibt es nur in den Zellkernen der Eukaryoten und in Vorstufen bei Archaeen). Das dichte Packen der DNA in ein Nucleoid wird bei diesen Gruppen durch Proteine bewerkstelligt, deren Funktion daher histonähnlich ist (d. h. analog zu Histonen). Diese sind HU in Bakterien, Abf2 in Mitochondrien und HC (Akronym für englisch histone-like protein of chloroplast) in den Chloroplasten von Rotalgen wie Cyanidioschyzon merolae (Cyanidiales). Die histonähnlichen Proteine (HLPs, nach englisch histone like proteins) dieser drei Gruppen sind untereinander wiederum homolog, d. h., es wird ein gemeinsamer evolutionärer Ursprung der HLPs angenommen.[17][18][16]
DNA-Reparatur
In Chloroplasten des Kleinen Blasenmützenmooses (Physcomitrella patens) interagiert das DNA-Mismatch-Reparaturprotein (MMR-Protein) Msh1 mit den rekombinanten Reparaturproteinen RecA und RecG, um die Stabilität des Chloroplasten-Genoms zu erhalten.[19] In Chloroplasten der Pflanze Acker-Schmalwand (Arabidopsis thaliana) erhält das RecA-Protein die Integrität der Chloroplasten-DNA durch einen Prozess aufrecht, der wahrscheinlich die rekombinante Reparatur von DNA-Schäden beinhaltet.[20]
DNA-freie Plastiden
Im Jahr 2014 wurde sogar in der nicht photosynthetisch aktiven Grünalge Polytomella (Chlamydomonadales, syn. Volvocales) ein Plastid ohne Genom gefunden. Offenbar können Chloroplasten/Plastiden durch endosymbiotischen Gentransfer ihr gesamtes Genom verlieren.[21]
Einzelnachweise
- ↑ The Oxford Dictionary of Abbreviations. ctDNA—Dictionary definition 1998 (englisch, encyclopedia.com).
- ↑ Kim JI, Moore CE, Archibald JM, Bhattacharya D, Yi G, Yoon HS, Shin W: Evolutionary dynamics of cryptophyte plastid genomes. In: Genome Biology and Evolution. 9. Jahrgang, Nr. 7, Juli 2017, S. 1859–1872, doi:10.1093/gbe/evx123, PMID 28854597, PMC 5534331 (freier Volltext) – (englisch).
- ↑ C. Cleveland (Hrsg.); C. Michael Hogan, S. Draggan: DNA, The Encyclopedia of Earth, National Council for Science and the Environment. Washington DC, 18. Juli 2012 (via WebArchiv)
- ↑ a b c Leighton Dann: Green DNA - Simple isolation, restriction and electrophoresis of chloroplast DNA. BIOSCIENCE EXPLAINED, Science and Plants for Schools, Homerton College, Cambridge 2002 (englisch, bioscience-explained.org ( des vom 17. September 2012 im Internet Archive) [abgerufen am 22. März 2019]). (via WebArchiv)
- ↑ Chloroplasts and Other Plastids. Archiviert vom am 20. Oktober 2013; abgerufen am 22. März 2019 (englisch). (via WebArchiv)
- ↑ a b c d e f g Anna Stina Sandelius, Henrik Aronsson: The Chloroplast: Interactions with the Environment. Springer, 2009, ISBN 978-3-540-68696-5, S. 18 (englisch, google.com).
- ↑ T. Wakasugi, M. Sugita, T. Tsudzuki, M. Sugiura: Updated Gene Map of Tobacco Chloroplast DNA, in: Plant Molecular Biology Reporter, Volume 16, Issue 3, S. 231–241, September 1998. doi:10.1023/A:1007564209282
- ↑ M. T. Clegg, B. S. Gaut, G. H. Learn Jr, B. R. Morton: Rates and Patterns of Chloroplast DNA Evolution. In: Proceedings of the National Academy of Sciences. 91. Jahrgang, Nr. 15, 1994, S. 6795–6801, doi:10.1073/pnas.91.15.6795, PMID 8041699, PMC 44285 (freier Volltext), bibcode:1994PNAS...91.6795C (englisch).
- ↑ a b c d J. Shaw, E. B. Lickey, E. E. Schilling, R. L. Small: Comparison of whole chloroplast genome sequences to choose noncoding regions for phylogenetic studies in angiosperms: The tortoise and the hare III. In: American Journal of Botany. 94. Jahrgang, Nr. 3, 2007, S. 275–288, doi:10.3732/ajb.94.3.275, PMID 21636401 (englisch).
- ↑ Ron Milo, Rob Philips: Cell Biology by the Numbers: How large are chloroplasts? In: book.bionumbers.org. Abgerufen am 16. März 2019 (englisch).
- ↑ a b Jeremy Burgess: An introduction to plant cell development. Cambridge university press, Cambridge 1989, ISBN 0-521-31611-1, S. 62 (englisch, google.com).
- ↑ Arnold J. Bendich: Circular Chloroplast Chromosomes: The Grand Illusion. In: The Plant Cell Online. 16. Jahrgang, Nr. 7, 2004, S. 1661–6, doi:10.1105/tpc.160771, PMID 15235123, PMC 514151 (freier Volltext) – (englisch).
- ↑ a b c Richard Kolodner, Krishna K. Tewari: Inverted Repeats in Chloroplast DNA from Higher Plants. In: Proceedings of the National Academy of Sciences. 76. Jahrgang, Nr. 1, 1979, S. 41–5, doi:10.1073/pnas.76.1.41, PMID 16592612, PMC 382872 (freier Volltext), bibcode:1979PNAS...76...41K (englisch).
- ↑ a b Jeffrey D. Palmer, William F. Thompson: Chloroplast DNA rearrangements are more frequent when a large inverted repeat sequence is lost. In: Cell. 29. Jahrgang, Nr. 2, 1982, S. 537–50, doi:10.1016/0092-8674(82)90170-2, PMID 6288261 (englisch).
- ↑ Hans-Walter Heldt, Fiona Heldt, Birgit Piechulla: Plant Biochemistry. 3. Auflage. Academic Press, 2005, ISBN 978-0-12-088391-2, S. 517 (englisch, google.com).
- ↑ a b T. Kobayashi, M. Takahara, S. Y. Miyagishima, H. Kuroiwa, N. Sasaki, N. Ohta, M. Matsuzaki, T. Kuroiwa: Detection and Localization of a Chloroplast-Encoded HU-Like Protein That Organizes Chloroplast Nucleoids. In: The Plant Cell Online. 14. Jahrgang, Nr. 7, 2002, S. 1579–1589, doi:10.1105/tpc.002717, PMID 12119376, PMC 150708 (freier Volltext) – (englisch).
- ↑ Biology, 8th Edition, Campbell & Reece. Benjamin Cummings (Pearson), 2009, ISBN 978-0-321-54325-7, S. 516 (englisch).
- ↑ John M. Archibald: The Puzzle of Plastid Evolution. In: Current Biology. 19. Jahrgang, Nr. 2, 2009, S. R81–8, doi:10.1016/j.cub.2008.11.067, PMID 19174147 (englisch).
- ↑ Masaki Odahara, Yoshihito Kishita, Yasuhiko Sekine: MSH1 maintains organelle genome stability and genetically interacts with RECA and RECG in the moss Physcomitrella patens. In: Plant J. 91. Jahrgang, Nr. 3, August 2017, S. 455–465, doi:10.1111/tpj.13573, PMID 28407383 (englisch).
- ↑ Beth A. Rowan, Delene J. Oldenburg, Arnold J. Bendich: RecA maintains the integrity of chloroplast DNA molecules in Arabidopsis. In: J. Exp. Bot. 61. Jahrgang, Nr. 10, Juni 2010, S. 2575–88, doi:10.1093/jxb/erq088, PMID 20406785, PMC 2882256 (freier Volltext) – (englisch).
- ↑ David Roy Smith, Robert W. Lee: A Plastid without a Genome: Evidence from the Nonphotosynthetic Green Algal Genus Polytomella. In: Plant Physiology. 164. Jahrgang, Nr. 4, 1. April 2014, S. 1812–1819, doi:10.1104/pp.113.233718, PMID 24563281, PMC 3982744 (freier Volltext) – (englisch).
Auf dieser Seite verwendete Medien
Autor/Urheber: Kelvinsong, Lizenz: CC BY-SA 3.0
Gene map of tobacco chloroplast DNA. Data was taken from this paper, and input into a Open document spreadsheet to calculate the degree and radian measures, and to generate the SVG data.
Each DNA segment is identified with a path id in the source SVG(albeit not labeled with a visible text object)
Segments with labels on the outside are located on the A strand, segments with labels on the inside are located on the B strand. Segments narrower than the surrounding ones (the notches) indicate introns. Unlabeled faded segments represent open reading frames.