Nukleinbasen
| Purine | Pyrimidine | ||
 Adenin |
| ||
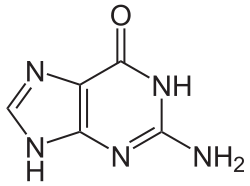
 Guanin | 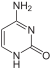 Cytosin | ||
| Strukturformeln von Nukleobasen in DNA (A,G,C,T) und RNA (A,G,C,U) – gebunden werden sie meist über die hier nach unten zeigende NH-Gruppe. | |||

Nukleinbasen, auch Nucleinbasen, Nukleobasen oder Nucleobasen (N[1]), sind ein Bestandteil von Nukleosiden und Nukleotiden und somit der Bausteine von Nukleinsäuren, in RNA wie DNA.
Als Basen werden sie bezeichnet, da sie an den Stickstoffatomen protoniert werden können und in wässriger Lösung schwach basisch reagieren. In den Nukleinsäuren sind sie meist N-glycosidisch an Ribose bzw. Desoxyribose gebunden. Über Wasserstoffbrücken zwischen Nukleinbasen können Basenpaare gebildet werden, die im Doppelstrang von DNA strukturtragend sind. Die Abfolge von Nukleobasen in einem RNA- oder DNA-Strang wird auch als Basensequenz bezeichnet.
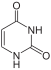
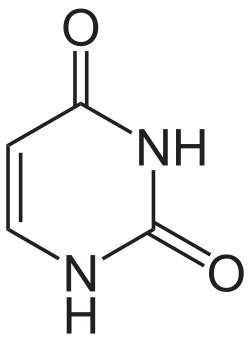
In DNA treten die vier Basen Adenin (A), Guanin (G), Cytosin (C) und Thymin (T) auf, sie werden daher auch DNA-Basen genannt. RNA enthält Uracil (U) statt Thymin, entsprechend heißen A, G, C und U auch RNA-Basen. Uracil unterscheidet sich von Thymin nur durch das Fehlen einer Methylgruppe. Das Grundgerüst von Uracil, Thymin und Cytosin ist das eines Pyrimidins, Guanin und Adenin beruhen auf dem Grundgerüst von Purin.
Bezeichnung
Den Ausdruck Nucleinbasen gebrauchte der spätere Nobelpreisträger Albrecht Kossel (1853–1927) schon 1891 als Bezeichnung für jene bei der Zersetzung von „Nucleïn“ erhaltenen und als Spaltungsprodukte der „Nucleïnsäure“ (aus Hefe) dargestellten „basischen Körper, des Adenins, Guanins und seiner Derivate, die ich alle unter dem Namen der Nucleïnbasen zusammenfassen will.“[2] 1893 entdeckte er das Thymin, und 1897 fasste er auch Cytosin unter diesen Begriff.[3] In seinem Nobelvortrag von 1910 über die chemische Beschaffenheit des Zellkerns verstand er die Nukleinbasen bereits als die vier stickstoffreichen „Bausteine“, die zusammen mit zwei weiteren andersartigen Komponenten – einem Kohlenhydrat und Phosphorsäure – das Nukleinsäure-Molekül aufbauen.[4]
Vorkommen
| Base | Kürzel | Vorkommen |
|---|---|---|
| Adenin | A | DNA, RNA |
| Guanin | G | DNA, RNA |
| Cytosin | C | DNA, RNA |
| Thymin | T | DNA |
| Uracil | U | RNA |
| Hypoxanthin | HX | DNA, RNA |
| Xanthin | X | DNA, RNA |
In der nebenstehenden Tabelle sind Namen, Abkürzungen und Vorkommen von Nukleinbasen aufgelistet. Als Teil von Nukleosiden und Nukleotiden tragen Nukleinbasen wichtige Funktionen. Zusammen mit Ribose oder Desoxyribose bilden sie Nukleoside, genauer Ribonukleoside bzw. Desoxyribonukleoside. Diese bilden mit einer zusätzlichen Phosphatgruppe als Nukleotide (Ribonukleotide bzw. Desoxyribonukleotide) wesentliche Bestandteile von Ribonukleinsäure (RNA) und Desoxyribonukleinsäure (DNA), sind aber auch in anderen wichtigen Biomolekülen enthalten.
Adenin beispielsweise tritt im Adenosin in Verbindung mit einer unterschiedlichen Anzahl an Phosphatgruppen als Adenosinmonophosphat (AMP), als cyclisches Adenosinmonophosphat (cAMP), als Adenosindiphosphat (ADP) und als Adenosintriphosphat (ATP) auf, in Verbindung mit Nicotinsäureamid in NADPH und NADH und in Verbindung mit Flavin in Flavin-Adenin-Dinukleotid (FAD), sowie als Teil von Coenzym A. Ähnliches gilt für Guanin in Guanosintriphosphat (GTP) und Cytosin in Cytidintriphosphat (CTP).
Hypoxanthin und Xanthin sind wichtige Zwischenprodukte bei der Synthese von Purinen. Sie sind weder reguläre Bestandteile von DNA oder RNA noch Elemente des genetischen Codes. Doch können sie unter Einwirkung von Mutagenen – durch Desaminierung und den Ersatz der Amino-Gruppe durch eine Hydroxygruppe sowie Umlagerung in das tautomere Keton – aus regulären Nukleobasen gebildet werden: Hypoxanthin entsteht so aus Adenin, Xanthin aus Guanin. Auf ähnliche Weise kann auch Uracil aus Cytosin entstehen.
Struktur
Purin-Basen

Das Grundgerüst von Adenin, Guanin, Hypoxanthin und Xanthin entspricht dem Purin. Deswegen werden diese Moleküle auch als Purin-Basen bezeichnet.
- Adenin
- Guanin
- Hypoxanthin
- Xanthin
Pyrimidin-Basen

Das Grundgerüst der Basen Cytosin, Uracil und Thymin ist das Pyrimidin, die deshalb auch als Pyrimidin-Basen bezeichnet werden. Ein Abkömmling von Cytosin ist beispielsweise 5-Hydroxymethylcytosin, das in der DNA mancher Bakteriophagen (Escherichia-Virus T4, englisch T-even phages) anstelle von Cytosin eingebaut ist.[5]
- Cytosin
- Uracil
- Thymin
- 5-Hydroxymethylcytosin
Basenpaarung

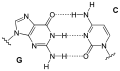
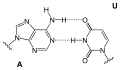
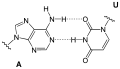
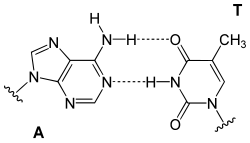
Die Purin-Base eines Nukleotids kann mit einer Pyrimidin-Base eines anderen Nukleotids ein Paar bilden, das über Wasserstoffbrücken miteinander verbunden ist. Ein solches Basenpaar bilden Guanin (G) und Cytosin (C) über drei Wasserstoffbrücken. Adenin (A) kann über zwei Wasserstoffbrücken ein Basenpaar bilden mit Thymin (T), ebenso mit Uracil (U). Die in diesen Paaren einander jeweils zugeordneten Nukleinbasen werden als komplementäre Basen bezeichnet.
Über die Bildung von Basenpaaren zwischen ihren Nukleotidbausteinen kann auch der Strang einer Nukleinsäure mit einem anderen Nukleinsäurestrang verbunden werden. Auf diese Weise können beispielsweise zwei DNA-Stränge einen DNA-Doppelstrang bilden, in dem sich jeweils die komplementären Basen des einen und des anderen Strangs gegenüberstehen (siehe Doppelhelix). Ähnlich ist über die Basenpaarung auch die Zuordnung von RNA-Nukleotiden zu den Nukleotiden eines DNA-Einzelstrangs möglich (siehe Transkription). Ebenso können zwischen Nukleotiden von RNA-Strängen Basenpaare gebildet werden, auch intramolekular im selben Strang, womit sich Strangabschnitte zu einer Haarnadelstruktur aneinanderlegen.
Mit den in DNA vorkommenden vier (DNA-)Basen – G und A sowie C und T – können komplementär gepaart die Basenpaare G-C bzw. C-G und A-T bzw. T-A gebildet werden.
- Guanin-Cytosin-Paar
- Adenin-Thymin-Paar
- Adenin-Uracil-Paar
- reverse Paarung A-U
Mit den in RNA vorkommenden vier (RNA-)Basen G, A, C und U ist in komplementärer Paarung neben G-C bzw. C-G das Basenpaar U-A bzw. A-U möglich, selten auch als reverse Paarung.
Bausteine von Nukleinsäuren

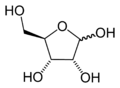
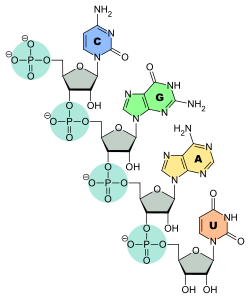
In den Nukleinsäuren treten die Nukleinbasen je gebunden an ein Zuckermolekül mit 5 C-Atomen auf, eine Pentose, die jeweils über eine Phosphatgruppe mit zwei benachbarten gleichartigen Pentosen verestert ist. Diese über Phosphodiester miteinander verbundenen Pentosemoleküle bilden das Rückgrat eines Nukleinsäurestranges, der so eine Reihe von verschiedenen Basen trägt.
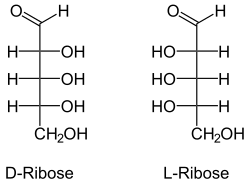
Benannt werden Nukleinsäuren nach der Art ihrer Pentosen, welche hier ringförmig als Furanosen vorliegen. Ebenso wie die Ribose in RNA kommt die 2’-Desoxyribose (englisch Deoxyribose) in DNA natürlicherweise nicht als L-Enantiomer vor. In beiden Fällen ist jeweils das β-Anomer der D-Pentose eingebaut, also β-D-Ribofuranose beziehungsweise β-2'-Desoxy-D-ribofuranose. Letztere trägt keine OH-Gruppe am C2'-Atom, sodass sie allein um ein fehlendes Sauerstoffatom verschieden ist.
- Strukturformeln von Ribose und Desoxyribose
- D-Ribose kettenförmig
als Keilstrichformel - D-Ribofuranose ringförmig als Keilstrichformel
- β-D-Ribofuranose
als Haworth-Formel
- D- und L-Desoxyribose
- 2-Desoxy-L-ribose (!) kettenförmig
- 2-Desoxy-D-ribofuranose ringförmig
- β-2-Desoxy-D-ribofuranose
Nukleoside
Die Verbindungen aus Nukleinbase plus Pentose werden Nukleoside genannt. Dazu gehören mit der Ribose als Monosaccharid und einem von der jeweiligen Base hergeleiteten Namen beispielsweise Adenosin, Guanosin, Cytidin, Thymidin und Uridin. Die mit Desoxyribose gebildeten Nukleoside sind dementsprechend Desoxyadenosin, Desoxyguanosin, Desoxycytidin, Desoxythymidin und Desoxyuridin. Die Nukleinbase wird dabei mit der Pentose jeweils in β-glycosidischer Bindung verknüpft. In der Regel geschieht dies N-glycosidisch am Stickstoffatom, also in 1-N-β-glycosidischer Bindung. Als Ausnahmen kommen auch C-glycosidische Bindungen am Kohlenstoffatom der Nukleinbase vor, so des Uracils im Pseudouridin (Ψ), das in der TΨC-Schleife einer tRNA zu finden ist. Nukleoside sind somit Glycoside, ihr Aglycon ist die Nukleinbase.
Nukleotide

In den Baueinheiten von Nukleinsäuren ist an die Pentose jeweils noch eine Phosphat-Gruppe gebunden. Diese Verbindungen aus einer Nukleinbase plus Pentose plus Phosphat werden als Nukleotide bezeichnet. Die Namen der Nukleotide in RNA ergeben sich aus denen der Nukleoside plus der Endsilbengruppe –monophosphat. Sind die Nukleotide Bausteine von DNA, so wird Desoxy- vorangestellt, zum Beispiel Desoxyadenosinmonophosphat, abgekürzt dAMP.
Nukleinsäuren sind polymere Makromoleküle, genauer Polynukleotide. Sie werden durch RNA-Polymerasen und DNA-Polymerasen aufgebaut aus reaktionsfähigen Monomeren. Diese Nukleotide sind Nukleosid-Triphosphate, zum Beispiel Desoxyadenosintriphosphat, abgekürzt dATP.
Nukleinsäuren
RNA kommt zumeist als einzelner Polynukleotid-Strang vor, kann aber auch durch Paarungen komplementärer Basen Doppelstränge bilden. Häufiger sind Schleifenbildungen infolge intramolekularer Paarungen von Strangabschnitten, die gegenläufig zueinander komplementäre Sequenzen tragen, auch Palindrome genannt.
Die Paare der Stickstoff (blau) enthaltenden Nukleinbasen stellen hier quer liegende Verbindungen zwischen den beiden längs verlaufenden Strängen dar (Kohlenstoff grün), deren Rückgrat reich an Sauerstoff (rot) ist.
DNA besteht dagegen meist nicht aus einem Polynukleotid-Strang, sondern aus zweien, von denen jeder eine Kette aus zahlreichen Nukleotiden darstellt. Die beiden Stränge sind über Basenpaare komplementär miteinander verbunden zu einem Doppelstrang (siehe Doppelhelix). Darin steht ein Adenin je einem Thymin gegenüber, ein Cytosin je einem Guanin.
Die genaue Reihenfolge der vier DNA-Basen eines Stranges wird als Basensequenz bezeichnet. In dem Muster dieser Basenfolge ist Erbinformation niedergelegt und gespeichert. Bestimmte Abschnitte codieren dabei für die Reihenfolge von Aminosäuren beim Aufbau von Proteinen. DNA-Doppelstränge lassen sich auch verdoppeln, duplizieren, indem je zu einem Strang noch ein anderer komplementärer Strang aufgebaut wird, sodass zwei identische Doppelstränge entstehen (siehe Replikation).
- Basenmodifikationen
Außer den aufgeführten primären Nukleinbasen treten verschiedene, eher seltene Abwandlungen natürlich auf. Neben den oben genannten Purin-Basen Xanthin und Hypoxanthin sind dies modifizierte Basen wie 7-Methylguanin oder, als Pyrimidin-Basen, 5-Methylcytosin, 5-Hydroxymethylcytosin und 5,6-Dihydrouracil. Mit β-D-Ribofuranose bilden diese Basen die korrespondierenden Nukleoside Xanthosin, Inosin, 7-Methylguanosin, 5-Methylcytidin, 5-Hydroxymethylcytidin und Dihydrouridin.
Daneben ermöglichen technische Synthesen durch Einführen weiterer Substituenten eine Vielzahl von Derivaten, als Basenanaloga wie 5-Fluoruracil oder auch in xDNA oder Hachimoji-DNA.
Siehe auch
Literatur
- Jeremy M. Berg, John L. Tymoczko, Lubert Stryer: Biochemie. 6. Auflage. Spektrum Akademischer Verlag, Heidelberg 2007, ISBN 978-3-8274-1800-5.
- Donald Voet, Judith G. Voet: Biochemistry. 3. Auflage. John Wiley & Sons, New York 2004, ISBN 0-471-19350-X.
- Bruce Alberts, Alexander Johnson, Peter Walter, Julian Lewis, Martin Raff, Keith Roberts: Molecular Biology of the Cell. 5. Auflage. Taylor & Francis, 2007, ISBN 978-0-8153-4106-2.
Weblinks
Einzelnachweise
- ↑ Carl R. Woese: Bacterial evolution. In: Microbiological Reviews. Band 51, Nr. 2, Juni 1987, S. 221–271, doi:10.1128/mr.51.2.221-271.1987, PMID 2439888, PMC 373105 (freier Volltext) – (Tbl. 1).
- ↑ Albrecht Kossel: Ueber die chemische Zusammensetzung der Zelle. (Vortrag am 30. Januar 1891) In: Archiv für Physiologie. Jahrgang 1891, S. 184 (online).
- ↑ Albrecht Kossel und Albert Neumann: Ueber Nucleïnsäure und Thyminsäure. In: Zeitschrift für Physiologische Chemie. Band 22, Nr. 1, Januar 1897, S. 77. doi:10.1515/bchm2.1897.22.1.74.
- ↑ Albrecht Kossel: The Chemical Composition of the Cell Nucleus. Nobel Lecture, 12. Dezember 1910.
- ↑ T-Phages. Harvard Catalyst Profiles; abgerufen am 31. Januar 2021.
Auf dieser Seite verwendete Medien
chemical structure of 5-Hydroxymethylcytosine
Struktur von Ribose
Struktur von Cytosin
Basenpaar Adenin Thymin (AT)
Struktur von Xanthin
Ribofuranose
Basenpaar Guanin Cytosin (GT)
reverses Basenpaar Adenin Uracil (AU)
Struktur von Adenin
Struktur von Pyrimidin
Struktur von Desoxyribose
Die Stereoisomere der D-Ribose in Haworth-Schreibweise
Struktur von Hypoxanthin
Basenpaar Adenin Uracil (AU)
Structure of 9H-purine
Struktur von Thymin
Chemical structure of L-deoxyribose
Autor/Urheber: Richard Wheeler (Zephyis), Lizenz: CC BY-SA 3.0
Ribose structure, illustrating the 2′, 3′ and 5′ carbon atoms.
(c) Zephyris, CC BY-SA 3.0
Strukturmodell einer DNA-Helix in B-Konformation (Animation). Die Stickstoff (blau) enthaltenden Nukleinbasen liegen waagrecht zwischen zwei Rückgratsträngen, welche sehr reich an Sauerstoff (rot) sind. Kohlenstoffatome sind grün dargestellt. Wasserstoff: weiß, Phosphor: orange
Chemical structure of D-deoxyribose
Verknüpfung der Nukleinbasen C, G, A und U mit dem Ribosephosphat-Rückgrat der RNA
Autor/Urheber: Sponk (talk), Lizenz: CC BY-SA 3.0
Gegenüberstellung eines RNA-Strangs und eines DNA-Doppelstrangs mit Darstellung der jeweiligen Nukleobasen
Autor/Urheber: Iquo, Lizenz: CC BY-SA 3.0
This image represents a chemical structure of RNA paring complementarily.