Nodaviridae
| Nodaviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
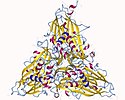
 EM-Aufnahme von GNNV-Virionen | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Nodaviridae | ||||||||||||||
| Links | ||||||||||||||
|
Nodaviridae ist die Bezeichnung einer Familie unbehüllter Einzelstrang-RNA-Viren von positiver Polarität.[2] Als natürliche Wirte dienen sowohl Wirbeltiere als auch Wirbellose. Zu den Krankheiten, die mit Viren dieser Familie in Verbindung gebracht werden, gehören virale Enzephalopathie und Retinopathie bei Fischen. Es gibt derzeit (Stand März 2021) in der Familie neun vom International Committee on Taxonomy of Viruses (ICTV) bestätigte Spezies (Arten), die sich auf zwei Gattungen verteilen.[3][4]
Etymologie
Der Name der Familie leitet sich ab von dem japanischen Dorf Nodamura in der Präfektur Iwate, wo das Nodamuravirus erstmals aus Mücken der Spezies Culex tritaeniorhynchus isoliert wurde.
Beschreibung
Aufbau



Das Virus ist nicht umhüllt und hat ein ikosaedrisches Kapsid (Triangulationszahl T=3) mit einem Durchmesser von 29 bis 35 nm. Das Kapsid ist aus 32 Kapsomeren aufgebaut.[3]
Genom



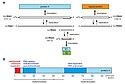
Das Genom ist segmentiert: es besteht aus zwei Segmenten, ist also bipartit. Beide Segmente sind lineare Einzelstrang-RNA positiver Polarität. Ihre Länge beträgt 3,1 kb (Kilobasen) bei Segment RNA1 und 1,4 kb bei RNA2. Das 5'-Ende hat ein methyliertes Cap, das 3'-Ende ist nicht polyadenyliert.[3][4]
RNA1 kodiert ein Protein, das mehrere funktionelle Domänen besitzt:
- mitochondriales Targeting (eine Art Adressaufkleber)
- Transmembrandomäne (um durch die Membran der Mitochondrien durchgelassen zu werden)
- RNA-abhängige RNA-Polymerase (RdRp)
- eine selbst-interagierende Domäne (englisch self-interaction domain)
- RNA-Capping (RNA-Schutzdomäne)
Zusätzlich kodiert RNA1 mir ORF 1b für eine subgenomische RNA3 (Länge 0,4 kb), die für das Protein B2, einen RNA-Silencing-Inhibitor, kodiert.[3]
RNA2 kodiert für einen viralen Kapsidprotein-Vorläufer mit Bezeichnung Protein α. Dieser Vorläufer wird der während der Virusassemblierung an einer konservierten Asn/Ala-Stelle in zwei reife Proteine, das β-Protein (mit 38 kDa – Kilodalton) und das γ-Protein (5 kDa) zerschnitten (auto-cleaved).[3]
Reproduktionszyklus
Die Virus-Replikation erfolgt im Cytoplasma. Der Eintritt in die Wirtszelle erfolgt durch Penetration. Die Replikation folgt dem Modell der für Einzelstrang-RNA-Viren positiver Polarität.[3][4]
Als natürliche Wirte dienen Wirbeltiere und Wirbellose. Die Viruspartikel werden durch Kontakt und Kontamination übertragen.[3][4]
Pathologie
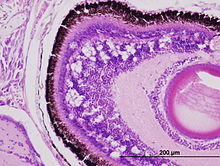
Zu den Anzeichen einer viralen Nervennekrose verursacht durch Viren der Gattung Bettanodavirus gehören: abnormales Verhalten wie Lethargie, Anorexie, Spiralschwimmen und Veränderung der Pigmentierung. Die Sterblichkeit der betroffenen Populationen kann bis zu 100 % betragen.[5] Mikroskopische Läsionen befinden sich meist im Gehirn, in der Netzhaut und im Rückenmark, wo Nekrosen der Neuronen und das Runde leere Räume, Vakuolen genannt, sind häufig mit der Krankheit assoziiert.
Systematik
Die Mitglieder der Gattung Alphanodavirus wurden ursprünglich aus Insekten isoliert, während die der Gattung Betanodavirus aus Fischen isoliert wurden. Eine kleine Anzahl von Nodoviridae scheint aber außerhalb einer dieser beiden Kladen zu liegen.[3] Das Flock House Virus (FHV) das inzwischen am besten untersuchte Mitglied der Familie, dennoch bleibt Nodamuravirus die Typusspezies der Gattung Alphanodavirus. Nach ICTV setzt sich die Familie mit Stand Mai/Juni 2024 wie folgt zusammen (ergänzt um einige Vorschläge nach NCBI):[3]

Familie Nodaviridae
- Gattung Alphanodavirus[6]
- Spezies Alphanodavirus boolarraense mit
- Boolarra virus (BoV, Boolarravirus)
- Spezies Alphanodavirus flockense mit
- Spezies Alphanodavirus heteronychi mit
- Black beetle virus (BBV)
- Spezies Alphanodavirus nodamuraense (ehem. Typus) mit
- Nodamura virus (NoV, Nodamuravirus)
- Spezies Alphanodavirus pariacotoense mit
- Pariacoto virus (PaV, Pariacotovirus)[9]
- Spezies „Alphanodavirus HB-2007/CHN“ (Vorschlag)[10]
- Spezies „Newington virus“ (NeV, „Newingtonvirus“, Vorschlag)[11]
- Spezies Alphanodavirus boolarraense mit

- Gattung Betanodavirus (früher Nervous necrosis virus, NNV)[12][13]
- Spezies Betanodavirus epinepheli mit
- Redspotted grouper nervous necrosis virus (RGNNV, Zackenbarsch-Nervennekrose-Virus), Isolat SGWak97
- Dragon grouper nervous necrosis virus (DGNNV), Isolat China
- Greasy grouper nervous necrosis virus (GGNNV), Isolat Singapore
- Japanese flounder nervous necrosis virus (JFNNV), Isolate WD und JF-HI93
- Lates calcarifer encephalitis virus (LcEV), Isolat T442286
- Malabaricus grouper nervous necrosis virus (MGNNV)
- Seabass nervous necrosis virus (SBNNV)
- Spezies Betanodavirus pseudocarangis (ehem. Typus) mit
- Striped jack nervous necrosis virus (SJNNV)
- Solea senegalensis nervous necrosis virus (SSNNV), Isolate SpSsIAusc16003 und 03-160
- Spezies Betanodavirus takifugui mit
- Tiger puffer nervous necrosis virus (TPNNV), Isolat TPKag93
- Spezies Betanodavirus verasperi mit
- Barfin flounder nervous necrosis virus (BFNNV), Isolat BF93Hok
- Atlantic cod nervous necrosis virus (ACNNV), Isolat GmMR11/06
- Atlantic halibut nodavirus (AHNV), Isolate AH99NorA und AH95NorA
- vorgeschlagene Spezies siehe NCBI[14]
- Spezies Betanodavirus epinepheli mit
- ohne Gattungszuordnung
Einzelnachweise
- ↑ a b c d e ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ↑ A. S. Sahul Hameed, A. S. Ninawe, T. Nakai, S. C. Chi, K. L. Johnson, Consortium ICTV Report: ICTV Virus Taxonomy Profile: Nodaviridae. In: The Journal of General Virology. 100. Jahrgang, Nr. 1, Januar 2019, S. 3–4, doi:10.1099/jgv.0.001170, PMID 30431412.
- ↑ a b c d e f g h i ICTV Report Nodaviridae.
- ↑ a b c d e Viral Zone. Expasy
- ↑ Misao Arimoto, Jun Sato, Keigo Maruyama, Gen Mimura, Iwao Furusawa: Effect of chemical and physical treatments on the inactivation of striped jack nervous necrosis virus (SJNNV). In: Aquaculture. 143. Jahrgang, Nr. 1, 1996, S. 15–22, doi:10.1016/0044-8486(96)01261-6.
- ↑ SIB: Alphanodavirus. Auf: ViralZone.
- ↑ Nuruddin Unchwaniwala, Hong Zhan, Janice Pennington, Mark Horswill, Johan A. den Boon, Paul Ahlquist: Subdomain cryo-EM structure of nodaviral replication protein A crown complex provides mechanistic insights into RNA genome replication. In: Proceedings of the National Academy of Sciences. 20. Juli 2020, doi:10.1073/pnas.2006165117, PMID 32690711 (pnas.org [PDF]).
- ↑ NCBI: Flock House virus
- ↑ Batsal Devkota et al.: Structural and electrostatic characterization of pariacoto virus: implications for viral assembly. In: Biopolymers, Band 91, Nr. 7, Juli 2009, S. 530–538; doi:10.1002/bip.21168, PMID 19226622 (englisch).
- ↑ NCBI: Alphanodavirus HB-2007/CHN (species)
- ↑ NCBI: Newington virus (species)
- ↑ SIB: Betanodavirus. Auf: ViralZone.
- ↑ Charlotte Axén, Niccolò Vendramin, Anna Toffan: Outbreak of Mortality Associated with Acipenser Iridovirus European (AcIV-E) Detection in Siberian Sturgeon (Acipenser baerii) Farmed in Sweden , in: MDPI Fishes 2018, Band 3, Nr. 4, Special Issue: Viral Diseases of Fish and Shellfish, 42, 16. Oktober 2018, doi:10.3390/fishes3040042
- ↑ NCBI: unclassified Betanodavirus (list)
- ↑ Carl J. Franz, Hilary Renshaw, Lise Frezal, Yanfang Jiang, Marie-Anne Félix, David Wang: Orsay, Santeuil and Le Blanc viruses primarily infect intestinal cells in Caenorhabditis nematodes. In: Virology, Band 448, 5. Januar 2014, S. 255–264; doi:10.1016/j.virol.2013.09.024, PMID 24314656, Epub 1. November 2013 (englisch).
- ↑ NCBI Taxonomy Browser: Orsay virus (species, equivalent: Orsay nodavirus).
- ↑ Orsay virus. Auf: Virus-Host DB.
- ↑ Hongbing Jiang, Carl J. Franz, Guang Wu, Hilary Renshaw, Guoyan Zhao, Andrew E. Firth, David Wang: Orsay virus utilizes ribosomal frameshifting to express a novel protein that is incorporated into virions. In: Virology, Band 450–451, Nr. 100, 2. Februar 2014, S. 213–221; doi:10.1016/j.virol.2013.12.016, PMC 3969245 (freier Volltext), PMID 24503084 (englisch).
- ↑ NCBI Taxonomy Browser: Santeuil nodavirus (species).
- ↑ NCBI Taxonomy Browser: Le Blanc nodavirus (species).
- ↑ Carl J. Franz, Guoyan Zhao, Marie-Anne Félix, David Wang: Complete Genome Sequence of Le Blanc Virus, a Third Caenorhabditis Nematode-Infecting Virus. In: American Society for Microbiology Journals: Journal of Virology, Band 86, Nr. 21, 6. Oktober 2012; doi:10.1128/JVI.02025-12, PMID 23043172, PMC 3486331 (freier Volltext) (englisch).
- ↑ D. Qian et al.: Extra small virus-like particles (XSV) and nodavirus associated with whitish muscle disease in the giant freshwater prawn, Macrobrachium rosenbergii. In: Journal of Fish Diseases, Band 26, Nr. 9, September 2003, S. 521–527; doi:10.1046/j.1365-2761.2003.00486.x, PMID 14575370 (englisch).
- ↑ J. Widada, S. Widada, J. R. Bonami: Characteristics of the monocistronic genome of extra small virus, a virus-like particle associated with Macrobrachium rosenbergii nodavirus: possible candidate for a new species of satellite virus. In: Journal of General Virology, Band 85, Nr. 3, März 2004, S. 643–646; doi:10.1099/vir.0.79777-0, PMID 14993649 (englisch).
- ↑ NCBI Taxonomy Browser: Macrobrachium rosenbergii nodavirus (species).
- ↑ SIB: Macronovirus. Auf: ViralZone.
- ↑ NCBI Taxonomy Browser: unclassified Nodaviridae (Liste).
Weblinks
Auf dieser Seite verwendete Medien
Autor/Urheber:
Deposition authors: Natarajan, P., Johnson, J.E.;
visualization author: User:Astrojan, Lizenz: CC BY 4.0
Nodamura virus coat protein trimer (3 beta and 3 gamma chain, blue)
Autor/Urheber: ViralZone, SIB Swiss Institute of Bioinformatics, Philippe Le Mercier et al., Lizenz: CC BY 4.0
Genomkarte der Familie Nodaviridae
Autor/Urheber: A. S. Sahul Hameed, A. S. Ninawe, T. Nakai, S. C. Chi, K. L. Johnson, Lizenz: CC BY-SA 4.0
Genome organization of Macrobrachium rosenbergii nodavirus (MrNV). RdRP: RNA-dependent RNA polymerase, CP: capsid protein. Numbers indicate nucleotide positions.
Autor/Urheber: Nai-Chi Chen, Masato Yoshimura, Hong-Hsiang Guan, Ting-Yu Wang, Yuko Misumi, Chien-Chih Lin, Phimonphan Chuankhayan, Atsushi Nakagawa, Sunney I. Chan, Tomitake Tsukihara, Tzong-Yueh Chen, Chun-Jung Chen, Lizenz: CC BY 4.0
Electron micrographs of negatively stained grouper nervous necrosis virus like particles used for crystallization. (A) T = 3 GNNV-LPs; (B) T = 1 SVPs of the N-ARM deletion mutant; (C) the delta-P-domain mutant. Bar: 100 nm.
Autor/Urheber: Kenneth J Ertel, Desirée Benefield, Daniel Castaño-Diez, Janice G Pennington, Mark Horswill, Johan A den Boon, Marisa S Otegui, and Paul Ahlquist, Lizenz: CC BY 4.0
Flock house virus genome organization and functional map of replicase protein A. (A) The bipartite FHV RNA genome encodes the viral replicase protein A on RNA1 and the capsid protein precursor on RNA2. Protein A mediates synthesis of a negative strand copy of the genomic RNAs which serves as template for synthesis of progeny positive strand RNAs, and an additional partial negative strand copy of RNA1 to template a 3’ co-terminal subgenomic RNA3. RNA3 potentially encodes protein B1, co-terminal with the C-terminus of protein A, and protein B2 which has anti-RNAi function. (B) A linear representation of protein A annotated to show membrane-association and enzymatic domains, and several regions independently capable of homotypic multimerization.
Autor/Urheber: Natarajan, P., Johnson, J.E., Lizenz: CC0
X-ray diffraction of nodamura virus
Autor/Urheber: Nai-Chi Chen, Masato Yoshimura, Hong-Hsiang Guan, Ting-Yu Wang, Yuko Misumi, Chien-Chih Lin, Phimonphan Chuankhayan, Atsushi Nakagawa, Sunney I. Chan, Tomitake Tsukihara, Tzong-Yueh Chen, Chun-Jung Chen, Lizenz: CC BY 4.0
The overall structure of grouper nervous necrosis virus (GNNV). Surface domain-colored diagram (left) and central cavity (right) representations of the T = 3 GNNV-LP. The tip-to-tip distance is ~350 Å, the diameter of the central cavity is ~228 Å, and the spike protrusion on the capsid surface is ~47 Å. The S-domains of the subunits A, B and C are shown in orange, blue and red, respectively, and the P-domains are shown in cyan. The structure of the GNNV-LP is viewed along the I2, I3 and I5 axes.
Autor/Urheber: Dianajmt, Lizenz: CC BY 3.0
microphotography of histological section of retina stained with HE of Australian bass larvae experimentally infected with NNV
Autor/Urheber: ViralZone, SIB Swiss Institute of Bioinformatics, Philippe Le Mercier et al., Lizenz: CC BY 4.0
Schemazeichnung eines Virions der Nodaviridae (Querscnitt und Seitenansicht)
Autor/Urheber: Nai-Chi Chen, Masato Yoshimura, Hong-Hsiang Guan, Ting-Yu Wang, Yuko Misumi, Chien-Chih Lin, Phimonphan Chuankhayan, Atsushi Nakagawa, Sunney I. Chan, Tomitake Tsukihara, Tzong-Yueh Chen, Chun-Jung Chen, Lizenz: CC BY-SA 4.0
EM image and the overall structure of grouper nervous necrosis virus (GNNV). A representative negative-staining EM image of the purified GNNV-LPs after self-assembly.