Humanes Coronavirus HKU1
| Humanes Coronavirus HKU1 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
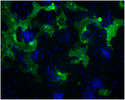
 Bildung großer, mehrkerniger Riesenzellen (Synzytien) von menschlichen alveolären Zellen, die mit HCoV-HKU1 infiziert sind. Die Zellen wurden mit einem speziellen Verfahren per Fluorescein grün, die Zellkerne mit DAPI blau gefärbt. Eine effiziente Infektion mit der Ausbreitung von Zelle zu Zelle und der Bildung von Synzytien ist deutlich zu erkennen. | ||||||||||||||||||||||||
| Systematik | ||||||||||||||||||||||||
| ||||||||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||||||||
| ||||||||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||||||||
| Human coronavirus HKU1 | ||||||||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||||||||
| HCoV-HKU1 | ||||||||||||||||||||||||
| Links | ||||||||||||||||||||||||
|
Das Humane Coronavirus HKU1 (wissenschaftlich Human coronavirus HKU1, HCoV-HKU1) ist eine Art (Spezies) in der Virus-Familie Coronaviridae. Beim Menschen führt eine Infektion zu einer Erkrankung der oberen Atemwege mit Symptomen der Erkältung, kann jedoch auch zu Lungenentzündung und Bronchiolitis führen. Das Virus stammt ursprünglich von infizierten Mäusen.[3][4] Es wurde erstmals im Januar 2005 bei zwei Patienten in Hongkong entdeckt.[5][6][7] Nachfolgende Untersuchungen ergaben, dass es eine globale Verbreitung aufweist und schon früher entstanden sein muss.
Das Virus ist behüllt und hat ein Genom aus einer Einzelstrang-RNA mit positiver Polarität. Die Virusteilchen (Virionen) treten in die Wirtszelle ein, indem sie an ihre N-Acetylneuraminsäure-Rezeptoren binden.[8] HCoV-HKU1 hat das Hämagglutinin-Esterase-Gen (HE-Gen, englisch hemagglutinin esterase gen), das es als Mitglied der Gattung Betacoronavirus und Untergattung Embecovirus (Equine und Murine Betacoronaviren) ausweist.[9]
Entdeckung
HCoV-HKU1 wurde erstmals im Januar 2005 von Woo et al. bei einem 71-jährigen Mann in Hongkong identifiziert, der mit einer akuten Atemnot und einer radiologisch bestätigten zweiseitigen Lungenentzündung (bilateralen Pneumonie) ins Krankenhaus eingeliefert wurde. Der Patient war kurz zuvor aus Shenzhen (Festlandchina) nach Hongkong zurückgekehrt.[10][5]
Systematik
Äußere Systematik
HCoV-HKU1 ist eines von sieben bekannten Coronaviren, die Menschen infizieren, neben HCoV-229E, HCoV-NL63, HCoV-OC43 – diese vier summarisch auch englisch common cold Coronaviruses (ccCoVs) genannt;[11] sowie MERS-CoV, dem ursprünglichen SARS-CoV (oder SARS-CoV-1) und SARS-CoV-2 (dem Erreger von COVID-19).
Woo et al. waren zwar erfolglos bei ihren Versuchen, eine Zelllinie mit HCoV-HKU1 zu züchten. Sie konnten jedoch die vollständige Genomsequenz ermitteln. Die phylogenetische Analyse zeigte, dass HCoV-HKU1 am engsten mit dem Maus-Hepatitis-Virus (MHV, engl. „murine hepatitis virus“, Spezies Murine coronavirus[12]) verwandt ist. In derselben Spezies befindet sich auch das puffinosis coronavirus (PV), das Atlantiksturmtaucher (Puffinus puffinus) befällt. Etwas weiter verwandt ist die Spezies Betacoronavirus 1 mit dem humanpathogenen Vertreter HCoV-OC43.[5] Alle diese Coronaviren gehören zur Untergattung Embecovirus.
Bei der Analyse der Gene RNA-abhängigen RNA-Polymerase (RdRp), Spike (S) und Nukleokapsid (N) wurden widersprüchliche phylogenetische Beziehungen entdeckt. Die vollständige Genomsequenzierung von 22 Stämmen von HCoV-HKU1 bestätigte, dass dies auf eine natürliche Rekombination (Reassortment) zurückzuführen war.[13] Diese Ergebnisse bestätigen einmal mehr, dass Coronaviren diese Fähigkeit besitzen, obwohl ihr RNA-Genom im Gegensatz zu Influenza-A-Viren aus nur einem einzigen Stück besteht (unsegmentiert oder monopartit). Einen solchen Vorgang muss es auch bei der Entstehung des ursprünglichen SARS-Virus SARS-CoV-1 gegeben haben. Auch bei der Entstehung von SARS-CoV-2, dem Erreger von COVID-19, wird ähnliches vermutet. Voraussetzung ist in jedem Fall – wie beim Influenza-A-Virus – die Doppelinfektion eines Wirts mit genetisch ähnlichen Virusstämmen.
Innere Systematik
Nach NCBI werden heute drei Stämme oder Isolate N1 bis N3 der Spezies HCoV-HKU1 unterschieden:[14]
Epidemiologie
Eine Rückverfolgungsanalyse von SARS-CoV-negativen nasopharyngealen Aspiraten (das sind Proben aus dem Nasen-Rachenraum mit eingeatmetem Fremdmaterial) von Patienten mit Atemwegserkrankungen während der SARS-Pandemie 2002/2003 ergab bei einer Probe einer 35-jährigen Patientin mit Lungenentzündung den Nachweis von RNA von CoV-HKU1.[5]
Nach den ersten Berichten über die Entdeckung von HCoV-HKU1 wurde das Virus im selben Jahr bei 10 Patienten in Nordaustralien identifiziert. Atemproben wurden zwischen Mai und August (d. h. im australischen Winter) entnommen. Dabei zeigte sich, dass die meisten HCoV-HKU1-positiven Proben in den späteren Wintermonaten von Kindern stammten.[15]
Die ersten bekannten Fälle in der westlichen Hemisphäre wurden 2005 durch Virologen am Yale-New Haven Hospital, New Haven (Connecticut) entdeckt. Sie führten eine Studie mit Proben durch, die in einem Zeitraum von 7 Wochen (im Dezember 2001 bis Februar 2002) bei 851 Säuglingen und Kindern gesammelt worden waren. Diese Kinder hatten zum Zeitpunkt der Probenentnahme Infektionen der Atemwege (bei einem Mädchen so schwer, dass eine mechanische Beatmung erforderlich war). Proben von neun Kindern waren auf HCoV-HKU1 positiv (vorhanden) getestet worden, hingegen fielen Tests auf andere mögliche Ursachen wie das
- Humane Respiratorische Synzytialvirus (HRSV),
- Parainfluenzaviren (Typ 1–3) (HPIV-1 – -3)
- Influenza-A- und -B-virus (FLUA, FLUB) und
- Humane Adenoviren (HAdV)
durch direkte Immunfluoreszenztests, sowie
- Humanes Metapneumovirus (HMPV) und
- New Haven Coronavirus (HCoV-NH, einem Stamm der Spezies Humanes Coronavirus NL63, HCoV-NL63)[16]
durch Reverse-Transkriptase-Polymerase-Kettenreaktion (englisch reverse transcription polymerase chain reaction, RT-PCR) negativ aus.
Die dabei in New Haven gefundenen Stämme sind daher insbesondere nicht mit dem Stamm HCoV-NH (New Haven Coronavirus) zu verwechseln, einem Stamm von HCoV-NL63. Stattdessen fanden die Forscher, dass die im Rahmen dieser Studie in New Haven identifizierten Stämme den in Hongkong gefundenen Stämmen ähnlich waren, was eine weltweite Verbreitung von HCoV-HKU1 nahelegt.[17]
Im Juli 2005 wurden in Frankreich sechs weitere Fälle gemeldet. Die französischen Forscher verwendeten verbesserte Techniken zur Gewinnung des Virus aus nasopharyngealen Aspiraten und aus Stuhlproben.[5]
Einzelnachweise
- ↑ a b c d ICTV: ICTV Taxonomy history: Severe acute respiratory syndrome-related coronavirus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ ICTV Master Species List 2018b v2 MSL #34v, März 2019.
- ↑ Yvonne Xinyi Lim, Yan Ling Ng, James P. Tam, Ding Xiang Liu: Human Coronaviruses: A Review of Virus–Host Interactions. In: Diseases. 4. Jahrgang, Nr. 3, 25. Juli 2016, ISSN 2079-9721, S. 26, doi:10.3390/diseases4030026, PMID 28933406, PMC 5456285 (freier Volltext): „See Table 1.“
- ↑ David Cyranoski: Virologie: Porträt eines Killers, Online-Ausgabe des Artikels in Spektrum der Wissenschaft Nr. 8, August 2020, S. 40–49
- ↑ a b c d e S. K. P. Lau, P. C. Y. Woo, C. C. Y. Yip, H. Tse, H.-w. Tsoi, V. C. C. Cheng, P. Lee, B. S. F. Tang, C. H. Y. Cheung, R. A. Lee, L.-y. So, Y.-l. Lau, K.-h. Chan, K.-y. Yuen: Coronavirus HKU1 and Other Coronavirus Infections in Hong Kong. In: Journal of Clinical Microbiology. 44. Jahrgang, Nr. 6, 2006, S. 2063–2071, doi:10.1128/JCM.02614-05, PMID 16757599, PMC 1489438 (freier Volltext).
- ↑ A. Vabret, J. Dina, S. Gouarin, J. Petitjean, S. Corbet, F. Freymuth: Detection of the New Human Coronavirus HKU1: A Report of 6 Cases. In: Clinical Infectious Diseases. 42. Jahrgang, Nr. 5, 2006, S. 634–639, doi:10.1086/500136, PMID 16447108.
- ↑ Alex Knapp: The secret history of the first coronavirus 229E, in Forbes vom 12. April 2020
- ↑ Yvonne Xinyi Lim, Yan Ling Ng, James P. Tam, Ding Xiang Liu: Human Coronaviruses: A Review of Virus–Host Interactions. In: Diseases. 4. Jahrgang, Nr. 3, 25. Juli 2016, ISSN 2079-9721, S. 26, doi:10.3390/diseases4030026, PMID 28933406, PMC 5456285 (freier Volltext): „See Table 1.“
- ↑ Patrick C. Y. Woo, Yi Huang, Susanna K. P. Lau, Kwok-Yung Yuen: Coronavirus Genomics and Bioinformatics Analysis. In: Viruses. 2. Jahrgang, Nr. 8, 24. August 2010, ISSN 1999-4915, S. 1804–1820, doi:10.3390/v2081803, PMID 21994708, PMC 3185738 (freier Volltext): „In all members of Betacoronavirus subgroup A, a haemagglutinin esterase (HE) gene, which encodes a glycoprotein with neuraminate O-acetyl-esterase activity and the active site FGDS, is present downstream to ORF1ab and upstream to S gene. See Figure 1.“
- ↑ P. C. Y. Woo, S. K. P. Lau, C.-m. Chu, K.-h. Chan, H.-w. Tsoi, Y. Huang, B. H. L. Wong, R. W. S. Poon, J. J. Cai, W.-k. Luk, L. L. M. Poon, S. S. Y. Wong, Y. Guan, J. S. M. Peiris, K.-y. Yuen: Characterization and Complete Genome Sequence of a Novel Coronavirus, Coronavirus HKU1, from Patients with Pneumonia. In: Journal of Virology. 79. Jahrgang, Nr. 2, 2004, S. 884–895, doi:10.1128/JVI.79.2.884-895.2005, PMID 15613317, PMC 538593 (freier Volltext).
- ↑ Alexandra M. Johansson: Cross-reactive and mono-reactive SARS-CoV-2 CD4+ T cells in prepandemic and COVID-19 convalescent individuals. In: PLOS Pathogens, Band 17, Nr. 2, 29. Dezember 2021, S. 1010203; doi:10.1371/journal.ppat.1010203, PDF.
- ↑ Raoul J. de Groot: Revision of the family Coronaviridae. In: International Committee on Taxonomy of Viruses (ICTV). 2009, S. 36, abgerufen am 23. Januar 2020 (englisch): „Murine coronavirus (new) (comprised of existing species Murine hepatitis virus, Rat coronavirus and puffinosis virus) […] Species Murine hepatitis virus; Puffinosis coronavirus; Rat coronavirus (these are to be united in a new species Murine coronavirus in a new genus Betacoronavirus)“
- ↑ P. C. Y. Woo, S. K. P. Lau, C. C. Y. Yip, Y. Huang, H.-W. Tsoi, K.-H. Chan, K.-Y. Yuen: Comparative Analysis of 22 Coronavirus HKU1 Genomes Reveals a Novel Genotype and Evidence of Natural Recombination in Coronavirus HKU1. In: Journal of Virology. 80. Jahrgang, Nr. 14, 2006, S. 7136–45, doi:10.1128/JVI.00509-06, PMID 16809319, PMC 1489027 (freier Volltext) – (hku.hk [PDF]).
- ↑ NCBI: Human coronavirus HKU1 (species)
- ↑ T. Sloots, P. McErlean, D. Speicher, K. Arden, M. Nissen, I. MacKay: Evidence of human coronavirus HKU1 and human bocavirus in Australian children. In: Journal of Clinical Virology. 35. Jahrgang, Nr. 1, 2006, S. 99–102, doi:10.1016/j.jcv.2005.09.008, PMID 16257260.
- ↑ Lia van der Hoek, Ben Berkhout: Questions concerning the New Haven Coronavirus, The Journal of Infectious Diseases 192(2):350-351, August 2005; doi:10.1086/430795, PMID 15962232
- ↑ Frank Esper, Carla Weibel, David Ferguson, Marie L. Landry, Jeffrey S. Kahn: Coronavirus HKU1 Infection in the United States. In: Emerging Infectious Diseases. 12. Jahrgang, Nr. 5, 2006, S. 775–779, doi:10.3201/eid1205.051316, PMID 16704837, PMC 3374449 (freier Volltext) – (medscape.com).
Weblinks
- Krzysztof Pyrc, Ben Berkhout, Lia van der Hoek: The Novel Human Coronaviruses NL63 and HKU1, in: Journal of Virology 81 (7), März 2007, S. 3051-3057, doi:10.1128/JVI.01466-06
- Anthony King: What four coronaviruses from history can tell us about covid-19, auf: New Scientist (newscientist.com) vom 29. April 2020
Auf dieser Seite verwendete Medien
Autor/Urheber: © 2013 Dominguez et al., Lizenz: CC BY 4.0
Formation of large syncytia of primary human alveolar type II cells infected with HCoV-HKU1. Cells were inoculated with a 1∶10 dilution of the clinical isolate HKU1/DEN/2010/21 at 34oC and fixed 120 hours post infection. Type II cell cultures were immunolabeled with polyclonal rabbit antibodies to purified HCoV-HKU1 spike protein and fluorescein labeled anti rabbit IgG (green). Nuclei were stained with DAPI (blue). Viral antigen is seen only within the cytoplasm of the cells. Efficient infection with cell to cell spread and formation of large, multinucleated giant cells is clearly evident.