Gelextraktion
Die Gelextraktion umfasst biochemische Methoden zur Isolierung von Biomolekülen wie DNA, RNA und Proteine aus Agarosegelen oder Polyacrylamidgelen.
Prinzip

Das Gelstück mit der zu untersuchenden, gefärbten Bande wird per Skalpell oder Stanze aus dem Gel herausgeschnitten und mit einer der folgenden Methoden behandelt. Die Methoden lassen sich nach ihrem jeweiligen Trennprinzip unterteilen. Im Falle einer Verwendung von Fluoreszenzfarbstoffen wird eine Bestrahlung mit UV-Licht, z. B. auf einem Transilluminator (UV-Leuchttisch), so kurz wie nötig gehalten, da die Bestrahlung unter anderem zur Bildung von Thymindimeren oder Strangbrüchen führen kann. Die durch UV-Licht beschädigten Nukleinsäuren können teilweise nachfolgende Reaktionen hemmen. Bei der Verwendung von nichtfluoreszierenden Farbstoffen wie Methylenblau oder Kristallviolett erfolgt das Herausschneiden ohne UV-Transilluminator unter Tageslicht. Bei Verwendung eines Kristallviolett-gefärbten Gels sollte im Probenpuffer kein Bromphenolblau oder Xylencyanolblau enthalten sein, da die elektrophoretische Trennung unter einer Wechselwirkung dieser Farbstoffe leidet.[1]
Extraktion
Agarosegele können durch Zugabe von einem Puffer mit Chaotropen und eine Erwärmung auf etwa 55 °C aufgelöst werden. Die chaotropen Salze denaturieren die Proteine und lösen die Agarose-Gelmatrix auf und halten sie in Lösung.[2] Als Chaotrop wird meistens Natriumiodid verwendet.[3] In der vorangehenden Agarosegelelektrophorese und im Gel sollte für eine chemische Gelextraktion kein TBE-Puffer verwendet werden, da das enthaltene Borat die chaotrope Wirkung des Natriumiodids mindert. Das aufgelöste Gel wird anschließend in einer DNA-Extraktion eingesetzt, um die enthaltene DNA zu isolieren.
In der Massenspektrometrie werden nach einem In-Gel-Verdau die gespaltenen Proteinfragmente aus Polyacrylamidgelen extrahiert,[4][5][6][7] z. B. mit einem Ammoniumhydrogencarbonat-Puffer oder mit einer Lösung von Ameisensäure, Acetonitril und teilweise auch Isopropanol.[8][9]
Gefrieren
Das Gelstück wird bei −20 °C eingefroren. Anschließend wird das Gelstück in ein Stück Kunststofffolie (meistens Parafilm) eingefaltet. Durch Quetschen des gefrorenen Gelstücks in der Folie wird das Gel durch die enthaltenen Eiskristalle zerkleinert. Die daraus freigesetzte Flüssigkeit mit dem zu untersuchenden Molekül wird anschließend in ein neues Reaktionsgefäß überführt. Daher wird die Methode auch als Freeze and Squeeze (engl. für ‚Frieren und Quetschen‘) bezeichnet.[10][11] Zur Vermeidung einer unerwünschten Verschleppung von Gelresten wird gegebenenfalls die Lösung bis zu 30 Sekunden zentrifugiert und die überstehende Lösung in ein neues Reaktionsgefäß überführt. Die Lösung wird anschließend zur Ethanolfällung eingesetzt, um die enthaltene DNA zu isolieren.
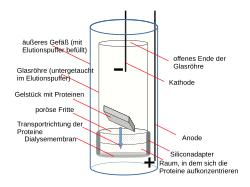
Elektroelution

Die Elektroelution erfolgt in einer Gelelutions-Elektrophoresekammer unter Anlegen einer Spannung.[12][13] Zu diesem Zweck werden die Gelstücke mit einem kleinen Volumen des Elektrophoresepuffers in einen Dialyseschlauch überführt. Alternativ kommen Apparaturen verschiedener Hersteller zur Anwendung. Ebenso wie geladene Moleküle während einer Gelelektrophorese in ein Gel einwandern, können sie aus dem Gel auch in eine Lösung herauswandern.
Literatur
- Friedrich Lottspeich, Joachim W. Engels, Solodkoff Zettlmeier Lay: Bioanalytik. 3. Auflage, Spektrum, Heidelberg, 2012, ISBN 978-3-8274-0041-3.
- Hubert Rehm, Thomas Letzel: Der Experimentator: Proteinbiochemie / Proteomics. 6. Auflage, Spektrum, Heidelberg 2009, ISBN 978-3-8274-2312-2.
- Cornel Mülhardt: Der Experimentator: Molekularbiologie/Genomics. Sechste Auflage. Spektrum Akademischer Verlag, Heidelberg 2008, ISBN 3-8274-2036-9.
- J. Sambrook, T. Maniatis, D. W. Russel: Molecular cloning: a laboratory manual. 3rd edition, Cold Spring Harbor Laboratory Press, 2001, ISBN 0-87969-577-3.
Einzelnachweise
- ↑ N. K. Rand: Crystal Violet Can Be Used to Visualize DNA Bands During Gel Electrophoresis and to Improve Cloning Efficiency. In: Elsevier Trends Journals Technical Tips Online (1996), T40022.
- ↑ T. A. Borodina, H. Lehrach, A. V. Soldatov: DNA purification on homemade silica spin-columns. In: Anal Biochem. Band 321, Nr. 1, 2003, S. 135–7, PMID 12963065.
- ↑ B. Vogelstein, D. Gillespie: Preparative and analytical purification of DNA from agarose. In: Proc Natl Acad Sci U S A. Band 76, Nr. 2, 1979, S. 615–619, PMID 284385, PMC 382999 (freier Volltext).
- ↑ A. Shevchenko, M. Wilm, O. Vorm, M. Mann: Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. In: Analytical chemistry. Band 68, Nummer 5, März 1996, ISSN 0003-2700, S. 850–858, PMID 8779443.
- ↑ F. Gharahdaghi, C. R. Weinberg, D. A. Meagher, B. S. Imai, S. M. Mische: Mass spectrometric identification of proteins from silver-stained polyacrylamide gel: a method for the removal of silver ions to enhance sensitivity. In: Electrophoresis. Band 20, Nummer 3, März 1999, ISSN 0173-0835, S. 601–605, doi:10.1002/(SICI)1522-2683(19990301)20:3<601::AID-ELPS601>3.0.CO;2-6, PMID 10217175.
- ↑ J. Havlis, H. Thomas, M. Sebela, A. Shevchenko: Fast-response proteomics by accelerated in-gel digestion of proteins. In: Analytical chemistry. Band 75, Nummer 6, März 2003, ISSN 0003-2700, S. 1300–1306, PMID 12659189.
- ↑ C. R. Jiménez, L. Huang, Y. Qiu, A. L. Burlingame: In-gel digestion of proteins for MALDI-MS fingerprint mapping. In: Current protocols in protein science / editorial board, John E. Coligan ... [et al.]. Chapter 16, Mai 2001, S. Unit 16.4, ISSN 1934-3663, doi:10.1002/0471140864.ps1604s14, PMID 18429131.
- ↑ A. Shevchenko, H. Tomas, J. Havlis, J. V. Olsen, M. Mann: In-gel digestion for mass spectrometric characterization of proteins and proteomes. In: Nature protocols. Band 1, Nummer 6, 2006, ISSN 1750-2799, S. 2856–2860, doi:10.1038/nprot.2006.468, PMID 17406544.
- ↑ H. Ehring, S. Strömberg, A. Tjernberg, B. Norén: Matrix-assisted laser desorption/ionization mass spectrometry of proteins extracted directly from sodium dodecyl sulphate-polyacrylamide gels. In: Rapid communications in mass spectrometry : RCM. Band 11, Nummer 17, 1997, ISSN 0951-4198, S. 1867–1873, doi:10.1002/(SICI)1097-0231(199711)11:17<1867::AID-RCM46>3.0.CO;2-Z, PMID 9404036.
- ↑ R. W. Thuring, J. P. Sanders, P. Borst: A freeze-squeeze method for recovering long DNA from agarose gels. In: Analytical biochemistry. Band 66, Nummer 1, Mai 1975, ISSN 0003-2697, S. 213–220, PMID 1096670.
- ↑ D. Tautz, M. Renz: An optimized freeze-squeeze method for the recovery of DNA fragments from agarose gels. In: Analytical biochemistry. Band 132, Nummer 1, Juli 1983, ISSN 0003-2697, S. 14–19, PMID 6312834.
- ↑ H. Seelert, F. Krause: Preparative isolation of protein complexes and other bioparticles by elution from polyacrylamide gels. In: Electrophoresis. Band 29, Nummer 12, Juni 2008, ISSN 0173-0835, S. 2617–2636, doi:10.1002/elps.200800061, PMID 18494038.
- ↑ D. Moore, J. Chory, R. K. Ribaudo: Isolation and purification of large DNA restriction fragments from agarose gels. In: Current protocols in immunology / edited by John E. Coligan ... [et al.]. Chapter 10Mai 2001, S. Unit 10.5, ISSN 1934-368X, doi:10.1002/0471142735.im1005s08, PMID 18432696.
Auf dieser Seite verwendete Medien
Autor/Urheber: Andi schmitt, Lizenz: CC BY-SA 4.0
Ein Gelstück mit der/den interessierenden Proteinbande(n) wird in eine Elektroelutionskammer gegeben und Gleichstrom angelegt. Da die Proteine aufgrund der Behandlung mit Natriumdodecylsulfat (SDS) in der Regel negativ geladen vorliegen, wandern sie in Richtung Pluspol. So verlassen sie das Gel und werden durch eine poröse Membran (Fritte) transportiert. Sie sammeln sich in dem folgenden Flüssigkeitsraum, da sie die Dialysemembran aufgrund ihrer Größe nicht passieren können. In der Flüssigkeit liegen sie nun aufgereinigt vor und können weiter analysiert werden.