Filoviridae
| Filoviridae | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 Elektronenmikroskopische | ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| Filoviridae | ||||||||||||||||
| Links | ||||||||||||||||
|
Filoviridae oder Filoviren sind eine Virusfamilie, zu der meist fadenförmige (lateinisch filum ‚Faden‘), behüllte Einzel(−)-Strang-RNA-Viren [ss(−)RNA] gehören. Sie zählen zu den größten bekannten RNA-Viren. Innerhalb dieser Familie verursachen die Gattungen Marburgvirus und Ebolavirus beim Menschen akute Erkrankungen mit hohem Fieber und einer hohen Letalität, die als hämorrhagisches Fieber zusammengefasst werden.
Die Filoviridae wurden erst 1982 als Gruppe definiert und die Virusfamilie hat seither und mit zunehmendem Kenntnisstand regelmäßige Veränderungen und Erweiterungen erfahren. Zu Zeiten der Ebolafieber-Epidemie 2014 bis 2016 gab es nur die beiden Gattungen Ebolavirus und Marburgvirus, mit der 2019 vom Executive Committee des für die Normierung zuständigen International Committee on Taxonomy of Viruses (ICTV) vorgeschlagenen Anpassung werden 2020 sechs Gattungen gezählt. Von diesen sind aber nur die beiden vorbenannten als Humanpathogen eingestuft, allerdings liegen für die meisten anderen identifizierten Filoviridae noch keine Isolate vor und ihr zoonotisches und humanpathogenes Potenzial sind ungeklärt.
Merkmale

Zu den Filoviridae gehören meist fadenförmige, manchmal auch bazillusförmige, behüllte Einzel(−)-Strang-RNA-Viren [ss(−)RNA]. In ihrer Grundstruktur können diese Viren manchmal u-förmig gebogen sein. In ihrer Länge variieren sie von 1.000 bis zu maximal 14.000 nm (Nanometer), ihr Durchmesser beträgt 60–80 nm und sie gehören damit zu den größten bekannten RNA-Viren.[3] Als weitere Besonderheit besitzen diese Viren die beiden Matrixproteine VP40 und VP24.[4]
Die Filoviren schädigen eine infizierte Zelle nicht unmittelbar durch Lyse, da sie sich aus der Zelle abschnüren und diese somit intakt bleibt. Ihr genauer Replikationsmechanismus ist jedoch noch unbekannt.
Vorkommen


Innerhalb dieser Familie sind die Gattungen Marburgvirus und Ebolavirus als humanpathogen beschrieben, wobei die Gattung Ebolavirus in sechs verschiedene Spezies unterteilt ist.[5] Sowohl das Marburgvirus als auch die meisten Vertreter der Gattung Ebolavirus verursachen beim Menschen eine akute Infektionskrankheit mit fulminantem Krankheitsverlauf.
Das Marburgvirus stammt primär aus Afrika und kommt in den Ländern Uganda, Kenia (West-Kenia) und vermutlich Simbabwe vor. Eine weitere Ausdehnung wird von Wissenschaftlern für wahrscheinlich gehalten. In Europa wurden 1967 die ersten Erkrankungsfälle dokumentiert, dies war die erste Beschreibung eines Filovirus, siehe dazu Marburgfieber.
Bei der Gattung Ebolavirus werden sechs Spezies voneinander unterschieden und jeweils nach den Orten ihres ersten Auftretens benannt. Mit Ausnahme der Spezies Reston Ebolavirus stammen alle anderen Virusspezies dieser Gruppe aus Afrika und verursachen beim Menschen hämorrhagisches Fieber mit sehr hoher Letalitätsrate und leichter Übertragbarkeit, siehe hierzu Ebolafieber. Die Spezies Reston Ebolavirus stammt ursprünglich von den Philippinen und verursacht bei Affen eine tödlich verlaufende Erkrankung, bei Menschen erfolgt jedoch nur eine subklinische Infektion ohne Krankheitssymptome.[4]
Die Gattung Cuevavirus mit der Spezies Lloviu Cuevavirus ist 2011 beschrieben worden. Sie wurde aus toten Langflügelfledermäusen (Miniopterus schreibersii) in Spanien und Ungarn isoliert.[6] Das Virus unterscheidet sich genetisch vom Marburg- und Ebolavirus und ist der erste Vertreter der Filoviridae, der in Europa entdeckt wurde.[6]
Die Suche nach den natürlichen Reservoirwirten für diese Viren dauert an. Bei mehreren Arten von Flughunden und Fledermäusen wurden Vertreter der Gattungen Ebolavirus oder Marburgvirus gefunden, ohne dass die Tiere erkrankt waren.[6]
Es wird diskutiert, dass Filoviren die Cyprianische Pest 250–270 n. Chr. ausgelöst haben könnte.[7]
Systematik
Die Virus-Taxonomie wird durch das International Committee on Taxonomy of Viruses (ICTV) geregelt. Die Aufgabe des ICTV liegt in der Klassifizierung der Viren in Taxa, ohne dabei die Nomenklatur der Vertreter dieser Taxa zu bestimmen. Dies wird von internationalen Expertengruppen übernommen, deren Vorschläge vom ICTV übernommen werden können. Die für die Filoviren zuständige Gruppe, die ICTV Filoviridae Study Group, hat 2010 eine aktualisierte Systematik und Nomenklatur der Vertreter der Filoviridae vorgeschlagen,[8] die in dem 9th ICTV Report umgesetzt wurden.[9] Allerdings hat sich die neue Nomenklatur noch nicht in allen Bereichen durchgesetzt, z. B. bei der Einstufung in Risikogruppen[10] nach der Biostoffverordnung oder in Meldungen über die Ebolafieber-Epidemie 2014. In diesem Zusammenhang verwendet die Weltgesundheitsorganisation (WHO) jedoch bereits die neue Nomenklatur.[11] Aus diesem Grund ist es sinnvoll, beide Systeme vorzustellen.
Aktuelle Systematik

Taxonomie mit Stand 30. April 2024 (Master Species List #39v1),[13][14][9]
Familie Filoviridae
- Gattung Cuevavirus
- Spezies Cuevavirus lloviuense (früher Lloviu cuevavirus)
- Virus: Lloviu Virus (LLOV) wurde in Fledermäusen (Miniopterus schreibersii) in Spanien und Ungarn sequenziert
- Spezies Cuevavirus lloviuense (früher Lloviu cuevavirus)
- Gattung Dianlovirus
- Spezies Dianlovirus menglaense (früher Mengla dianlovirus)
- Virus: Měnglà virus (MLAV) wurde in chinesischen Rosettenflughunden identifiziert
- Spezies Dianlovirus menglaense (früher Mengla dianlovirus)
- Gattung Loebevirus
- Spezies Loebevirus percae
- Virus: Lötschberg virus (LTBV) gefunden am Lötschberg
- Spezies Loebevirus percae
- Gattung Oblavirus
- Spezies Oblavirus percae
- Virus: Oberland virus (OBLV) gefunden im Flussbarsch im Berner Oberland
- Spezies Oblavirus percae
- Gattung Orthoebolavirus (früher Ebolavirus)[9][15] – Details siehe dort.
- Gattung Orthomarburgvirus (früher Marburgvirus) – 1967 das erste entdeckte Filovirus, Details siehe dort.
- Gattung Striavirus
- Spezies Striavirus antennarii (früher Xilang striavirus)
- Virus: Xīlǎng virus (XILV), gefunden bei Fischen
- Spezies Striavirus antennarii (früher Xilang striavirus)
- Gattung Tapjovirus
- Spezies Tapjovirus bothropis
- Virus: Tapajós virus (TAPV), gefunden im Tapajós National Forest, Brasilien, in Amerikanischen Lanzenottern
- Spezies Tapjovirus bothropis
- Gattung Thamnovirus
- Spezies Thamnovirus kanderense
- Virus: Kander virus (KNDV), gefunden im Schweizer Fluss Kander
- Spezies Thamnovirus percae
- Virus: Fiwi virus (FIWIV), gefunden im Flussbarsch
- Spezies Thamnovirus thamnaconi (früher Huangjiao thamnovirus)
- Virus: Huángjiāo virus (HUJV), bei Fischen
- Spezies Thamnovirus kanderense
Diese Systematik wird in ähnlicher Form (bis zur Spezies-Ebene) vom National Center for Biotechnology Information (NCBI)[5] und im Rahmen der Ebolafieber-Epidemie 2014 von der WHO[11] verwendet. Für die Nomenklatur der Taxa unterhalb der Spezies-Ebene wird von der zuständigen Expertengruppe geraten, auf die Bezeichnung als „Stamm“ oder „Virusstamm“ zu verzichten. Sie werden entweder als „natürliche Filovirus-Variante“ (englisch natural filovirus variant) oder „natürliches Filovirus-Isolat“ (engl. natural filovirus isolate) bezeichnet.[9]
Alte Systematik
Diese historische Taxonomie und Nomenklatur folgt dem 8th ICTV Report,[9][16] wie er zu Zeiten der Ebolafieber-Epidemie 2014 bis 2016 gültig war:
Familie Filoviridae
- Gattung Marburgvirus
- Spezies Lake Victoria Marburgvirus
- Virus: Lake Victoria Marburgvirus (MARV)
- Spezies Lake Victoria Marburgvirus
- Gattung Ebolavirus
- Spezies Côte d’Ivoire Ebolavirus [sic]
- Virus: Côte d’Ivoire Ebolavirus [sic] (CIEBOV)
- Spezies Reston Ebolavirus
- Virus: Reston Ebolavirus (REBOV)
- Spezies Sudan Ebolavirus
- Virus: Sudan Ebolavirus (SEBOV)
- Spezies Zaire Ebolavirus
- Virus: Zaire Ebolavirus (ZEBOV)
- Spezies Côte d’Ivoire Ebolavirus [sic]
Weblinks
Einzelnachweise
- ↑ a b ICTV: ICTV Taxonomy history: Akabane orthobunyavirus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ ICTV Master Species List 2018b v1 ( des vom 30. März 2019 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. MSL #34, Feb. 2019
- ↑ Herbert Hof, Rüdiger Dörries: Duale Reihe: Medizinische Mikrobiologie. 3. Auflage. Thieme Verlag, Stuttgart 2005, ISBN 978-3-13-125313-2, S. 207–208.
- ↑ a b J. Maruyama, H. Miyamoto u. a.: Characterization of the envelope glycoprotein of a novel filovirus, lloviu virus. In: Journal of Virology. Band 88, Nr. 1, Januar 2014, S. 99–109, ISSN 1098-5514. doi:10.1128/JVI.02265-13. PMID 24131711. PMC 3911722 (freier Volltext).
- ↑ a b Taxonomy Browser Filoviridae. In: Website National Center for Biotechnology Information (NCBI). Abgerufen am 13. August 2014.
- ↑ a b c A. Negredo, G. Palacios u. a.: Discovery of an ebolavirus-like filovirus in europe. In: PLoS pathogens. Band 7, Nr. 10, Oktober 2011, S. e1002304, ISSN 1553-7374. doi:10.1371/journal.ppat.1002304. PMID 22039362. PMC 3197594 (freier Volltext).
- ↑ Kyle Harper: Fatum. Das Klima und der Untergang des Römischen Reiches. Verlag C.H. Beck, München 2020, ISBN 978-3-406-74933-9, S. 214 ff.
- ↑ J. H. Kuhn, S. Becker u. a.: Proposal for a revised taxonomy of the family Filoviridae: classification, names of taxa and viruses, and virus abbreviations. In: Archives of Virology. Band 155, Nr. 12, Dezember 2010, S. 2083–2103, ISSN 1432-8798. doi:10.1007/s00705-010-0814-x. PMID 21046175. PMC 3074192 (freier Volltext).
- ↑ a b c d e J. H. Kuhn, Y. Bao u. a.: Virus nomenclature below the species level: a standardized nomenclature for laboratory animal-adapted strains and variants of viruses assigned to the family Filoviridae. In: Archives of Virology. Band 158, Nr. 6, Juni 2013, S. 1425–1432, ISSN 1432-8798. doi:10.1007/s00705-012-1594-2. PMID 23358612. PMC 3669655 (freier Volltext).
- ↑ TRBA (Technische Regeln für Biologische Arbeitsstoffe) 462: Einstufung von Viren in Risikogruppen. In: Website der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. April 2012, S. 4, 27, abgerufen am 9. August 2014.
- ↑ a b Ebola virus disease – WHO Fact Sheet No. 103. In: Website der WHO. April 2014, abgerufen am 12. August 2014.
- ↑ Jonathan S. Towner, Tara K. Sealy et al.: Newly Discovered Ebola Virus Associated with Hemorrhagic Fever Outbreak in Uganda. In: PLoS Pathogens., Bd. 4, Nr. 11, November 2008, S. e1000212, ISSN 1553-7374; doi:10.1371/journal.ppat.1000212 (englisch).
- ↑ ICTV: Taxonomy Browser.
- ↑ ICTV: Virus Metadata Resource (VMR).
- ↑ Man vergleiche in der englischsprachigen Wikipedia die Artikel en:Ebola virus und en:Ebolavirus
- ↑ H. Feldmann, T. W. Geisbert u. a.: Family Filoviridae. In: C. M. Fauquet, M. A. Mayo, J. Maniloff, U. Desselberger, L. A. Ball (Hrsg.): Virus Taxonomy – Eighth Report of the International Committee on Taxonomy of Viruses. Elsevier/Academic Press, San Diego (CA), USA 2005, S. 645–653 (online).
Auf dieser Seite verwendete Medien
3D model of a Filovirus
Autor/Urheber: Madiiha Bibi Mandary, Malihe Masomian, and Chit Laa Pooh, Lizenz: CC BY 4.0
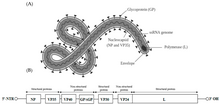
(A) Schematic representation of mature Ebola virion consisting of two main components—the nucleocapsid and envelope. The matrix comprising the virion proteins VP24 and VP40 is located between the nucleocapsid and envelope. Glycoprotein (GP) spikes are located on the surface of the envelope (B) The genome contains 7 genes which encode the six structural proteins and one non-structural protein [79]. The gene order is 5′-NTR-NP (nucleoprotein)-VP35-VP40 (Major matrix protein)-GP/sGP (Glycoprotein)-VP30-VP24 (Minor matrix protein)-RNA-dependent RNA polymerase (l)-3′-NTR [80].
Autor/Urheber:
Source authors: Jens H. Kuhn, Gaya K. Amarasinghe, Christopher F. Basler, Sina Bavari, Alexander Bukreyev, Kartik Chandran, Ian Crozier, Olga Dolnik, John M. Dye, Pierre B. H. Formenty, Anthony Griffiths, Roger Hewson, Gary P. Kobinger, Eric M. Leroy, Elke Mühlberger, Sergey V. Netesov (Нетёсов Сергей Викторович), Gustavo Palacios, Bernadett Pályi, Janusz T. Pawęska, Sophie J. Smither, Ayato Takada (高田礼人), Jonathan S. Towner and Victoria Wahl
Image courtesy of:
Jiro Wada, IRF-Frederick, Fort Detrick, MD, USA. (US government), Lizenz: CC BY 4.0Replication cycle of filoviruses (possibly excluding striaviruses and thamnoviruses). Virions attach to cell-surface attachment factors (orange Ys) and are taken into the cell via endocytosis. The filovirion glycoproteins (yellow clubs) bind to endosomal Niemann-Pick disease, type C1 (NPC1, white zigzag) catalyze the fusion of viral and cellular membranes to release the filovirus RNP complex (green helix). The polymerase complex (consisting of VP35 [purple dots] and L [blue semicircles]) transcribes filovirus mRNAs that are translated into filovirus proteins and replicates filovirus genomic RNA via antigenomic intermediates. Genomic RNA and antigenomic RNA occur only as ribonucleoprotein complexes which serve as templates for replication and/or transcription. Assembly of filoviral proteins and progeny genomes occurs in the cytoplasm and results in budding of progeny virions at the plasma membrane.
Marburg-Virus.
Autor/Urheber: Illustration: Jennifer Matthews https://www.eurekalert.org/pub_releases/2021-01/uoc--npw010521.php, Lizenz: CC BY-SA 4.0
Replikationszyklus von Viren der Familie Filoviridae
Phylogenetic tree comparing full-length genomes of ebolavirus and marburgvirus by Bayesian analysis. Posterior probabilities greater than 0.5 and maximum likelihood bootstrap values greater than 50 are indicated at the nodes.





