Fiersviridae
| Fiersviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
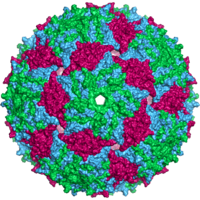 Kapsid des Bakteriophagen MS2 | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Fiersviridae | ||||||||||||||
| Links | ||||||||||||||
|
Die Fiersviridae (veraltet: Leviviridae) sind eine Familie von Viren, die als Bakteriophagen verschiedene Bakterien, einschließlich Enterobakterien, Caulobacter, Pseudomonas und Acinetobacter befallen. Die Taxonomie des International Committee on Taxonomy of Viruses (ICTV) mit Stand November 2018 kennt zwei Gattungen zu je zwei Spezis.[4][5]
Es handelt sich um kleine RNA-Viren mit linearem, einzelsträngigem RNA-Genom positiver Polarität, die nur vier Proteine kodieren. Alle Phagen dieser Familie benötigen bakterielle Pili, um sich an die Wirtszelle (Bakterienzelle) zu binden und diese zu infizieren.[6]
Im März 2021 wurde vorgeschlagen, diese Viren aus der bisherigen Klasse Allassoviricetes in eine neue Klasse Leviviricetes zu verschieben.[7] Dem hat das ICTV in der Ausgabe 36 seiner Master Species List (MSL) 2021 entsprochen, und dabei gleichzeitig die Ordnung (von Levivirales zu Norzivirales) und die Familie (von Leviviridae zu Fersviridae) umbenannt. Gleichzeitig hat sich die Anzahl der Gattungen von zwei auf 185 erhöht.[1]
Systematik
Äußere Systematik
Die Familie ist Mitglied der Ordnung Norzivirales (früher Levivirales). Die meisten Vertreter dieser Ordnung wurden per Metagenomik gefunden.[8] Zu dieser gehören die folgenden Familien:[1]
- Familie Atkinsviridae
- Familie Duinviridae
- Familie Fiersviridae (veraltet Leviviridae)
- Familie Solspiviridae
Innere Systematik
Die Familie hat mit ICTV Stand 7. Mai 2024 dreihundertacht (308) Gattungen (Genera). Diese sind – mit einer Auswahl an Arten (Spezies):[9][10]
- Familie Fiersviridae (veraltet Leviviridae)
- Genus Amubhivirus
- Genus Andhasavirus
- Genus Andhaxevirus
- Genus Ashucavirus
- Genus Bahscuvirus
- Genus Behevivirus
- Genus Behlfluvirus
- Genus Bihdovirus
- Genus Bisdanovirus
- Genus Blafavirus
- Genus Bohnovirus
- Genus Bohwovirus
- Genus Boschuvirus
- Genus Breudwovirus
- Genus Buhdavirus
- Genus Cahdavirus
- Genus Cahrpivirus
- Genus Caloevirus
- Genus Cauhldivirus
- Genus Cehakivirus
- Genus Chaedoavirus
- Genus Chahsmivirus
- Genus Chihyovirus
- Genus Chobevirus
- Genus Choctavirus
- Genus Cintrevirus
- Genus Cunavirus
- Genus Dahmuvirus
- Genus Darnbovirus
- Genus Decadevirus
- Genus Denfovirus
- Genus Depandovirus
- Genus Dihsdivirus
- Genus Dohlivirus
- Genus Dortuvirus mit ehem. Creshivirus fonticola
- Genus Dorudgevirus
- Genus Dosmizivirus
- Genus Draycavirus
- Genus Dreytovirus
- Genus Duhcivirus
- Genus Dunchevirus
- Genus Dursuvirus inkl. ehem. Genus Apukhovirus
- Genus Eahsevirus
- Genus Eahvevirus
- Genus Eedhovirus
- Genus Eerlswovirus
- Genus Eistcuvirus
- Genus Emesvirus (früher Levivirus)[11]
- Spezies Emesvirus japonicum (veraltet: Escherichia virus BZ13)
- Spezies Emesvirus zinderi (mit Escherichia virus MS2 alias Enterobakteriophage MS2, Coliphage MS2)
- Spezies Emesvirus piscicola
- Genus Empivirus
- Genus Etongtovirus
- Genus Fagihyuvirus
- Genus Fenycovirus
- Genus Fihluvirus
- Genus Firnpuvirus
- Genus Fiyodovirus
- Genus Flehknovirus
- Genus Frinctavirus
- Genus Fulbrouvirus
- Genus Gahlovirus
- Genus Galoycavirus
- Genus Garovuvirus
- Genus Gehnevirus
- Genus Giydovirus
- Genus Glyciruvirus
- Genus Gmuhndevirus
- Genus Gorodievirus
- Genus Grandpovirus
- Genus Greatevirus
- Genus Greatpavirus
- Genus Grehavirus
- Genus Grendovirus
- Genus Grendvuvirus
- Genus Gunawavirus
- Genus Haeldovirus
- Genus Hagavirus
- Genus Hahdsevirus
- Genus Hahmbtovirus
- Genus Hahsevirus
- Genus Halcalevirus
- Genus Hampdavirus
- Genus Hamptovirus
- Genus Helfovirus
- Genus Herbuvirus
- Genus Hihdivirus
- Genus Hihlwaovirus
- Genus Hirbarovirus
- Genus Hirtshivirus
- Genus Hisilivirus
- Genus Hisilovirus
- Genus Hitdovirus
- Genus Hohnivirus
- Genus Honleyivirus
- Genus Honolovirus
- Genus Hudnivirus
- Genus Huhnivirus
- Genus Huhnuvirus
- Genus Hukohnovirus
- Genus Hurlivirus
- Genus Icumivirus
- Genus Ideskevirus
- Genus Idlucavirus
- Genus Imeberivirus
- Genus Ineyimevirus
- Genus Iruqauvirus
- Genus Ishugivirus
- Genus Jiesduavirus
- Genus Johnovirus
- Genus Jupbevirus
- Genus Kahfsdivirus
- Genus Keghovirus
- Genus Kehmevirus (inkl. ehem. Genus Aldhiuvirus)
- Genus Kehrevirus
- Genus Kemicevirus
- Genus Kihryuvirus
- Genus Kinehtovirus
- Genus Kinolwovirus
- Genus Kirnavirus
- Genus Kiwsmaevirus
- Genus Kongskuvirus
- Genus Konkivirus
- Genus Konwavirus
- Genus Koteshevirus inkl. ehem. Brudgevirus terrivivens (jetzt Koteshevirus terrivivens), ehem. Condavirus edaphovicinum, ehem. Condavirus edaphohabitans
- Genus Kowinovirus
- Genus Kuhshuvirus
- Genus Kungsnevirus
- Genus Kungspuvirus
- Genus Lahbwuvirus
- Genus Lahdbrovirus
- Genus Langlevirus
- Genus Leahmovirus
- Genus Lecwavirus
- Genus Lehamuvirus
- Genus Lighthovirus
- Genus Lihdtlovirus
- Genus Lihtdlivirus mit ehem. Condavirus edaphenecus
- Genus Litdlevirus
- Genus Loamavirus
- Genus Loghdhuvirus
- Genus Lohmavirus
- Genus Lohngkovirus inkl. Teile von ehem. Genus Brudgevirus
- Genus Lohwevirus
- Genus Lohwovirus
- Genus Longmavirus
- Genus Longovirus
- Genus Loslovirus
- Genus Lowerquvirus
- Genus Lowsuvirus
- Genus Loxlovirus
- Genus Ludtlevirus
- Genus Ludtlovirus
- Genus Ludtovirus
- Genus Lunjlavirus
- Genus Luthavirus inkl. ehem. Genus Bathrivirus
- Genus Lyejrivirus
- Genus Mahqeavirus
- Genus Mahraivirus
- Genus Manrohovirus
- Genus Mahrtovirus
- Genus Manrohovirus
- Genus Martavirus
- Genus Martovirus
- Genus Maxstovirus
- Genus Meblowovirus
- Genus Mehraxmevirus
- Genus Mehrevirus
- Genus Mekintivirus
- Genus Merlkluvirus
- Genus Methovirus
- Genus Mihkrovirus inkl. ehem. Genus Bertavirus
- Genus Mincetevirus
- Genus Mintuvirus
- Genus Mohredovirus
- Genus Molcatevirus
- Genus Molvevirus
- Genus Monkskivirus
- Genus Moxhuvirus
- Genus Mutledovirus
- Genus Muyegivirus
- Genus Nadsecevirus
- Genus Napdanuvirus
- Genus Nedhivirus
- Genus Nehwtovirus
- Genus Newbovirus
- Genus Newpuvirus
- Genus Niankuvirus
- Genus Niuhvovirus
- Genus Niwnhavirus
- Genus Niwtovirus
- Genus Nohmivirus
- Genus Nohrdovirus
- Genus Northevirus
- Genus Oceshuvirus
- Genus Oldperovirus
- Genus Olmsdivirus
- Genus Omohevirus
- Genus Onohmuvirus
- Genus Opdykovirus
- Genus Overwhuvirus
- Genus Owenocuvirus
- Genus Oxychlovirus
- Genus Pailtovirus
- Genus Paloswovirus
- Genus Palsdevirus
- Genus Paysduvirus
- Genus Pedhlovirus
- Genus Pehohrivirus
- Genus Pehsaduvirus
- Genus Pepevirus
- Spezies Pepevirus rubrum (mit Pseudomonas phage PP7)[12]
- …
- Genus Perrunavirus
- Genus Philtcovirus
- Genus Phobpsivirus
- Genus Piceduvirus
- Genus Pihlevirus
- Genus Pilordavirus
- Genus Piponevirus
- Genus Pohlevirus
- Genus Poncivirus
- Genus Prihavirus
- Genus Prihovirus
- Genus Prisdovirus
- Genus Prosdovirus
- Genus Pruncivirus
- Genus Psimevirus
- Genus Pudlivirus
- Genus Punlevirus
- Genus Qubevirus (früher Allolevivirus)[13]
- Spezies Qubevirus durum (mit Escherichia virus Qbeta alias Enterobakteriophage Qbeta, Coliphage Qβ)
- Spezies Qubevirus escherichienecus (mit Escherichia virus Qbeta 1)
- Spezies Qubevirus faecium (mit Escherichia virus FI)
- Genus Quihndavirus
- Genus Radbaivirus
- Genus Rahdfavirus
- Genus Ratlevirus
- Genus Redbauvirus
- Genus Rehihmevirus
- Genus Rehudzovirus
- Genus Ridwavirus
- Genus Rusvolovirus
- Genus Rytunovirus
- Genus Sahwbrivirus
- Genus Sambuevirus
- Genus Saudhavirus
- Genus Sbehrnivirus
- Genus Scuadavirus
- Genus Sdadlevirus
- Genus Sdoctuvirus
- Genus Sdohnivirus
- Genus Sdredfovirus
- Genus Sdredtuvirus
- Genus Sdretovirus
- Genus Seckivirus
- Genus Sehpovirus
- Genus Selfuvirus
- Genus Seybrovirus
- Genus Shebanavirus
- Genus Shehrpovirus
- Genus Shestuvirus
- Genus Shihovirus
- Genus Shipsduvirus
- Genus Shodtevirus
- Genus Sholavirus
- Genus Shomudavirus
- Genus Shrewlivirus
- Genus Shuldovirus
- Genus Shutevirus
- Genus Shutovirus
- Genus Sincthavirus
- Genus Skhembuvirus
- Genus Smudhfivirus
- Genus Snitdevirus
- Genus Soetuvirus
- Genus Stanolovirus
- Genus Stehlmavirus
- Genus Sterodovirus
- Genus Stoartovirus
- Genus Stretavirus
- Genus Stritovirus
- Genus Suhtovirus
- Genus Swihdzovirus
- Genus Tahluvirus
- Genus Tapikevirus
- Genus Teciucevirus
- Genus Tehnexuvirus
- Genus Temblevirus
- Genus Templevirus
- Genus Templovirus
- Genus Tenwovirus mit ehem. Creshivirus pelohabitans
- Genus Thiwvovirus
- Genus Thiyevirus
- Genus Thobivirus
- Genus Thurlavirus
- Genus Ticahravirus
- Genus Todtivirus
- Genus Tohvovirus
- Genus Trotivirus
- Genus Trucevirus
- Genus Tudmivirus
- Genus Tysohivirus
- Genus Ulinhavirus
- Genus Upborqevirus (inkl. ehem. Genus Boloprevirus)
- Genus Ureyisuvirus
- Genus Ushumevirus
- Genus Vohsuavirus
- Genus Whietlevirus
- Genus Whilavirus
- Genus Wohudhevirus
- Genus Wyahnevirus
- Genus Yahnavirus (inkl. ehem. Genus Adahivirus)
- Genus Yemegivirus
- Genus Yohcadevirus
- Genus Yuhrihovirus
Bei Reorganisationen wurden beispielsweise verschoben:
- Genus Condavirus aufgeteilt Koteshevirus + Lihtdlivirus
- Genus Creshivirus aufgeteilt Dortuvirus + Tenwovirus
Aufbau

(a) Enterobacteriaphage MS2 (Gattung Levivirus)
(b) Enterobacteriaphage Qß (Gattung Allolevivirus)
Die Viruspartikel (Virionen) der Leviviridae sind nicht umhüllt mit ikosaedrischer oder sphärischer Geometri und mit Triangulationszahl (Symmetrie) T=3. Der Durchmesser beträgt etwa 26 nm. Das Genom ist linear und nicht segmentiert mit einer Größe von etwa 4000 Nukleotidbasen. Es kodiert für 4 Proteine.[4]
Wirkungsweise

Nach Eintritt die Wirtszelle erfolgt die Replikation dem Modell für RNA-Viren positiver Polarität. Das Virus tritt durch Bakterienlyse aus der Wirtszelle aus.[4]
Die vier kodierten Proteine sind das Mantel- oder Hüllprotein (für das Kapsid), die Replikase, das Reifungsprotein (A-Protein) und das Lyse-Protein, die zugehörigen Gene heißen cp, rep, mat und lys.[6]
Einzelnachweise
- ↑ a b c d ICTV:ICTV Master Species List 2020.v1 ( des vom 24. September 2021 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis., New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- ↑ a b ICTV:ICTV Master Species List 2019.v1 ( des vom 2. August 2022 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis., New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ↑ ICTV Master Species List 2018b v1 ( des vom 30. März 2019 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. MSL #34, Feb. 2019
- ↑ a b c ViralZone – Fiersviridae. ExPASy, abgerufen am 19. Dezember 2018 (englisch).
- ↑ ICTV: ICTV Master Species List 2018a v1. Archiviert vom am 14. März 2019; abgerufen am 19. Dezember 2018 (englisch). Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis.
- ↑ a b JP Bollback, JP Huelsenbeck: Phylogeny, genome evolution, and host specificity of single-stranded RNA bacteriophage (family Leviviridae). In: Journal of Molecular Evolution. 52. Jahrgang, Nr. 2, Februar 2001, S. 117–28, doi:10.1007/s002390010140, PMID 11231891 (englisch).
- ↑ Dann Turner, Andrew M. Kropinski, Evelien M. Adriaenssens: A Roadmap for Genome-Based Phage Taxonomy, in: MDPI Viruses Band 13, Nr. 3, Section Bacterial Viruses, 18. März 2021, 506, doi:10.3390/v13030506
- ↑ J. Callanan, S. R. Stockdale, E. M. Adriaenssens, J. H. Kuhn: Rename one class (Leviviricetes - formerly Allassoviricetes), rename one order (Norzivirales - formerly Levivirales), create one new order (Timlovirales), and expand the class to a total of six families, 420 genera and 883 species. In: Researchgate. Januar 2021, doi:10.13140/RG.2.2.25363.40481 (englisch, researchgate.net).
- ↑ ICTV: Taxonomy Browser.
- ↑ ICTV: Virus Metadata Resource (VMR).
- ↑ SIB: Levivirus, auf: ViralZone
- ↑ Jirapat Thongchol, Zihao Yu, Laith Harb, Yiruo Lin, Matthias Koch, Matthew Theodore, Utkarsh Narsaria, Joshua Shaevitz, Zemer Gitai, Yinghao Wu, Junjie Zhang, Lanying Zeng: Removal of Pseudomonas type IV pili by a small RNA virus. In: Science, Band 384, Nr. 6691, 5. April 2024; doi:10.1126/science.adl0635 (englisch). Dazu:
- Researchers resolve old mystery of how phages disarm pathogenic bacteria. New study details long-sought mechanisms and structures. Auf: EurekAlert! vom 15. April 2024.
- Cutting-Edge Phage Research Promises New Solutions for Old Pathogens. Auf: ciTechDaily vom 28. Mai 2024.
- ↑ SIB: Allolevivirus, auf: ViralZone
Weblinks
Auf dieser Seite verwendete Medien
Autor/Urheber: Óttar Rolfsson, Stefani Middleton, Iain W. Manfield, Simon J. White, Baochang Fan, Robert Vaughan, Neil A. Ranson, Eric Dykeman, Reidun Twarock, James Ford, C. Cheng Kao, Peter G. Stockley, Lizenz: CC BY 4.0
Schematic of the bacteriophage MS2 lifecycle. Virions initially bind to the sides of the bacterial pilus via the MP. MPs are an essential single-copy structural component of RNA phages. By a mechanism that remains largely obscure, the RNA–MP complex but not the remaining capsid shell enters the cell. MP also binds to its own PSs located toward either end of the MS2 genome. Recent asymmetric reconstruction of the MS2–pilus complex suggests that MP replaces a CP dimer of the C/C conformer in an otherwise entirely icosahedral protein shell, from which position it is ideally placed to contact the cellular receptor and escort the RNA into the cytoplasm. Note that the presence of the asymmetric MP component could not be detected in averaged X-ray and EM density maps. Once internalized, the MP is cleaved into two separate fragments by protease, allowing translation and replication to start. Temporal control of phage gene expression then regulates the production of progeny genomes and structural proteins that assemble prior to the action of the phage lysis protein.
Autor/Urheber: Julie Callanan, Stephen R. Stockdale, Andrey Shkoporov,Lorraine A. Draper, Paul Ross, Colin Hill, Lizenz: CC BY 4.0
Schemazeichnung eines Virusteilchens der Fiersviridae, früher Leviviridae (Querschnitt) und Genom von
(a) Enterobacteriaphage MS2 (Gattung Emesvirus, früher Levivirus)
(b) Enterobacteriaphage Qß (Gattung Emesvirus, früher Allolevivirus)Autor/Urheber: Naranson, Lizenz: CC BY-SA 3.0
Cartoon representation of the entire bacteriophage MS2 protein capsid (pdb id 1AQ3). The three quasi-equivalent conformers in the structure are labelled blue (chain a), green (chain b) and magenta (chain c). The view is approximately down an icosahedral 5-fold symmetry axis


