Enterobakterien
| Enterobakterien | ||||||||
|---|---|---|---|---|---|---|---|---|
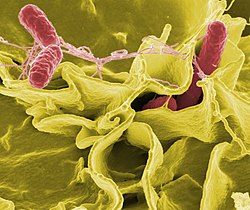
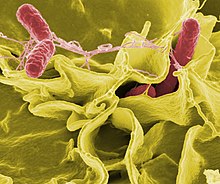
 Citrobacter freundii (sekundärelektronenmikroskopisches Bild) | ||||||||
| Systematik | ||||||||
| ||||||||
| Wissenschaftlicher Name | ||||||||
| Enterobacterales | ||||||||
| Adeolu et al. 2016 |
Die Enterobakterien bzw. die Vertreter der 2016 etablierten Ordnung Enterobacterales sind eine große Gruppe gramnegativer Bakterien. Nach dem phylogenetischen System gehören sie zur Klasse der Gammaproteobacteria in der Abteilung (Divisio, bei den Prokaryoten auch als Phylum bezeichnet) PseudomonadotaGarrity et al. 2021 (ehemals Proteobacteria) und bilden dort eine eigene Ordnung.
Vor 2016 wurde für diese Bakteriengruppe der Name „Enterobacteriales“ verwendet, der jedoch nach den Regeln des Bakteriologischen Codes ICNB (die vor 2019 gültige Version des International Code of Nomenclature of Prokaryotes, ICNP) nicht gültig war. Weiterhin gab es nur eine Familie – die Enterobacteriaceae – innerhalb der Ordnung. Mit der Etablierung der Ordnung Enterobacterales verbunden ist eine Aufteilung der bisher bekannten Taxa auf mehrere neue Familien.[1] Die neue Systematik führt beispielsweise dazu, dass die Gattungen Escherichia, Morganella und Yersinia drei unterschiedlichen Familien angehören (vgl. Abschnitt Systematik).
Der Name Enterobakterien leitet sich von enteron (gr. ἔντερον ‚Darm‘) ab, weil viele von ihnen typische Darmbewohner sind. Aber auch viele freilebende und ubiquitär vorkommende, nicht darmbewohnende Bakterienarten gehören in diese Ordnung. Unter den Enterobacterales finden sich bedeutende Krankheitserreger, ebenso gibt es zahlreiche Vertreter, die als nicht pathogen angesehen werden. Sie können mit oder ohne Sauerstoff wachsen, ihr Stoffwechsel ist fakultativ anaerob.
Merkmale
Erscheinungsbild
Die Zellen sind stäbchenförmig und gewöhnlich 1 bis 5 µm (Mikrometer) lang und besitzen einen Durchmesser von etwa 0,5–1,0 µm. Es werden keine Endosporen gebildet. Die meisten können sich mit Flagellen aktiv bewegen, sie sind motil, es kommen jedoch auch Gattungen vor, die sich nicht aktiv bewegen können. Da die Bakterienzellwand aus wenigen Mureinschichten und einer zweiten, äußeren Membran besteht, sind die Enterobakterien gramnegativ, werden also in der Gram-Färbung durch die verwendeten Farbstoffe rosa bis rot angefärbt. Bei manchen Arten, z. B. aus der Gattung Klebsiella ist die Zellwand von einer Kapsel umgeben, die aus Polysacchariden besteht. Sie verleihen den auf einem Nährboden gewachsenen Bakterienkolonien ein schleimiges Aussehen.
Wachstum und Stoffwechsel
Ihr Stoffwechsel ist fakultativ anaerob, daher können sie sowohl über Oxidation unter Anwesenheit von Sauerstoff Stoffe abbauen, als auch unter anaeroben Bedingungen (kein Sauerstoff) Gärung betreiben. Als Kohlenstoff- und Energiequellen verwerten sie D-Glucose und weitere Kohlenhydrate. Zwei wichtige anaerobe Stoffwechselwege, die zur Unterscheidung der einzelnen Gattungen genutzt werden, sind die 2,3-Butandiol-Gärung und die gemischte Säuregärung (mixed acid fermentation). Bei der gemischten Säuregärung treten als End- und Nebenprodukte vorwiegend organische Säuren, wie Essigsäure, Milchsäure und Bernsteinsäure (Succinat), aber kein Butandiol auf. Bei der 2,3-Butandiol-Gärung entstehen aus der Gärung von Glucose als End- und Nebenprodukte geringere Mengen von Säuren, aber vor allem in großen Mengen der Alkohol 2,3-Butandiol. Ein weiteres Merkmal der 2,3-Butandiol-Gärung ist das Zwischenprodukt Acetoin und die wesentlich höhere Gasproduktion (CO2). Man findet Butandiolgärung z. B. bei Enterobacter, Klebsiella, Erwinia und Serratia. Gemischte Säuregärung nutzen u. a. Gattungen wie Escherichia, Salmonella und Proteus. Weiterhin reduzieren die Enterobakterien Nitrat zu Nitrit. Der Oxidase-Test verläuft bei ihnen negativ, der Katalase-Test bei vielen, aber nicht allen Arten positiv.
Zur Bestimmung der einzelnen Gattungen wird eine Vielzahl von Diagnosetests genutzt. Zum Beispiel wird mit Hilfe des Voges-Proskauer-Tests das Zwischenprodukt Acetoin der 2,3-Butandiol-Gärung nachgewiesen. Auch der Nachweis des Enzyms β-Galactosidase wird häufig zur Unterscheidung verwendet. Bakterien, die über dieses Enzym verfügen, können das Disaccharid Lactose (Milchzucker) hydrolytisch in die Monosaccharide Glucose und Galactose spalten, um sie im Stoffwechsel zu nutzen. Sie werden als lactosepositiv charakterisiert und auch als Coliforme bezeichnet. Bei Selektivmedien erfolgt der Nachweis des Lactoseabbaus häufig über die Säurebildung beim fermentativen Abbau des Kohlenhydrats, durch die Säurebildung verändert der im Nährmedium enthaltene pH-Indikator seine Farbe. Bei manchen Arten ergibt sich in Bezug auf die Lactoseverwertung kein einheitliches Bild, da zu wenig Säure produziert wird, wobei dann die Angabe „variabel“ verwendet wird, hier ist es empfehlenswert, den ONPG-Test zu nutzen. Einen Überblick über biochemische Nachweise oder weitere Methoden zur Identifizierung findet sich in den jeweiligen Gattungs- oder Spezies-Artikeln.

Die meisten Enterobakterien sind in Bezug auf die benötigten Nährstoffe nicht anspruchsvoll und wachsen auf üblichen Nährmedien, die Pepton oder Fleischextrakt enthalten, als nicht-selektives Nährmedium kann auch Blutagar genutzt werden. Sie tolerieren einen Massenanteil von 0–6 % an Natriumchlorid (NaCl) im Nährmedium, ein NaCl-Zusatz im Nährmedium ist aber nicht notwendig. Wenn sie auf MacConkey-Agar kultiviert werden, lassen sich bereits lactosepositive und lactosenegative Vertreter unterscheiden, auch Eosin-Methylen-Blau-Agar wird dafür eingesetzt. Sie wachsen auf oder in Nährmedien, die Gallensalze enthalten, dies kann zur Isolierung genutzt werden. Auf festen Nährmedien wachsen die Kolonien mit einem Durchmesser von 2–3 mm. Auffällig ist hier die Gattung Proteus, insbesondere P. vulgaris und P. mirabilis, bei denen man das sogenannte „Schwärm-Phänomen“ beobachtet. Wenn sich wachsende Kolonien dieser Bakterien auf einer Agar-Platte ausbreiten, sieht man einen Bakterienrasen mit konzentrischen Ringen.[2]
Die meisten Vertreter der Enterobacterales sind mesophil, d. h. sie bevorzugen mittlere Temperaturen, medizinisch bedeutsamen Arten wachsen meist gut bei 35 – 37 °C. Das bakterielle Wachstum findet allerdings in einem weiten Bereich von ungefähr 4 – 45 °C statt, je nachdem, welche Art kultiviert wird. Einige Vertreter zeigen bei niedrigeren Temperaturen (25 – 30 °C im Vergleich zu 37 °C) eine höhere Stoffwechselaktivität, was in den oben genannten biochemischen Tests dann beispielsweise zu einem positiven Ergebnis führt, während der Test bei einer Inkubation bei 37 °C negativ oder nicht eindeutig ausfällt. Viele Yersinia-Arten sind bei niedrigeren Temperaturen motil, aber nicht bei 37 °C. Ähnliche Ergebnisse findet man bei Arten aus den Gattungen Enterobacter, Hafnia und Tatumella.[2]
Chemotaxonomie
Wie für gramnegative Bakterien typisch, besitzen die Enterobakterien eine äußere Membran, bei ihr sind die Phospholipide überwiegend durch Polysaccharide ersetzt, die mit Lipiden komplexe Lipopolysaccharide (LPS) bilden. Diese Lipopolysaccharide wirken als Antigene, Pyrogene und Endotoxine. Die Enterobacterales besitzen ein gemeinsames Antigen, das ECA (enterobacterial common antigen), welches chemisch gesehen ein Polymer aus Aminozuckern ist, die teilweise mit Fettsäuren verestert sind. Das ECA liegt normalerweise als freies Glykolipid in der äußeren Membran vor, bei einigen Bakterienstämmen hingegen ist es mit den LPS verbunden.[2]

Folgende Bestandteile der Bakterienzelle wirken ebenfalls als Antigene: Es kommen einmal die sogenannten somatischen O-Antigene vor, sie beruhen auf den nach außen ragenden Oligosacchariden der Lipopolysaccharide in der äußeren Membran der Zellen, sie werden auch O-spezifische Oligosaccharide oder O-spezifische Kette genannt. Die H-Antigene sind durch die Flagellen begründet, die F-Antigene durch die Fimbrien und die K-Antigene durch eine Kapsel. Die O- und H-Antigene wurden ursprünglich bei Untersuchungen von Proteus-Stämmen beschrieben, sehr motile Stämme überziehen beim Wachstum das Nährmedium wie mit einem Film, ohne sichtbare Bildung von Kolonien, dies wurde als „Hauch“ bezeichnet, darauf basiert der verwendete Buchstabe ‚H‘. Stämme, die nicht motil sind, wachsen „ohne Hauch“ (‚O‘).[3] Die Bezeichnung und die zelluläre Zuordnung der Antigene ist bei dem für Salmonellen angewendeten Kauffmann-White-Schema beschrieben, es erlaubt die serologische Einordnung von Serovaren (Serotypen) zur Identifizierung.
Bei pathogenen Stämmen von Enterobakterien spielt das O-Antigen eine Rolle, bestimmte O-Antigene verursachen eine höhere Virulenz, ein Beispiel ist das Serovar E. coli O157:H7, ein Vertreter der Enterohämorrhagischen Escherichia coli (EHEC).[3] Das glatte (smooth) Aussehen der Kolonien wird mit den O-spezifischen Oligosacchariden in Verbindung gebracht, die ein Teil der LPS sind. Manche Mutanten oder bestimmte Stämme wachsen als raue (rough) Kolonien, bei ihnen sind die LPS zwar ebenfalls vorhanden, aber bei den enthaltenen Polysaccharidketten liegt nur der Kernteil (core) vor, die variable O-spezifische Kette fehlt.[2] Die Antigene sind auch die Grundlage für serologische Untersuchungen von Patientenseren, beispielsweise bei der Bestimmung des Antikörpertiters, dies wird jedoch nur selten durchgeführt.[3]
Pathogenität
Angesichts der vielen Arten, die zur Ordnung der Enterobacterales gehören, sind keine allgemeinen Aussagen möglich. Zur Ordnung gehören drei bedeutsame Krankheitserreger: Yersinia pestis, der Erreger der Lungen- und Beulenpest, Salmonella enterica subsp. enterica Serovar Typhi (kurz: Salmonella Typhi) als Typhus-Erreger und Shigella dysenteriae Serovar 1, verantwortlich für die Bakterienruhr. Sie werden durch die Biostoffverordnung in Verbindung mit der TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466 der Risikogruppe 3 zugeordnet, der höchsten für Bakterien vergebenen Risikogruppe.[4] Auch die weiteren Shigella- und Salmonella-Arten oder -Serovare verursachen beim Menschen Diarrhoe (Durchfall) und intestinale Infektionen, also Infektionen im Darm („Enteritis“), sie werden der Risikogruppe 2 zugeordnet.[4]
Ebenfalls sind einige, aber nicht alle Arten der Gattungen Escherichia und Yersinia typische Darm-Pathogene. Eine Auswahl weiterer pathogener Enterobakterien ist im Abschnitt Medizinische Bedeutung genannt. Weitere Vertreter können bei gesundheitlich geschwächten Personen krankheitserregend wirken, sie sind also fakultativ pathogene (opportunistische) Krankheitserreger. Sie kommen auch bei Gesunden häufig im Darm vor und lösen nicht notwendigerweise Krankheiten aus. Daneben gibt es zahlreiche Vertreter der Ordnung, die nicht pathogen für den Menschen sind und der Risikogruppe 1 zugeordnet werden. Manche Arten sind jedoch pathogen für Pflanzen, beispielsweise Erwinia und Pectobacterium oder für Tiere, beispielsweise Arsenophonus nasoniae für die Erzwespe Nasonia vitripennis.[2]
Die Lipopolysaccharide der äußeren Membran, genauer gesagt die darin enthaltene Komponente Lipid A wirkt als Endotoxin, das erst beim Zerfall der Bakterien, also nach ihrem Tod, freigesetzt wird. Wenn es in den Blutkreislauf gelangt, kann es einen Zytokinsturm auslösen und dadurch u. a. Fieber verursachen, die Wirkung ist also auch pyrogen. Einige wenige Spezies oder auch nur Serovare einzelner Arten bilden Enterotoxine, diese werden so genannt, da sie den Darm (griechisch: enteron) angreifen. Dazu gehört das von Shigella dysenteriae produzierte Shiga-Toxin und das von den Enterohämorrhagischen E. coli (EHEC) produzierte Vero-Toxin, beides sind Proteine. Die Vero-Toxine 1 und 2 werden aufgrund ihrer Ähnlichkeit zu denen von Shigella auch Shiga-like-toxin I/II (SLT I/II) genannt. Enterotoxische E. coli (ETEC) produzieren zwei verschiedene hitzelabile Enterotoxine (LT I und II) und ein hitzestabiles Enterotoxin (ST).[3]
Genetik
Die Desoxyribonukleinsäure (DNA) liegt in den Zellen als ein ringförmiges Bakterienchromosom vor. Das Genom von zwei Stämmen von Escherichia coli wurde im Jahr 2001 vollständig sequenziert. Die Ergebnisse der Sequenzierungen bei verschiedenen Enterobakterien zeigen einen GC-Gehalt (den Anteil der Nukleinbasen Guanin und Cytosin) in der Bakterien-DNA im Bereich von 39–59 Mol-Prozent, bei den meisten Gattungen liegt er im Bereich von 49–59 Mol-Prozent. Die DNA von Stämmen der gleichen Art kann deutlich variieren, was evolutionär begründet ist. Darauf basieren die in Laboratorien angewendeten molekularbiologischen Methoden zur Unterscheidung bzw. Identifizierung.[2]
Viele Stämme der Enterobacterales enthalten zusätzliches genetisches Material in Form von Plasmiden oder Bakteriophagen. Deren Gene codieren wichtige Proteine, wie verschiedene Toxine und Kolonisationsfaktoren, die von Bedeutung für die Pathogenität und Virulenz sind. Die Plasmide enthalten weiterhin Gene, die für Enzyme codieren, die im Stoffwechsel der Kohlenhydrate oder bei der Synthese der Zellwand beteiligt sind und Gene für die Antibiotikaresistenz. Sie können durch horizontalen Gentransfer zwischen den verschiedenen Enterobakterien ausgetauscht bzw. weitergegeben werden.[2]
Durch Verwendung phylogenetischer Methoden kann die Stammesgeschichte und die verwandtschaftlichen Beziehungen der Bakterien untereinander geklärt werden. In der 2016 veröffentlichten Beschreibung der Ordnung Enterobacterales werden fünf ‚konservierte charakteristische Indels‘ (engl. conserved signature inserts and deletions, CSI; Näheres dazu im Abschnitt Systematik und Taxonomie) festgelegt, die typisch für die Vertreter der Ordnung sind, aber nicht bei anderen Bakterien vorkommen. Die fünf CSI kommen in den Gensequenzen vor, die für die Proteine L-Arabinose-Isomerase, Elongationsfaktor-P ähnliches Protein YeiP, Peptid-ABC-Transporter Permease, Pyrophosphatase und ein hypothetisches Protein codieren.[1]
Vorkommen
Der wahrscheinlich bekannteste Vertreter der Enterobakterien ist Escherichia coli, einer der wichtigsten Modellorganismen der Genetik, Biochemie sowie der Mikrobiologie und verbunden mit der Entdeckung der Bakteriophagen, die auf ihn spezialisiert sind. Das Bakterium gehört zur normalen menschlichen Darmflora und wird daher als Indikatororganismus für fäkale Verunreinigungen z. B. in Gewässern oder Lebensmitteln verwendet. Viele Enterobakterien sind Teil der gesunden Darmflora von Menschen und Tieren. Sie sind jedoch ubiquitär verbreitet und kommen nahezu überall in der Umwelt vor, beispielsweise im Boden, im Wasser, bei Pflanzen einschließlich deren Früchte (Obst, Gemüse, Körner) und anderen Lebensmittel. Manche Arten sind pathogen für Pflanzen, vergleiche Abschnitt Pathogenität. Einige Spezies besetzen spezielle ökologische Nischen, so wurde Photorhabdus luminescens aus Nematoden isoliert, mit denen das Bakterium eine Symbiose bildet.[2] Serratia-Arten sind häufig bei verschiedenen Insekten zu finden. Weitere tierische Habitate sind Schnecken, Reptilien und Vögel, beispielsweise Hühnervögel.[3]
Systematik und Taxonomie
Verwendete Methoden der phylogenetische Systematik
Bei den Vertretern der „Enterobacteriales“ führten die Ergebnisse phylogenetischer Methoden der letzten Jahre, die insbesondere auf Untersuchung der 16S rRNA beruhten, ein für Prokaryoten typischer Vertreter der ribosomalen RNA, zu keinem zufriedenstellenden Ergebnis. Daher war es das Ziel mehrerer Wissenschaftler (Adeolu et al.) der McMaster University in Ontario, Kanada eine phylogenetische Systematik zu erstellen, die auf Vergleich von genetischen Merkmalen möglichst vieler Vertreter der Gruppe basiert. Zum Zeitpunkt der Untersuchung (2016) waren bereits 14.000 Genome von 54 den Enterobakterien zugehörigen Gattungen in der Genomdatenbank des Nationalen Zentrums für Biotechnologieinformation (NCBI) verfügbar. Für die Analyse wurden 179 repräsentative Genomsequenzen herangezogen und verschiedene molekularer Marker ausgesucht und verglichen, um jeweils einen phylogenetischen Baum zu erstellen, der die evolutionären Beziehungen zwischen den Vertretern der Enterobakterien darstellt.[1]
Zum einen wurden dafür 1548 Hauptproteine herangezogen, die typisch für die Gruppe sind und DNA-Sequenzen verglichen, die für die Proteine codieren. Analog dazu wurden 53 ribosomale Proteine (rProteine) verwendet und weiterhin wurde die DNA-Sequenzanalyse von mehreren Loci (MLSA), die für vier Proteine codieren, durchgeführt. Bei den Proteinen handelt es sich um GyrB (Gyrase B), RpoB, AtpD and InfB. Die so konstruierten drei phylogenetischen Bäume ergeben ein einheitliches Bild mit jeweils sieben Kladen, die als Enterobacter-Escherichia-Klade, Erwinia-Pantoea-Klade, Pectobacterium-Dickeya-Klade, Yersinia-Serratia-Klade, Hafnia-Edwardsiella-Klade, Proteus-Xenorhabdus-Klade und Budvicia-Klade bezeichnet werden. Darauf basierend wird die Ordnung Enterobacterales mit sieben Familien definiert. Das Ergebnis dieser phylogenetischen Analyse wird durch die Resultate der Analyse der Genomverwandtschaft unterstützt. Diese Methode erfolgt ebenfalls in silico (computerbasiert) als Ersatz für die DNA-DNA-Hybridisierung und ist hilfreich für die Einordnung taxonomisch höherer Rangstufen.[1]
Die Genomanalyse ermöglicht auch das Entdecken bzw. Festlegen von ‚konservierten (bewahrten) molekularen Eigenschaften‘, die sich innerhalb einer Gruppe von nah miteinander verwandten Organismen nicht oder kaum voneinander unterscheiden, während sie sich bei Organismen einer anderen Gruppe durch Evolution deutlich verändert haben. Für die phylogenetische Systematik der Prokaryoten werden dazu ‚konservierte charakteristische Indels‘ (engl. conserved signature inserts and deletions, CSI) verwendet. Gruppenspezifische CSI sind charakteristisch für ein bestimmtes Taxon (beispielsweise eine Ordnung), da sie in allen Vertretern dieser Gruppe und nicht in anderen Gruppen auftreten. Das ursprüngliche Indel eines gruppenspezifischen CSI trat vermutlich bereits vor der Aufspaltung im letzten gemeinsamen Vorfahren auf und wurde von den daraus entstandenen Vertretern der Gruppe übernommen („vererbt“).[5] Ein weiteres wichtiges Ergebnis der Untersuchung von Adeolu et al. ist die Entdeckung von 71 CSI, darunter fünf, die einzigartig für alle Vertreter der Ordnung Enterobacterales sind und daher für ihre Beschreibung verwendet werden. Die übrigen 66 CSI sind spezifisch für die sieben Gruppen und können bei zukünftigen Genomanalysen von neu entdeckten Spezies der Enterobakterien für deren systematische Klassifikation verwendet werden.[1]
| Kladogramm zu den Verwandtschaftsverhältnissen innerhalb der Ordnung Enterobacterales (Stand 2016)[1] | |||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| |||||||||||||||||||||||||||||||||||||||
| Das Kladogramm gibt die Anzahl der dazugehörigen konservierten charakteristischen Indels (CSI) an. Später beschriebene Familien sind nicht enthalten. |
Das Ergebnis der umfangreichen genomweiten Analysen aus dem Jahr 2016 ist Grundlage der im Folgenden dargestellte Systematik, die bereits weiter aktualisiert wurde.
Nomenklatur und Taxonomie der Ordnung
Die Gattung Enterobacter ist nicht die Typusgattung der Familie Enterobacteriacea, dies ist die Gattung Escherichia. Damit wird von der festgelegten Nomenklatur gemäß dem Prokaryotischen Code (International Code of Nomenclature of Prokaryotes, ICNP, früher: Bakteriologischer Code) abgewichen, was 1958 durch Festlegung in der Judicial Opinion 15 der Judicial Commission (in etwa „richterliche oder unparteiische Kommission“) der Internationalen Kommission für die Systematik der Prokaryoten (International Committee on Systematics of Prokaryotes, ICSP) bestätigt wurde.[6] Nach den Regeln des Prokaryotischen Codes müsste eine Ordnung mit der Typusgattung Escherichia folglich „Escherichiales“ genannt werden, eine Ordnung mit der Typusgattung Enterobacter müsste „Enterobacterales“, jedoch nicht „Enterobacteriales“ genannt werden.[6] Um die Akzeptanz der neuen Systematik sicherzustellen und die mögliche Verwirrung bei Benennung der Ordnung als „Escherichiales“ zu vermeiden, wurde der Name Enterobacterales ord. nov. mit der Typusgattung Enterobacter gewählt.[1]
Aktuelle Systematik

Zu der 2016 etablierten Ordnung der Enterobacterales gehören acht Familien (Stand 2024)[7] mit insgesamt etwa 70 Gattungen. Eine weitere Familie, die ThorselliaceaeKämpfer et al. 2015 wurde durch eine andere Gruppe von Wissenschaftlern erstbeschrieben,[8] sie ist jedoch in der List of Prokaryotic names with Standing in Nomenclature (LPSN) nicht in der Ordnung enthalten, sondern mit incertae sedis gekennzeichnet.[9] Im Taxonomy Browser des National Center for Biotechnology Information (NCBI) gehört sie zu den Enterobacterales.[10] Die Familie Gallaecimonadaceae „entstammt“ einer Genom-gestützten Phylogenie der Genome Taxonomy Database (GTDB) und wurde mit Hunderten weiterer Taxa 2023 dem ICNP entsprechend beschrieben[11] und als GallaecimonadaceaeChuvochina et al. 2024 in der Validation List no. 215 gültig veröffentlicht[12] und den Enterobacterales zugeordnet.[7]
Die neu festgelegte und damit den Regeln des Prokaryotischen Codes (ICNP) entsprechende Typusgattung der Ordnung ist die Gattung Enterobacter. Die Beschreibung der Ordnung EnterobacteralesAdeolu et al. 2016 ord. nov. entspricht der Beschreibung der Familie der Enterobacteriaceae im Bergey’s Manual of Systematic Bacteriology von 2005, mit der Erweiterung, dass die Vertreter der Ordnung von allen anderen Bakterien durch fünf CSI unterschieden werden können.[1]
Im Folgenden eine Auflistung der zugehörigen Familien mit Nennung den meisten der dazugehörigen Gattungen (Stand 2024).[7]
- BudviciaceaeAdeolu et al. 2016 fam. nov.
- BudviciaBouvet et al. 1985 emend.Lang et al. 2013, Typusgattung der Familie
- LeminorellaHickman-Brenner et al. 1985
- PragiaAldová et al. 1988
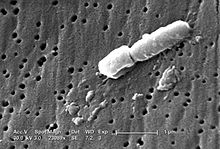
(sekundärelektronenmikroskopische Aufnahme)
- EnterobacteriaceaeRahn 1937 emend.Adeolu et al. 2016
- ButtiauxellaFerragut et al. 1982
- CedeceaGrimont et al. 1981
- CitrobacterWerkman andGillen 1932
- CronobacterIversen et al. 2008
- EnterobacterHormaeche andEdwards 1960 emend.Brady et al. 2013
- EscherichiaCastellani andChalmers 1919, Typusgattung der Familie, z. B.: Escherichia coli
- GibbsiellaBrady et al. 2011 emend.Kim et al. 2013
- KlebsiellaTrevisan 1885 (Approved Lists 1980) emend.Drancourt et al. 2001, z. B.: Klebsiella pneumoniae
- KluyveraFarmer et al. 1981
- LeclerciaTamura et al. 1987
- MangrovibacterRameshkumar et al. 2010
- Plesiomonas corrig.Habs andSchubert 1962, z. B.: Plesiomonas shigelloides
- PseudescherichiaAlnajar andGupta 2017
- RaoultellaDrancourt et al. 2001
- SaccharobacterYaping et al. 1990
- SalmonellaLignieres 1900
- ShigellaCastellani andChalmers 1919
- ShimwelliaPriest andBarker 2010
- TrabulsiellaMcWhorter et al. 1992
- YokenellaKosako et al. 1985
- ErwiniaceaeAdeolu et al. 2016 fam. nov.
- GallaecimonadaceaeChuvochina et al. 2024 fam. nov.
- GallaecimonasRodríguez-Blanco et al. 2010, Typusgattung der Familie
- HafniaceaeAdeolu et al. 2016, fam. nov.
- EdwardsiellaEwing andMcWhorter 1965
- HafniaMøller 1954, Typusgattung der Familie
- ObesumbacteriumShimwell 1963

- MorganellaceaeAdeolu et al. 2016 fam. nov.
- ArsenophonusGherna et al. 1991
- MoellerellaHickman-Brenner et al. 1984
- MorganellaFulton 1943, Typusgattung der Familie
- PhotorhabdusBoemare et al. 1993
- Proteus Hauser 1885, z. B.: Proteus vulgaris, Proteus mirabilis
- ProvidenciaEwing 1962
- XenorhabdusThomas andPoinar 1979 (Approved Lists 1980) emend.Thomas andPoinar 1983
- PectobacteriaceaeAdeolu et al. 2016 fam. nov.
- Biostraticola Verbarg et al. 2008
- BrenneriaHauben et al. 1999 emend.Brady et al. 2012 emend.Brady et al. 2015
- DickeyaSamson et al. 2005
- LonsdaleaBrady et al. 2012
- PectobacteriumWaldee 1945 (Approved Lists 1980) emend.Hauben et al. 1998, Typusgattung der Familie
- SodalisDale andMaudlin 1999

- YersiniaceaeAdeolu et al. 2016 fam. nov.
- EwingellaGrimont et al. 1984
- RahnellaIzard et al. 1981 emend.Brady et al. 2017
- SamsoniaSutra et al. 2001
- SerratiaBizio 1823, z. B.: Serratia marcescens
- Yersiniavan Loghem 1944, Typusgattung der Familie, z. B.: Yersinia pestis, Auslöser der Lungen- und Beulenpest
Historische Entwicklung und einige Synonyme
Der Name Enterobacteriaceae für die Familie, deren Vertreter nun der Ordnung Enterobacterales angehören, wurde 1937 von Rahn vorgeschlagen. Allerdings sind einige Arten innerhalb dieser Gruppe schon davor beschrieben worden, beispielsweise wurde Serratia marcescens 1823 von Bizio so benannt und erstbeschrieben. Andere „alte“ Vertreter sind Yersinia pseudotuberculosis (1883), Salmonella Typhi (1884), Salmonella cholerae-suis, Proteus vulgaris und Proteus mirabilis (alle 1885 erstbeschrieben) sowie Yersinia pestis (1894). Escherichia coli wurde 1885 erstbeschrieben, dabei jedoch von Migula als „Bacillus coli“ bezeichnet, dieser Name gilt als Basonym. Diese durch die Regeln des Bakteriologischen Codes (mittlerweile der Prokaryotische Code) notwendigen Änderungen in der Nomenklatur führten zu zahlreichen Synonymen für Arten oder Gattungen, was Kritiker als „quälenden Prozess“ (torturous process) bezeichnen, was jedoch mit dem erweiterten Wissen über die Bakterien erklärbar ist.[2]
Hier einige Beispiele für Synonyme und Umstellungen (z. B. Combinatio nova): Alle Arten von LevineaYoung et al. 1971 wurden zu der Gattung CitrobacterWerkman & Gillen 1932 gestellt. Verschiedene Erwinia-Arten wurden in die Gattungen Pantoea, Enterobacter, Pectobacterium und Brenneria aufgeteilt. Liquidobacterium genauso wie Cosenzaea ist ein Synonym für Proteus. Die Gattung CalymmatobacteriumAragão & Vianna 1913 ist ein Synonym für Klebsiella.[7]
Medizinische Bedeutung
In der Ordnung der Enterobacterales finden sich einige bedeutende Krankheitserreger. Nach Schätzungen aus dem Jahr 2010 machen sie etwa 80 % der klinisch relevanten isolierten gramnegativen Bakterien aus und etwa 50 % insgesamt, bezogen auf alle Bakterien. Enterobakterien findet man bei knapp 50 % der Fälle von Septikämie und bei etwa 70 % der Fälle von Harnwegsinfektionen, vor allem werden intestinale Infektionen (Infektionen des Darms) durch sie verursacht. Zu den weiteren beim Menschen durch Enterobakterien verursachten Krankheiten gehören Diarrhoe (Durchfall), Meningitis, Bakteriämie, Pneumonie (Lungenentzündung), Wundinfektionen und Abszesse. Sie kommen vielfach als nosokomiale Erreger vor („Krankenhauskeime“) und befallen Menschen mit schwachem Immunsystem.[2]
Bedeutsame Infektionskrankheiten, die durch bestimmte Arten der Enterobakterien verursacht werden, sind unter anderem Lungen- und Beulenpest, Typhus und die Bakterienruhr, vergleiche Abschnitt Pathogenität. Durch Infektion mit Enterotoxin-bildenden Stämmen kann vor allem bei Kindern das hämolytisch-urämische Syndrom (HUS) ausgelöst werden.
Einige Enterobakterien verursachen extraintestinale Infektionen, also Infektionen außerhalb des Darms. Im Vergleich zur Vielzahl an Spezies in der Ordnung Enterobacterales sind dies nur wenige Arten. Dazu gehören beispielsweise Escherichia coli, Klebsiella pneumoniae, Proteus mirabilis, Klebsiella aerogenes (Synonym: Enterobacter aerogenes), Vertreter des Enterobacter cloacae Komplexes und Serratia marcescens.[2]
Antibiotikaresistenz und nosokomiale Infektionen
Einige Arten der Enterobakterien sind als Erreger von nosokomialen Infektionen von großer Bedeutung, sie machen etwa 50 % dieser Infektionen aus (Stand 2005).[3] In Deutschland gibt es seit 2016 eine Meldepflicht (vergleiche nächster Abschnitt), so dass mittlerweile durch das Robert Koch-Institut Fälle erfasst und Daten im Infektionsepidemiologischen Jahrbuch veröffentlicht werden. In dem Dokument für 2020 wird auf die Bedeutung der Carbapeneme als sogenannte Reserveantibiotika aufmerksam gemacht. Diese Antibiotika können durch Carbapenemasen (bakterielle Enzyme) gespalten werden, dadurch sind die Bakterien resistent oder zeigen zumindest eine verminderte Empfindlichkeit gegenüber den Carbapenemen, was deren therapeutischen Einsatz verhindert. Daher sind Carbapenemase-produzierende Bakterienstämme im Krankenhaus von großer Bedeutung. Unter den Enterobakterien sind dies vor allem Stämme von E. coli (65 % produzieren Carbapenemase) und K. pneumoniae (50 %), während dies bei K. aerogenes nur auf 3 % der Stämme zutrifft. Im Infektionsepidemiologischen Jahrbuch 2020 werden insgesamt 3.590 Fälle von Enterobacterales mit verminderter Carbapenem-Empfindlichkeit aufgeführt. Zum Vergleich: Bei Acinetobacter-Arten (z. B. Acinetobacter baumannii) wurden 481 Fälle mit verminderter Carbapenem-Empfindlichkeit erfasst.[13] Die Beschränkung der Daten auf Carbapenemase-produzierende Stämme ermöglicht keine generalisierte Aussagen zur Relevanz der Enterobakterien für nosokomiale Infektionen. Bei Daten über das Vorkommen von MRGN (multiresistente gramnegative Bakterien) ist zu beachten, dass zu dieser Gruppe neben den Enterobakterien auch Pseudomonas-, Acinetobacter- und weitere Arten gehören.
Neben erworbenen Antibiotikaresistenzen können Enterobakterien auch über eine primäre Resistenz (intrinsische Resistenz) verfügen, als genetisches Merkmal, das unabhängig vom klinischen Einsatz der Antibiotika ist. So sind beispielsweise mehrere oder sogar alle Arten aus den Gattungen Buttiauxella, Cedecea, Citrobacter, Enterobacter, Ewingella, Hafnia, Morganella, Providencia und Serratia resistent gegen Cefalotin. Weiterhin sind Cedecea-Arten, Citrobacter amalonaticus, Escherichia hermannii, Klebsiella pneumoniae, Kluyvera ascorbata, Kluyvera cryocrescens, Proteus vulgaris, Morganella morganii und Serratia fonticola resistent gegen Ampicillin und Edwardsiella tarda sogar gegen Colistin.[2]
Meldepflicht
In Deutschland ist der direkte Nachweis von Enterobacterales namentlich meldepflichtig nach § 7 des Infektionsschutzgesetzes (IfSG), aber nur bei Nachweis einer Carbapenemase-Determinante oder mit verminderter Empfindlichkeit gegenüber Carbapenemen außer bei natürlicher Resistenz. Die Meldepflicht besteht nur bei Infektion oder Kolonisation (§ 7 Abs. 1 Satz 1 Nr. 52 b) IfSG). Zudem ist das Auftreten von zwei oder mehr nosokomialen Infektionen nichtnamentlich zu melden, bei denen ein epidemischer Zusammenhang wahrscheinlich ist oder vermutet wird (§ 6 Absatz 3 IfSG).
Quellen
Literatur
- Volume 6: Proteobacteria: Gamma Subclass. In: Martin Dworkin, Stanley Falkow, Eugene Rosenberg, Karl-Heinz Schleifer, Erko Stackebrandt (Hrsg.): The Prokaryotes. A Handbook on the Biology of Bacteria. 3. Auflage. Springer-Verlag, New York 2006, ISBN 978-0-387-25496-8, doi:10.1007/0-387-30746-X_9.
- J. J. Farmer III, M. K. Farmer, Barry Holmes: The Enterobacteriaceae: general characteristics. In: S. Peter Borriello, Patrick R. Murray, Guido Funke (Hrsg.): Topley & Wilson's Microbiology and Microbial Infections - Bacteriology Volume 2. 10. Auflage. John Wiley & Sons, Ltd, Chichester, UK 2010, ISBN 978-0-470-68638-6.
- Michael T. Madigan, John M. Martinko, Jack Parker: Brock – Mikrobiologie, Spektrum Akademischer Verlag, Heidelberg, Berlin 2001, ISBN 3-8274-0566-1
Einzelnachweise
- ↑ a b c d e f g h M. Adeolu, S. Alnajar, S. Naushad, R. S. Gupta: Genome-based phylogeny and taxonomy of the ‘Enterobacteriales’: proposal for Enterobacterales ord. nov. divided into the families Enterobacteriaceae, Erwiniaceae fam. nov., Pectobacteriaceae fam. nov., Yersiniaceae fam. nov., Hafniaceae fam. nov., Morganellaceae fam. nov., and Budviciaceae fam. nov. In: International Journal of Systematic and Evolutionary Microbiology. Band 66, Dezember 2016, S. 5575–5599, doi:10.1099/ijsem.0.001485.
- ↑ a b c d e f g h i j k l J. J. Farmer III, M. K. Farmer, Barry Holmes: The Enterobacteriaceae: general characteristics. In: Topley & Wilson's Microbiology and Microbial Infections - Bacteriology Volume 2. 10. Auflage. John Wiley & Sons, Ltd, Chichester, UK 2010, ISBN 978-0-470-68638-6, S. 1317–1359.
- ↑ a b c d e f Herbert Hof, Rüdiger Dörries: Enterobacteriaceae. In: Duale Reihe: Medizinische Mikrobiologie. 3. Auflage. Thieme Verlag, Stuttgart 2005, ISBN 978-3-13-125313-2, S. 374–400.
- ↑ a b TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen. In: Webseite der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. August 2015, abgerufen am 27. März 2024 (letzte Änderung vom 20. März 2023).
- ↑ Radhey S. Gupta, Emma Griffiths: Critical Issues in Bacterial Phylogeny. In: Theoretical Population Biology. Band 61, Nr. 4, Juni 2002, S. 423–434, doi:10.1006/tpbi.2002.1589, PMID 12167362.
- ↑ a b International Code of Nomenclature of Prokaryotes. Prokaryotic Code (2022 Revision). In: Aharon Oren, David R. Arahal, Markus Göker, Edward R. B. Moore, Ramon Rossello-Mora, Iain C. Sutcliffe (Hrsg.): International Journal of Systematic and Evolutionary Microbiology. Band 73, 23. Mai 2023, S. 005585, doi:10.1099/ijsem.0.005585.
- ↑ a b c d Aidan C. Parte, Joaquim Sardà Carbasse, Jan P. Meier-Kolthoff, Lorenz C. Reimer, Markus Göker: List of Prokaryotic names with Standing in Nomenclature (LPSN) moves to the DSMZ. In: International Journal of Systematic and Evolutionary Microbiology. Band 70, Nr. 11, 1. November 2020, S. 5607–5612, doi:10.1099/ijsem.0.004332 (dsmz.de [abgerufen am 4. April 2024]).
- ↑ P. Kämpfer, S. P. Glaeser, L. K. J. Nilsson, T. Eberhard, S. Håkansson, L. Guy, S. Roos, H.-J. Busse, O. Terenius: Proposal of Thorsellia kenyensis sp. nov. and Thorsellia kandunguensis sp. nov., isolated from larvae of Anopheles arabiensis, as members of the family Thorselliaceae fam. nov. In: International Journal of Systematic and Evolutionary Microbiology. Band 65, Februar 2015, S. 444–451, doi:10.1099/ijs.0.070292-0.
- ↑ LPSN: Family Thorselliaceae. In: DSMZ. Abgerufen am 4. April 2024.
- ↑ Conrad L. Schoch et al.: NCBI Taxonomy: a comprehensive update on curation, resources and tools. In: Database: The Journal of Biological Databases and Curation. Band 2020, 1. Januar 2020, S. baaa062, PMID 32761142 (nih.gov [abgerufen am 4. April 2024]).
- ↑ Maria Chuvochina, Aaron J. Mussig, Pierre-Alain Chaumeil, Adam Skarshewski, Christian Rinke, Donovan H. Parks, Philip Hugenholtz: Proposal of names for 329 higher rank taxa defined in the Genome Taxonomy Database under two prokaryotic codes. In: FEMS Microbiology Letters. Band 370, 17. Januar 2023, S. 1–33, doi:10.1093/femsle/fnad071, PMID 37480240.
- ↑ Aharon Oren, Markus Göker: Validation List no. 215. Valid publication of new names and new combinations effectively published outside the IJSEM. In: International Journal of Systematic and Evolutionary Microbiology. Band 74, 1. Februar 2024, S. 006173, doi:10.1099/ijsem.0.006173.
- ↑ Infektionsepidemiologisches Jahrbuch für 2020. Robert Koch-Institut, 20. September 2021, abgerufen am 5. April 2024.
Weblinks
Auf dieser Seite verwendete Medien
Autor/Urheber: Bacteria in Photos, Lizenz: CC0
Morganella morganii ssp. morganii on blood agar. Cultivation 24 hours, 37°C.
Electonmicroscopic picture of bacteria Klebsiella pneumoniae
Autor/Urheber:
- Bacteria-.svg: JrPol
- derivative work: Matthias M.
schematische Darstellung eines Bakteriums aus der Familie der Enterobacteriaceae mit Abkürzungen für die Antigenstrukturen: K (Kapsel), O (somatisch), F (Fimbrien), H (Flagellen)
Autor/Urheber: A doubt, Lizenz: CC BY-SA 4.0
Cosenzaea myxofaciens (vorher als Proteus myxofaciens klassifizert) auf MacConkey-Agar, mit negativem Ergebnis (der Mikroorganismus kann keine Lactose verwerten, es erfolgt kein Farbumschlag des pH-Indikators Neutralrot).
Autor/Urheber: Microrao, Lizenz: CC BY-SA 4.0
Proteus swarming on Blood agar
Mikroskopisches Bild einer nach Gram gefärbten Bakterienkultur, dabei lassen sich zahlreiche, gramnegative Yersinia enterocolitica Bakterienzellen erkennen.
Color-enhanced scanning electron micrograph showing Salmonella Typhimurium (red) invading cultured human cells Credit: Rocky Mountain Laboratories, NIAID, NIH All the images, except specified ones from the World Health Organization (WHO), are in the public domain. For the public domain images, there is no copyright, no permission required, and no charge for their use.
Autor/Urheber: Ajay Kumar Chaurasiya, Lizenz: CC BY-SA 4.0
Escherichia coli growth on MacConkey agar a day f incubation at 37°C
Rough and smooth colony growth of en:Enterobacter cloacae bacteria on en:Tryptic Soy Broth agar.
Obtained from the CDC Public Health Image Library.
Image credit: CDC(PHIL #6552), 1983.