DRADA
| DRADA | ||
|---|---|---|
 | ||
Vorhandene Strukturdaten: 1QBJ, 1QGP, 1XMK, 2ACJ, 2GXB, 2L54, 2MDR, 3F21, 3F22, 3F23, 3IRQ, 3IRR | ||
| Eigenschaften des menschlichen Proteins | ||
| Masse/Länge Primärstruktur | 1226 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Homodimer | |
| Kofaktor | Zn2+ | |
| Isoformen | 5 | |
| Bezeichner | ||
| Gen-Namen | ADAR ADAR1; AGS6; DRADA; DSH; DSRAD; G1P1; IFI-4; IFI4; K88DSRBP; P136 | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 3.5.4.4, Adenosin-Desaminase | |
| Vorkommen | ||
| Übergeordnetes Taxon | Euteleostomi | |
| Orthologe | ||
| Mensch | Hausmaus | |
| Entrez | 103 | 56417 |
| Ensembl | ENSG00000160710 | ENSMUSG00000027951 |
| UniProt | P55265 | Q99MU3 |
| Refseq (mRNA) | NM_001025107 | NM_001038587 |
| Refseq (Protein) | NP_001020278 | NP_001033676 |
| Genlocus | Chr 1: 154.58 – 154.63 Mb | Chr 3: 89.72 – 89.75 Mb |
| PubMed-Suche | 103 | 56417 |
DRADA (Abk. für Doppelsträngige RNA-spezifische Adenosin-Desaminase) ist ein Enzym in den Zellen von Wirbeltieren. DRADA wandelt Adenosin-Nukleotidbausteine innerhalb von RNA zu Inosin um. Diese Reaktion ist einerseits Teil des RNA-Editing eigener mRNA im Zellkern. Andererseits kann damit die Erbinformation von in die Zelle eingedrungener Virus-RNA zerstört werden. Beim Menschen kommt DRADA in allen Gewebetypen vor, insbesondere aber in aktivierten T-Zellen, im Gehirn und der Lunge. Mutationen im ADAR-Gen können zu Enzymmangel, und dieser zu einer (seltenen) Hautkrankheit führen.[1][2]
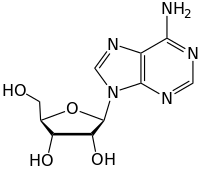
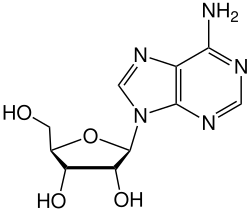
- Desaminierung von Adenosin zu Inosin
Inosin wird während der Translation von RNA als Guanosin betrachtet. Daher erzeugt eine Änderung der RNA von Adenosin zu Inosin am Ende ein verändertes Protein. Dadurch wird die Protein-Diversität im Organismus beträchtlich erhöht. Die durch DRADA erreichte Vielzahl an Proteinen ist für die Organismen lebensnotwendig, da beispielsweise das Glutamat-Rezeptor-Protein nur so erzeugt werden kann.[1]
Es gibt Hinweise darauf, dass Verminderung der Enzymaktivität in Mäusen hilfreich bei der Melanom-Therapie sein könnte. Überraschenderweise ist DRADA notwendig für das HI-Virus, um sich zu vervielfältigen. Offensichtlich wird der RNA-Editing-Mechanismus vom Virus zum Spleißen seiner RNA benutzt.[3][4]
Literatur
- A. Gallo, S. Galardi: A-to-I RNA editing and cancer: from pathology to basic science. In: RNA Biol., Band 5, Nr. 3, 2008, S. 135–139. PMID 18758244.
Weblinks
Einzelnachweise
- ↑ a b UniProt P55265
- ↑ Laxminarayana D, Khan IU, O’Rourke KS, Giri B: Induction of 150-kDa adenosine deaminase that acts on RNA (ADAR)-1 gene expression in normal T lymphocytes by anti-CD3-epsilon and anti-CD28. In: Immunology. 122. Jahrgang, Nr. 4, Dezember 2007, S. 623–633, doi:10.1111/j.1365-2567.2007.02709.x, PMID 17897325, PMC 2266038 (freier Volltext).
- ↑ Hong J, Zhao Y, Li Z, Huang W: esiRNA to eri-1 and adar-1 genes improving high doses of c-myc-directed esiRNA effect on mouse melanoma growth inhibition. In: Biochem. Biophys. Res. Commun. 361. Jahrgang, Nr. 2, September 2007, S. 373–378, doi:10.1016/j.bbrc.2007.07.003, PMID 17658462.
- ↑ Phuphuakrat A, Kraiwong R, Boonarkart C, Lauhakirti D, Lee TH, Auewarakul P: Double-stranded RNA adenosine deaminases enhance expression of human immunodeficiency virus type 1 proteins. In: J. Virol. 82. Jahrgang, Nr. 21, November 2008, S. 10864–10872, doi:10.1128/JVI.00238-08, PMID 18753201.
Auf dieser Seite verwendete Medien
Autor/Urheber: Emw, Lizenz: CC BY-SA 3.0
Structure of the ADAR protein. Based on PyMOL rendering of PDB 1qbj.
Struktur von Adenosin
Struktur von Inosin