Cre/loxP-System
Das Cre/loxP-System ist ein Rekombinations-System. Es ermöglicht das gezielte Entfernen von DNA-Sequenzen in lebenden Organismen. Mit dieser Technik können beispielsweise einzelne spezifische Zell- oder Gewebearten genetisch modifiziert werden, während andere Gewebe davon unberührt bleiben. Ursprünglich stammt dieses System aus dem Bakteriophagen P1 und findet heute v. a. in der molekularbiologischen Forschung und zur Herstellung gentechnisch veränderter Pflanzen Anwendung. Ein analoges System ist das Flp-FRT-System.
Prinzip
Cre (von engl. cyclization recombination oder causes recombination) bzw. Flp (benannt nach der Flippase-Aktivität, durch die Hefen Sequenzabschnitte invertieren) sind Enzyme der Klasse der Rekombinasen. Diese Proteine, die natürlicherweise in allen Organismen vorkommen, katalysieren die Spaltung und Neuverknüpfung von DNA zwischen spezifischen Basensequenzen. Diese Erkennungssequenz wird als loxP bzw. FRT bezeichnet. Das Cre/loxP-System wurde in den 1980er Jahren entwickelt und von DuPont patentiert.
Soll aus einem DNA-Strang eine bestimmte DNA-Sequenz bzw. ein bestimmtes Gen entfernt werden, setzt man gezielt vor und hinter diesen DNA-Abschnitt eine loxP-Stelle, was als floxen bezeichnet wird (floxed = flanked by loxP; dt. ‚von loxP-Stellen flankiert‘). Das Cre-Enzym erkennt und bindet die jeweiligen loxP-Stellen. Dabei schneidet es die entsprechende DNA-Sequenz bzw. das Gen heraus, wenn die beiden loxP-Sequenzen in gleicher Richtung orientiert sind (Exzision). Die beiden verbleibenden loxP-Enden werden zusammengefügt, der entstehende kurze ringförmige DNA-Abschnitt wird in der Zelle abgebaut. Sind die beiden loxP-Sequenzen auf der DNA in entgegengesetzter Richtung orientiert, katalysiert das Enzym die Inversion (d. h. das Vertauschen der beiden Enden) des gefloxten Abschnittes, der um 180° gedreht im DNA-Strang verbleibt.
Cre-Rekombinase
Die Cre-Rekombinase (von englisch causes recombination oder cyclization recombinase)[1][2] besteht aus vier Untereinheiten mit je 343 Aminosäuren in zwei Proteindomänen. Die C-terminale Domäne der Cre-Rekombinase enthält das aktive Zentrum und ist strukturell ähnlich zur Integrase-Familie aus dem Bakteriophagen λ.
loxP
Die loxP-DNA-Sequenz (von engl. locus of X-over P1) entstammt dem Bakteriophagen P1 und besteht aus 34 Basenpaaren, einschließlich einer asymmetrischen Sequenz von acht Basenpaaren (die beiden mittigen davon sind konserviert), die beidseitig von palindromischen dreizehn Basen flankiert sind.
| 13bp | 8bp | 13bp |
| ATAACTTCGTATA - | NNNTANNN | -TATACGAAGTTAT |
| Name | 13bp Erkennungsregion | 8bp Spacer-Region | 13bp Erkennungsregion |
|---|---|---|---|
| Lox 511 | ATAACTTCGTATA | ATGTATaC | TATACGAAGTTAT |
| Lox 5171 | ATAACTTCGTATA | ATGTgTaC | TATACGAAGTTAT |
| Lox 2272 | ATAACTTCGTATA | AaGTATcC | TATACGAAGTTAT |
| M2 | ATAACTTCGTATA | AgaaAcca | TATACGAAGTTAT |
| M3 | ATAACTTCGTATA | taaTACCA | TATACGAAGTTAT |
| M7 | ATAACTTCGTATA | AgaTAGAA | TATACGAAGTTAT |
| M11 | ATAACTTCGTATA | aGATAgaa | TATACGAAGTTAT |
| Lox71 | taccgTTCGTATA | NNNTANNN | TATACGAAGTTAT |
| Lox66 | ATAACTTCGTATA | NNNTANNN | TATACGAAcggta |
Anwendungen
Pflanzen
Das Cre/loxP-System wird beispielsweise dazu verwendet, gezielt Markergene aus transgenen Pflanzen zu eliminieren. So ist es möglich, transgene Organismen zu erzeugen, ohne dass anschließend eine Selektion auf Grundlage von Herbizid- oder Antibiotikaresistenzen notwendig wird. Hierzu muss zunächst Cre-Rekombinase vorübergehend (transient) in die Zelle übertragen werden. Diese Übertragung kann sowohl mit Hilfe eines Pflanzenvirus (PVX, TMV) als auch durch Agrobacterium tumefaciens erfolgen. Die durch die Rekombinase Cre ausgelösten Rekombinationsereignisse können auf die nächste Generation über Regeneration oder Selbstbestäubung übertragen werden. Die Nachkommen aus der Selbstbestäubung sind dann, wenn alle Reaktionsschritte erfolgreich verlaufen sind, markerfrei, tragen aber das interessierende Gen.
Tiere
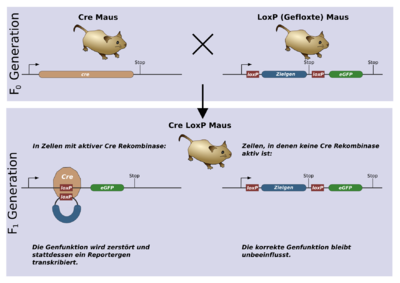
Das System funktioniert auch in Säugetierzellen zuverlässig und ohne weitere Kofaktoren. Mittlerweile ist es zur Herstellung gewebsspezifischer Knockout-Mäuse weit verbreitet. In welchem Gewebe und zu welchem Zeitpunkt in einem Lebewesen ein Gen angeschaltet wird, hängt hauptsächlich von dem zugehörigen Promotor ab. Promotor und Cre werden zusammen als Transgen in das Mausgenom eingebracht. Sollen z. B. Gene nur im Gehirn ausgeschaltet werden, sucht man sich ein Protein, das nur dort vorkommt und setzt den zugehörigen Promotor vor das Cre-Gen, wodurch Cre-Rekombinase nur im Gehirn gebildet wird.
Nun wird noch eine zweite Mauslinie benötigt, bei der das Gen, das ausgeschaltet werden soll, gefloxt ist. Werden die beiden Mäuse nun verpaart, erhält man unter anderem Nachkommen, die beide genetische Veränderungen in ihrem Erbgut tragen. Ein Protein, das in jeder Körperzelle vorkommt, wird so beispielsweise nur im Gehirn nicht mehr gebildet, da das Gen dort durch die Rekombinase unbrauchbar gemacht wird.
Ein Nachteil hierbei ist, dass die meisten Promotoren schon bald in der Embryonalentwicklung aktiviert werden. Wird ein wichtiges Gen schon früh ausgeschaltet, sind die Tiere oft nicht lebensfähig und können nicht untersucht werden. Um dieses Problem zu umgehen, wurden Methoden erfunden, dass Cre zu einem beliebigen Zeitpunkt angeschaltet werden kann. Dies gelingt z. B. mit Liganden-aktivierbaren Cre-Rekombinasen. Cre wurde dabei mit der modifizierten Ligandenbindungsdomäne (LBD) von Estrogenrezeptoren fusioniert, an die nicht mehr körpereigenes Estrogen bindet, wohl aber synthetisches Tamoxifen, ein Antiestrogen. Die Estrogenrezeptoren befinden sich im Zytoplasma und werden erst nach Bindung des Liganden in den Zellkern überführt, wo sich die DNA befindet. Somit verbleibt auch das mit dem Estrogenrezeptor assoziierte Cre-Enzym im Zytoplasma, bis den Tieren Tamoxifen verabreicht wird. Sobald dieses an den modifizierten Rezeptor bindet, wird er zusammen mit Cre in den Zellkern gebracht, wo Cre dann die gefloxten Gene ausschneiden kann.
Siehe auch
Literatur
- D. Metzger, P. Chambon: Site- and time-specific gene targeting in the mouse. In: Methods, 24(1), 2001, S. 71–80, PMID 11327805.
- R. Feil, J. Wagner, D. Metzger, P. Chambon: Regulation of Cre recombinase activity by mutated estrogen receptor ligand-binding domains. In: Biochem Biophys Res Commun, 237(3), 1997, S. 752–757, PMID 9299439.
Weblinks
Einzelnachweise
- ↑ Cre-Rekombinase in der GenBank.
- ↑ N. Sternberg, D. Hamilton: Bacteriophage P1 site-specific recombination. I. Recombination between loxP sites. In: Journal of molecular biology. Band 150, Nummer 4, August 1981, S. 467–486, ISSN 0022-2836. PMID 6276557.
- ↑ P. I. Missirlis, D. E. Smailus, R. A. Holt: A high-throughput screen identifying sequence and promiscuity characteristics of the loxP spacer region in Cre-mediated recombination. In: BMC genomics. Band 7, 2006, S. 73, ISSN 1471-2164. doi:10.1186/1471-2164-7-73. PMID 16595017. PMC 1479339 (freier Volltext).
Auf dieser Seite verwendete Medien
Autor/Urheber: Matthias Zepper (Curnen). Zur Erstellung wurde das Mouse.svg von Madprime verwendet., Lizenz: CC BY-SA 3.0
Illustration eines Genetikversuchs, der sich des Cre / LoxP Systems bedient. Die Funktion eines Zielgens wird durch einen konditionalen Knock-out zerstört. Typischerweise würde in einem solchen Experiment ein gewebespezifischer Promotor eingesetzt, um die Expression der Cre-Rekombinase zu steuern. Auch eine zeitliche Steuerung der Aktivität über einen nur zu einem bestimmten Ontogenesestadium aktiven Promotor wäre denkbar.