Chlorovirus
| Chlorovirus | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 Paramecium bursaria chlorella virus-1 | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Chlorovirus | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Chlorovirus, auch Chlorellavirus genannt, ist eine Gattung von Viren mit doppelsträngigem DNA-Genom in der Familie der Phycodnaviridae. Sie gehört damit zum Phylum Nucleocytoviricota (alias Nucleocytoplasmic large DNA viruses, NCLDV).[3][4]
Diese Gattung ist weltweit in Süßwasserumgebungen anzutreffen, wo mikroskopische Algen als natürliche Wirte dienen. Es gibt derzeit 19 Spezies in dieser Gattung, einschließlich der der ehemaligen Typusspezies Chlorovirus vanettense (mit Paramecium bursaria Chlorella virus 1).[5][6][7] Der Wortbestandteil Chloro- ist abgeleitet von altgriechisch χλωρόςchlōrós, deutsch ‚gelblich‘, ‚grün‘.
Das erste Chlorovirus wurde 1981 von Russel H. Meintz, James L. Van Etten, Daniel Kuczmarski, Kit Lee und Barbara Ang entdeckt, als sie versuchten, Chlorella-ähnliche Algen zu kultivieren. Während des Verfahrens wurden Viruspartikel (Virionen) in den Zellen 2 bis 6 Stunden nach der anfänglichen Isolierung entdeckt, gefolgt von der Lyse (Tod durch Auflösung der Wirtszelle) nach 12 bis 20 Stunden. Dieses Virus wird als HVCV-1 (Hydra viridis Chlorella virus 1) bezeichnet, da in dem Süßwasserpolyp Hydra viridis lebende einzellige Grünalgen (Zoochlorellen) infiziert wurden.[8][9] Vor einiger Zeit wurde festgestellt, dass eine Spezies, das in Seen häufig vorkommende Spezies ATCV-1 (Acanthocystis turfacea chlorella virus 1), auch Menschen infiziert. Genauere Untersuchungen sind aber noch nötig.[10] Es folgten neuere Studien über die Auswirkungen von Infektionen im Mausmodell.[10][11]
Systematik
Innere Systematik
Das International Committee on Taxonomy of Viruses (ICTV) hat mit Stand 30. April 2024 (Master Species List #39v1) folgende Spezies anerkannt:[12][13]
Gattung Chlorovirus
- Spezies Chlorovirus americanus mit Paramecium bursaria Chlorella virus NY2A (PBCV-NY2A)[14][15]
- Spezies Chlorovirus conductrix mit Paramecium bursaria Chlorella virus A1 (PBCV-A1)[14] mit Isolaten CVA-1, FR483,[16][15] MT325[17]
- Spezies Chlorovirus heliozoae mit Acanthocystis turfacea chlorella virus 1 (ATCV1, ATCV-1), assoziiert mit Acanthocystis turfacea[17][18][14][15]
- Spezies Chlorovirus illinoense mit Paramecium bursaria Chlorella virus IL3A (PBCV-IL3A, IL-3A)
- Spezies Chlorovirus newyorkense mit Paramecium bursaria Chlorella virus NYs1 (PBCV-NYs1)[14]
- Spezies Chlorovirus vanettense (ehem. Typus) mit Paramecium bursaria Chlorella virus 1 (PBCV-1), Hauptwirt Paramecium bursaria,[14][15][17] mit Isolat PBCV1 und Mutante CME6
Anders als früher werden vom ICTV nicht mehr geführt, da unzureichend charakterisiert:
- Spezies „Hydra viridis Chlorella virus 1“ (HVCV-1), Hauptwirt Hydra viridissima
- Spezies „Paramecium bursaria Chlorella virus AL1A“ (PBCV-AL1A)
- Spezies „Paramecium bursaria Chlorella virus AL2A“ (PBCV-AL2A)
- Spezies „Paramecium bursaria Chlorella virus BJ2C“ (PBCV-BJ2C)
- Spezies „Paramecium bursaria Chlorella virus CA4A“ (PBCV-CA4A)
- Spezies „Paramecium bursaria Chlorella virus CA4B“ (PBCV-CA4B)
- Spezies „Paramecium bursaria Chlorella virus NC1A“ (PBCV-NC1A)
- Spezies „Paramecium bursaria Chlorella virus NE8A“ (PBCV-NE8A)
- Spezies „Paramecium bursaria Chlorella virus SC1A“ (PBCV-SC1A)
- Spezies „Paramecium bursaria Chlorella virus XY6E“ (PBCV-XY6E)
- Spezies „Paramecium bursaria Chlorella virus XZ3A“ (PBCV-XZ3A)
- Spezies „Paramecium bursaria Chlorella virus XZ4A“ (PBCV-XZ4A)
- Spezies „Paramecium bursaria Chlorella virus XZ4C“ (PBCV-XZ4C)
Dazu kommen weitere Vorschläge, wie:
- Spezies „Acanthocystis turfacea Chlorella virus Br0604L“[19][2]
- Spezies „Acanthocystis turfacea Chlorella virus Can0610SP“[20][2]
- Spezies „Acanthocystis turfacea Chlorella virus Canal-1“[21][2]
- Spezies „Acanthocystis turfacea Chlorella virus GM0701.1“[22][2]
- Spezies „Acanthocystis turfacea Chlorella virus MO0605SPH “[23][2]
- Spezies „Acanthocystis turfacea Chlorella virus MN0810.1“[24][2]
- Spezies „Acanthocystis turfacea Chlorella virus NTS-1“[25][2]
- Spezies „Acanthocystis turfacea Chlorella virus NE-JV-2“[26][2]
- Spezies „Acanthocystis turfacea Chlorella virus NE-JV-3“[27][28]
- Spezies „Acanthocystis turfacea Chlorella virus OR0704.3“[29][2]
- Spezies „Acanthocystis turfacea Chlorella virus TN603“ (ATCV-TN603)[30][15]
- Spezies „Acanthocystis turfacea Chlorella virus TN603.4.2“[31][2]
- Spezies „Acanthocystis turfacea Chlorella virus WI0606“[32][2]
- Spezies „Paramecium bursaria Chlorella virus AN69C“ (alias „Chlorella virus AN69C“)[33][2]
- Spezies „Paramecium bursaria Chlorella virus AR158“ (PBCV-AR158)[34][15][2]
- Spezies „Paramecium bursaria Chlorella virus CZ-2“[35][28]
- Spezies „Paramecium bursaria Chlorella virus AP110A“[36][2]
- Spezies „Paramecium bursaria Chlorella virus Can18-4“[37][2]
- Spezies „Paramecium bursaria Chlorella virus CVB-1“[38][2]
- Spezies „Paramecium bursaria Chlorella virus CVG-1“[39][2]
- Spezies „Paramecium bursaria Chlorella virus CviKI“[40][2]
- Spezies „Paramecium bursaria Chlorella virus CVK2“ (PBCV-CVK2)[41][15]
- Spezies „Paramecium bursaria Chlorella virus CVM-1“[42][2]
- Spezies „Paramecium bursaria Chlorella virus CVR-1“[43][2]
- Spezies „Paramecium bursaria Chlorella virus CvsA1“[44][2]
- Spezies „Paramecium bursaria Chlorella virus CZ-2“[45][2]
- Spezies „Paramecium bursaria Chlorella virus Fr5L“[46][2]
- Spezies „Paramecium bursaria Chlorella virus IL-5-2s1“[47][2]
- Spezies „Paramecium bursaria Chlorella virus KS1B“[48][2]
- Spezies „Paramecium bursaria Chlorella virus MA-1D“[49][2]
- Spezies „Paramecium bursaria Chlorella virus MA-1E“[50][2]
- Spezies „Paramecium bursaria Chlorella virus MT325“ (PBCV-MT325)[41][15]
- Spezies „Paramecium bursaria Chlorella virus NE-JV-1“[51][2]
- Spezies „Paramecium bursaria Chlorella virus NE-JV-4“ mit Stamm NE-JV4[52][2]
- Spezies „Paramecium bursaria Chlorella virus NW665.2“[53][2]
- Spezies „Paramecium bursaria Chlorella virus NY-2B“[54][2]
- Spezies „Paramecium bursaria Chlorella virus NYs1“[55] mit Stamm NYs-1[2]
- Spezies „Paramecium bursaria Chlorella virus OR0704.2.2“[56][2]
- Spezies „Chlorella variabilis Virus NC64A“ (s. u.)
- Spezies „Chlorella variabilis Virus Syngen“ (s. u., Hauptwirt in der Gattung Chlorella)
- Spezies „Chlorella heliozoae Virus SAG“ (s. u.)
- Spezies „Only Syngen Nebraska virus 5“ (OSy-NE5)[57][2][17]
Phylogenetischer Baum nach Hao Chen et al. (2018):[14]
| Chlorovirus |
| ||||||||||||||||||||||||
Phylogenetischer Baum (Maximum-Likelihood) der Chloroviren (A) und ihrer Algenwirte (B) nach Van Etten at al. (2019) – als Outgroups dienten vier Ostreococcus-Virus-Sequenzen (Gattung Prasinovirus) in (A), Parachlorella-Arten in (B).
Das Kladogramm der Chloroviren (A) zeigt, dass die Gattung sich in die folgenden Kladen aufteilen lässt:
- NC64A-Viren (mit PBCV-1), Spezies Chlorovirus vanettense, sowie Chlorovirus illinoense
- SAG-Viren (mit ATCV-1), Spezies Chlorovirus heliozoae
- PBi-Viren (mit PBCV-A1), Spezies Chlorovirus conductrix
- NYs1-Viren (mit NYs-1), Spezies Chlorovirus newyorkense
- OSy-Viren (nur OSy-NE5)
Äußere Systematik
Die aktuelle offizielle Systematik ordnet die Gattung Chlorovirus zusammen mit einigen anderen Algen parasitierenden Riesenviren der Familie Phycodnaviridae innerhalb der Ordnung Algavirales zu.
Neuere Phylogenien könne eine gewisse Verwandtschaft allerdings nur mit den Gattungen Prasinovirus und Raphidovirus bestätigen.
Für die Klade der Phycodnaviridae vom Chlorovirus-Typ (per Vorschlag =„Prasinoviridae“) ergibt sich nach Koonin et al. (2019)[58] und Rolland et al. (2019, 2021),[59][60] ein Stammbaum, in den sich die Dishui-Lake-Phycodnaviren nach Xu (2020)[28] ebenfalls integrieren lassen:
| Chlorovirus-Typ[61] („Prasinoviridae“) |
| ||||||||||||||||||||||||||||||
Schwestergruppe dieser Klade – per Vorschlag Familie „Prasinoviridae“ – wäre dann die Gattung Raphidovirus, ebenfalls im Rang einer Familie (mit provisorischem Namen „AG_04“), zusammen bilden sie eine Klade Algavirales s. s.; andere bisherige Phycodnaviridae-Mitglieder (Gattungen Coccolithovirus, Phaeovirus) werden neuerdings zusammen mit den Gattungen bzw. vorgeschlagenen Gattungen Yaravirus, Medusavirus, „Pandoravirus“, „Mollivirus“, „Clandestinovirus“ (und anderen) verschiedenen Familien einer vorgeschlagenen Ordnung „Pandoravirales“ zugeordnet.[62][63] Ein Kladogramm findet sich unter Phycodnaviridae §Kladogramm (Zhang).
Ökologie
Chloroviren sind in Süßwasserumgebungen in allen Teilen der Welt weit verbreitet und wurden aus Süßwasserquellen in Europa, Asien, Australien sowie Nord- und Südamerika isoliert.[64][65]
Wirte
Zu den natürlichen Wirten der Chloroviren zählen verschiedene Arten einzelliger Chlorella-ähnlicher Algen, die Zoochlorellen genannt werden. Sie sind sehr spezies- und sogar stammspezifisch: Einzelne Virusspezies infizieren typischerweise nur Wirte einer bestimmten Linie (engl. strain). Diese Zoochlorellen bauen im Allgemeinen endosymbiotische Beziehungen zu größeren Protozoen (Protisten) und Wirbellosen (Invertebrata) des Süß- oder Salzwassers auf, beispielsweise:
- dem Grünen Pantoffeltierchen (Paramecium bursaria, ein Wimpertierchen – Ciliophora),
- der Grünen Hydra (Hydra viridissima alias H. viridis, Hydrozoa),[66]
- Acanthocystis turfacea (Hacrobia: Centroheliozoa, veraltet ‚Sonnentierchen‘, siehe auch Acanthocystis turfacea auf Microworld).[67]
Während ein einzelner Protist zu einem bestimmten Zeitpunkt bis zu mehrere hundert Algenzellen beherbergen kann, sind frei schwebende Algen sehr anfällig für Chloroviren, was darauf hindeutet, dass eine solche Endosymbiose eine Infektionsresistenz verleiht.[68]
Kürzlich wurde auch festgestellt, dass Chloroviren Menschen infizieren. Die Möglichkeit einer Infektionen von Mäusen wird untersucht (s. u.).[10]
Vorkommen
Die Chlorovirus-Titer variieren je nach Jahreszeit und Ort. Aufgrund der reichen genetischen Vielfalt und der hohen Spezialisierung einzelner Virusspezies sind Abweichungen in ihrer Ökologie nicht ungewöhnlich. Dies führt zu spezifischen räumlich-zeitlichen Mustern, die letztendlich vom Lebensstil und der Art des Wirts abhängen. Bisherige Übersichtsdaten zeigten zwei hervorstechende saisonale Häufigkeitsspitzen: für Chlorella variabilis NC64A-Viren im Spätherbst und für Chlorella variabilis Syngen-Viren im späten Frühling bis Mitte des Sommers, was wahrscheinlich auf die Tatsache zurückzuführen ist, dass sie den Wirt gemeinsam haben. Umgekehrt erreichten Chlorella heliozoae SAG-Viren zu verschiedenen Jahreszeiten ihren Höhepunkt und zeigten im Vergleich zu den NC64A- und Syngen-Viren im Allgemeinen eine größere Variabilität der Titer.[64]
Darüber hinaus zeigten Studien, dass Chloroviren eine gewisse Widerstandsfähigkeit gegen den winterlichen Temperaturabfall aufweisen, was durch das Vorhandensein von infektiösen Partikeln (Virionen) unter Eisschichten in einem Regenwassermanagement-Teich in Ontario, Kanada, belegt ist.[69] DeLong et al. vermuten 2016, dass Verfolgung durch kleine Krebstiere (Crustaceen) eine indirekte Rolle bei der Titerfluktuation spielen kann. Der Abbau von Protistenzellen, die den Verdauungstrakt der Krebstiere passieren, könnte zu einer Freisetzung einer großen Anzahl der einzelligen Algen führen, die – aufgrund des weggefallenen Endsymbiose-Wirts – anfällig für eine Virusinfektion werden.[68] In Konsequenz hängt die saisonale Häufigkeit von Chloroviren nicht nur von der eigenen Wirtsart ab, sondern auch von vielen anderen Mikroorganismen, dem allgemeinen Nährstoffstatus und ökologischen Rahmenbedingungen.[70]
In ihrer Gesamtheit können Chloroviren über den Phytoplanktonumsatz globale biogeochemische Zyklen beeinflussen. Bekannterweise verursacht Chlorella zusammen mit anderen Arten mikroskopischer Algen und Blaugrünbakterien (Cyanobakterien) wie Microcystis aeruginosa toxische Algenblüten, die in der nördlichen Hemisphäre (Erdhälfte) üblicherweise von Februar bis Juni dauern. Dies führt zu Sauerstoffmangel und in der Folge zum Tod größerer Organismen in den Süßwasserlebensräumen.[71][72]
Lytische (d. h. zellzerstörende) Infektion einzelliger Algen durch Chloroviren führt zum Abbruch der Algenblüten und anschließender Freisetzung des in den Algenzellen enthaltenen Kohlenstoffs, Stickstoffs und Phosphors, die verdünnt letztendlich der Nahrungskette wieder zugeführt werden.[70]
Aufbau
- Schemazeichnung eines Virions der Gattung Chlorovirus, Familie Phycodnaviridae (Querschnitt und Seitenansicht, ohne Dorn und Vertex)
Die Virionen der Gattung Chlorovirus haben eine Hülle mit ikosaedrischer oder sphärischer Geometrie und eine Symmetrie (Triangulationszahl) T=169. Der Durchmesser beträgt ca. 100–220 nm. Das Genom ist linear, in der Regel einfach vorhanden und besteht aus doppelsträngiger DNA (dsDNA), mit einer Länge von etwa 330 kb. Die dsDNA ist geschlossen mit einer Hairpin (Haarnadelstruktur) am Ende. Es gibt oft mehrere hundert ORFs (offene Leserahmen, englisch 'open reading frames').[5]
Die Gattung der Chloroviren kodiert in ihrer Gesamtheit in Summe 632 Proteinfamilien, jedes einzelne Virus hat jedoch nur 330–416 Gene, die Proteine kodieren. Chloroviren enthalten in bestimmten Abschnitten ihrer DNA-Sequenz methylierte Basen. Einige Chloroviren enthalten auch Introns und Inteine, obwohl dies innerhalb der Gattung selten ist.[66]
- Die Typenspezies Paramecium bursaria Chlorella virus 1 (PBCV-1) hat einen Durchmesser von 190 nm und eine Fünffach-Achse.[66][74] Die Verbindungsstelle seines Kopfes weist einen hervorstehenden Dorn (Spike) auf. Dies ist der erste Teil des Virions, das seinen Wirt kontaktiert.[67] Die Kryo-EM-Aufnahmen von PBCV-1 zeigen die lange schmale zylindrische Spike-Struktur an einem Scheitelpunkt (Vertex) und eine innere Membran (grün), die das Virus-Genom asymmetrisch umgibt.[2] Das äußere Kapsid bedeckt eine einzelne Membran aus einer Lipid-Doppelschicht, die aus dem endoplasmatischen Retikulum des Wirts gewonnen wird.[74] Die Genomlänge beträgt 330.611 bp, dabei werden vorhergesagt 802 Proteine kodiert. Der GC-Gehalt liegt bei 40 %.[75] Einige Kapsomere auf der äußeren Hülle haben Fasern, die vom Virusteilchen abstehen und die Anheftung an den Wirt unterstützen (vgl. Mimivirus).[66][67][76]
- Acanthocystis turfacea Chlorella virus 1 (AtCV-1, Spezies Chlorovirus heliozoae) hat eine Genomlänge von 288.047 bp, es werden vorhergesagt 860 Proteine kodiert und der GC-Gehalt beträgt 49 %.[75]
Replikationszyklus
- Die frühen Stadien einer PBCV-1-Infektion von Chlorella ähneln der Infektion von Bakterien mit Phagen der Ordnung Caudovirales.[77][Anm. 1]
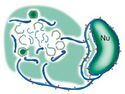
- Ein Modell der Assemblierung von PBCV-1 zu infektiösen Partikeln. Es zeigt die aus dem Zellkern (Nu) abgeleiteten Zisternen (englisch cisternae), die mit Ribosomen (rote Kügelchen) dekoriert sind. Sie dienen in den viralen Assemblierungszentren als Vorläufer (dunkelblau) für einlagige virale Membranen (hellblau), d. h. als Kapsidstrukturen.[2][Anm. 1]
- Kryo-EM-Querschnitt eines Kapsids von PBCV-1, das gerade seine DNA in die Wirtszelle freisetzt.[2][Anm. 1]
- Chlorella-Zellen und Paramecium bursaria Chlorella virus 1 (PBCV-1)[67]
Bei Paramecium bursaria chlorella virus 1 (PBCV-1), dem Prototyp des Chlorovirus, berührt zunächst der Dorn (Spike) die Zellwand des Wirts.[78] und wird dann durch Fasern unterstützt, um das Virusteilchen (Virion) am Wirt zu sichern. Die Anlagerung von PBCV-1 an seinen Rezeptor ist sehr spezifisch und schränkt den Bereich der möglichen Wirte stark ein. Virusassoziierte Enzyme ermöglichen den Abbau der Wirtszellwand und die interne Membran des Virus verschmilzt mit der Wirtsmembran. Diese Fusion ermöglicht den Transfer der Virus-DNA und von viralen Proteinen in die Wirtszelle und löst auch eine Depolarisation der Wirtsmembran aus.
Da PBCV-1 kein Gen für RNA-Polymerase besitzt, wandern seine DNA und viralen Proteine in den Zellkern, wo die Transkription 5–10 Minuten nach der Infektion beginnt. Es wird angenommen, dass diese schnelle Transkription durch ein Protein ermöglicht wird, das den Transfer der DNA in den Zellkern bewerkstelligt und durch das PBCV-a443r-Gen kodiert wird. Es ähnelt Proteinen, die am Durchschleusen durch die Kernmembran in Säugetierzellen beteiligt sind.
In dieser frühen Infektionsphase sinkt die (eigene) Transkriptionsrate des Wirts, und die Transkriptionskomponenten des Wirts werden zur Transkription der neuen viralen DNA umprogrammiert. Minuten nach der Infektion beginnt der Abbau der chromosomalen DNA des Wirts. Es wird vermutet, dass dies durch PBCV-1-kodierte und verpackte DNA-Restriktionsendonukleasen erfolgt. Durch den Abbau der chromosomalen Wirts-DNA kommt die Transkription des Wirts zum Erliegen.[79]
Die virale DNA-Replikation beginnt nach 60 bis 90 Minuten. Etwa 2–3 Stunden nach der Infektion beginnt der Zusammenbau der Virushüllen (Kapside). Dies tritt in lokalisierten Regionen des Zytoplasmas auf, wobei die Viruskapside 3–4 Stunden nach der Erstinfektion beobachtbar sind. 5–6 Stunden nach der PBCV-1-Infektion füllt sich das Zytoplasma der Wirtszelle mit infektiösen Viruspartikeln (der Nachkommenschaft). Kurz danach (6–8 Stunden nach der Infektion) setzt die lokalisierte Lyse (Auflösung) der Wirtszelle diese Nachkommen frei. Aus jeder infizierten Zelle werden ca. 1000 Viruspartikel freigesetzt.[67]
Infektion beim Menschen
Kürzlich wurde Chlorovirus ATCV-1-DNA in menschlichen Pharynx-Proben gefunden. Bis dato war nicht bekannt, dass das Chlorovirus Menschen infizieren könnte, daher ist das Wissen über Infektionen bei Menschen noch sehr begrenzt. Infizierte Personen hatten ein verzögertes Gedächtnis und verringerte Aufmerksamkeit sowie eine verminderte visuelle Verarbeitung und visuelle Motorik. Dies führte insgesamt zu einem Rückgang der Fähigkeit, Aufgaben basierend auf Sehen und räumlichem Denken durchzuführen.[10]
Die Studien zur Infektion von Mäusen mit ATCV-1 zeigten bei infizierten Tieren Veränderungen im Cdk5-Signalweg,[80] der das Lernen und die Gedächtnisbildung unterstützt, sowie Veränderungen der Genexpression im Dopamin-Signalweg.[10] Infizierte Mäuse erwiesen sich zudem als weniger sozial und interagierten weniger mit neu eingeführten Begleitmäusen als die gesunde Kontrollgruppe. Sie verbrachten längere Zeit in einem lichtexponierten Bereich der Testkammer, wohingegen die Kontrollmäuse wie üblich die dunkle Seite bevorzugten und das Licht mieden. Dies deutet auf eine Abnahme der Angstzustände bei einer ATCV-1-Infektion hin. Die Testmäuse waren auch weniger in der Lage, ein Objekt zu erkennen, das von seiner vorherigen Position verschoben worden war, was eine Abnahme des räumlichen Referenzspeichers belegt.[11] Wie beim Menschen nimmt die räumliche Aufgabenfähigkeit des Sehzentrums ab.[10][11]
Anmerkungen
- ↑ a b c d e f g h Das Material wurde von dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist.
Literatur
- Dickson Kinyanyi, George Obiero, Peris W Amwayi, Stephen Mwaniki, Mark Wamalwa: In silico structural and functional prediction of African swine fever virus protein-B263R reveals features of a TATA-binding protein. In: PeerJ. Band 6, Nr. 4, 2018, Artikel e4396, doi:10.7717/peerj.4396, S. 13, insbesondere Figur. 7.
- Weijia Zhang, Jinglie Zhou, Taigang Liu, Yongxin Yu, Yingjie Pan, Shuling Yan, Yongjie Wang: Four novel algal virus genomes discovered from Yellowstone Lake metagenomes. In: Scientific Reports. Band 5, 2015, Artikel Nr. 15131, doi:10.1038/srep15131, Abstract.
Weblinks
Einzelnachweise
- ↑ a b c d e ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ↑ a b c d e f g h i j k l m n o p q r s t u v w x y z aa ab ac ad ae af ag ah ai aj ak al am an ao James L. Van Etten, Irina V. Agarkova, David D. Dunigan: Chloroviruses. In: Viruses. Band 12, Nr. 1, Reihe: Viruses Ten-Year Anniversary. 13. Dezember 2019, Band 20, doi:10.3390/v12010020.
- ↑ N. Yutin, Y. I. Wolf, Eugene V. Koonin: Origin of giant viruses from smaller DNA viruses not from a fourth domain of cellular life. In: Virology. 466–467. Jahrgang, Oktober 2014, S. 38–52, doi:10.1016/j.virol.2014.06.032, PMID 25042053, PMC 4325995 (freier Volltext) – (englisch, elsevier.com).
- ↑ David D. Dunigan, L. A. Fitzgerald, James L. Van Etten: Phycodnaviruses: a peek at genetic diversity. In: Virus Research. 117. Jahrgang, Nr. 1, April 2006, S. 119–32, doi:10.1016/j.virusres.2006.01.024, PMID 16516998 (englisch).
- ↑ a b ViralZone. ExPASy, abgerufen am 23. Dezember 2018 (englisch).
- ↑ ICTV: Virus Taxonomy. Abgerufen am 22. Dezember 2015 (englisch).
- ↑ ICTV Master Species List 2018a v1 ( des vom 14. März 2019 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis., ICTV MSL including all taxa updates since the 2017 release.
- ↑ Russel H. Meints, James L. Van Etten, Daniel Kuczmarski, Kit Lee, Barbara Ang: Viral infection of the symbiotic chlorella-like alga present in Hydra viridis. In: Virology. 113. Jahrgang, Nr. 2, September 1981, S. 698–703, PMID 18635088 (englisch).
- ↑ Ryo Hoshina, Mayumi Shimizu, Yoichi Makino, Yoshihiro Haruyama, Shin-ichiro Ueda, Yutaka Kato, Masahiro Kasahara, Bun-ichiro Ono, Nobutaka Imamura: Isolation and characterization of a virus (CvV-BW1) that infects symbiotic algae of Paramecium bursaria in Lake Biwa, Japan. In: Virology Journal. 7. Jahrgang, 13. September 2010, ISSN 1743-422X, S. 222, doi:10.1186/1743-422X-7-222, PMID 20831832, PMC 2949830 (freier Volltext) – (englisch).
- ↑ a b c d e f R. H. Yolken, L. Jones-Brando, D. D. Dunigan, G. Kannan, F. Dickerson, E. Severance, S. Sabunciyan, C. C. Talbot, E. Prandovszky, J. R. Gurnon, I. V. Agarkova, F. Leister, K. L. Gressitt, O. Chen, B. Deuber, F. Ma, M. V. Pletnikov, J. L. Van Etten: Chlorovirus ATCV-1 is part of the human oropharyngeal virome and is associated with changes in cognitive functions in humans and mice. In: Proceedings of the National Academy of Sciences of the United States of America. 111. Jahrgang, Nr. 45, November 2014, S. 16106–11, doi:10.1073/pnas.1418895111, PMID 25349393, PMC 4234575 (freier Volltext) – (englisch, pnas.org).
- ↑ a b c Marilyn S. Petro, Irina V. Agarkova, Thomas M. Petro: Effect of Chlorovirus ATCV-1 infection on behavior of C57Bl/6 mice. In: Journal of Neuroimmunology. 297. Jahrgang, August 2016, S. 46–55, doi:10.1016/j.jneuroim.2016.05.009, PMID 27397075 (englisch).
- ↑ ICTV: Taxonomy Browser.
- ↑ ICTV: Virus Metadata Resource (VMR).
- ↑ a b c d e f Hao Chen, Weijia Zhang, Xiefei Li, Yingjie Pan, Shuling Yan, Yongjie Wang: The genome of a prasinoviruses-related freshwater virus reveals unusual diversity of phycodnaviruses. In: BMC Genomics. Dezember 2018, online 15. Januar 2018; doi:10.1186/s12864-018-4432-4.
- ↑ a b c d e f g h Guillaume Blanc, Garry Duncan, Irina Agarkova, Mark Borodovsky, James Gurnon, Alan Kuo, Erika Lindquist, Susan Lucas, Jasmyn Pangilinan, Juergen Polle, Asaf Salamov, Astrid Terry, Takashi Yamada, David D. Dunigan, Igor V. Grigoriev, Jean-Michel Claverie, James L. Van Etten: The Chlorella variabilis NC64A genome reveals adaptation to photosymbiosis, coevolution with viruses, and cryptic sex. In: Plant Cell, 22, 2010, S. 2943–2955, doi:10.1105/tpc.110.076406
- ↑ NCBI: Paramecium bursaria Chlorella virus FR483 (Species)
- ↑ a b c d e f g Timo Greiner, Anna Moroni, James L. Van Etten, Gerhard Thiel: Genes for Membrane Transport Proteins: Not So Rare in Viruses. In: MDPI Viruses. Band 10, Nr. 9, Special Issue Algae Virus. 26. August 2018, Band 456, doi:10.3390/v10090456.
- ↑ William H. Wilson, Ilana C. Gilg, Mohammad Moniruzzaman, Erin K. Field, Sergey Koren, Gary R. LeCleir, Joaquín Martínez Martínez, Nicole J. Poulton, Brandon K. Swan, Ramunas Stepanauskas, Steven W. Wilhelm: Genomic exploration of individual giant ocean viruses. In: ISME Journal. Band 11, Nr. 8, August 2017, S. 1736–1745; doi:10.1038/ismej.2017.61, PMC 5520044 (freier Volltext), PMID 28498373 (englisch). Hier offenbar irrtümlich als Acanthamoeba turfacea Chlorella virus (ATCV) bezeichnet.
- ↑ NCBI: Acanthocystis turfacea Chlorella virus Br0604L (Species)
- ↑ NCBI: Acanthocystis turfacea Chlorella virus Can0610SP (Species)
- ↑ NCBI: Acanthocystis turfacea Chlorella virus Canal-1 (Species)
- ↑ NCBI: Acanthocystis turfacea Chlorella virus GM0701.1 (Species)
- ↑ NCBI: Acanthocystis turfacea Chlorella virus MO0605SPH (Species)
- ↑ NCBI: Acanthocystis turfacea Chlorella virus MN0810.1 (Species)
- ↑ NCBI: Acanthocystis turfacea Chlorella virus NTS-1 (Species), Acanthocystis turfacea Chlorella virus NTS1 (Species)
- ↑ NCBI: Acanthocystis turfacea Chlorella virus NE-JV-2 (Species)
- ↑ NCBI: Acanthocystis turfacea Chlorella virus NE-JV-3 (Species)
- ↑ a b c Shengzhong Xu, Liang Zhou, Xiaosha Liang, Yifan Zhou, Hao Chen, Shuling Yan, Yongjie Wang; Julie K. Pfeiffer (Hrsg.): Novel Cell-Virus-Virophage Tripartite Infection Systems Discovered in the Freshwater Lake Dishui Lake in Shanghai, China. In: Journal of Virology, 18. Mai 2020; doi:10.1128/JVI.00149-20, PMID 32188734 (englisch).
- ↑ NCBI: Acanthocystis turfacea Chlorella virus OR0704.3 (Species)
- ↑ NCBI: Acanthocystis turfacea Chlorella virus TN603.4.2 (Species)
- ↑ NCBI: Acanthocystis turfacea Chlorella virus TN603.4.2 (Species)
- ↑ NCBI: Acanthocystis turfacea Chlorella virus WI0606 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus AN69C (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus AR158 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus CZ-2 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus AP110A (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus Can18-4 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus CVB-1 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus CVG-1 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus CviKI (Species)
- ↑ a b Julien Andreani, Jacques Y. B. Khalil, Emeline Baptiste, Issam Hasni, Caroline Michelle, Didier Raoult, Anthony Levasseur, Bernard La Scola: Orpheovirus IHUMI-LCC2: A New Virus among the Giant Viruses. In: Front. Microbiol., 22. Januar 2018; doi:10.3389/fmicb.2017.02643
- ↑ NCBI: Paramecium bursaria Chlorella virus CVM-1 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus CVR-1 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus CvsA1 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus CZ-2 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus Fr5L (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus IL-5-2s1 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus KS1B (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus MA-1D (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus MA-1E (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus NE-JV-1 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus NE-JV-4 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus NW665.2 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus NY-2B (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus NYs1 (Species)
- ↑ NCBI: Paramecium bursaria Chlorella virus OR0704.2.2 (Species)
- ↑ NCBI: Only Syngen Nebraska virus 5 (Species)
- ↑ Eugene V. Koonin, Natalya Yutin: Evolution of the Large Nucleocytoplasmatic DNA Viruses of Eukaryotes and Convergent Origins of Viral Gigantism. In: Advances in Virus research, Band 103, AP 21. Januar 2019, doi:10.1016/bs.aivir.2018.09.002, S. 167–202. In Fig. 4 ist phycodnaviruses wohl im engeren Sinne (s. s.) zu verstehen und dürfte der Chlorovirus/Prasinovirus/‚YLPV‘-Gruppe bei Schulz et al. (2018) entsprechen.
- ↑ Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, Said Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Felipe Assis, Fábio Dornas, Jacques Yaacoub Bou Khalil, Isabelle Pagnier, Christelle Desnues, Anthony Levasseur, Philippe Colson, Jônatas Abrahão, Bernard La Scola: Discovery and Further Studies on Giant Viruses at the IHU Mediterranee Infection That Modified the Perception of the Virosphere. In: MDPI: Viruses, Band 11, Nr. 4, März/April 2019, pii: E312; doi:10.3390/v11040312, PMC 6520786 (freier Volltext), PMID 30935049, MDPI (englisch); siehe Fig. 2a.
- ↑ Clara Rolland, Julien Andreani, Dehia Sahmi-Bounsiar, Mart Krupovic, Bernard La Scola, Anthony Levasseur: Clandestinovirus: A Giant Virus With Chromatin Proteins and a Potential to Manipulate the Cell Cycle of Its Host Vermamoeba vermiformis. In: Frontiers in Microbiology, Band 12, 10. August 2021, Nr. 715608; doi:10.3389/fmicb.2021.715608, PMID 34447361, PMC 8383183 (freier Volltext) (englisch).
- ↑ Fumito Maruyama, Shoko Ueki: Evolution and Phylogeny of Large DNA Viruses, Mimiviridae and Phycodnaviridae Including Newly Characterized Heterosigma akashiwo Virus. In: Frontiers in Microbiology. 7. Jahrgang, 30. November 2016, ResearchGate:311160794, S. 1942, doi:10.3389/fmicb.2016.01942, PMID 27965659, PMC 5127864 (freier Volltext) – (englisch).
- ↑ Frank O. Aylward, Mohammad Moniruzzaman, Anh D. Ha, Eugene V. Koonin: A phylogenomic framework for charting the diversity and evolution of giant viruses. In: PLOS Biology, 27. Oktober 2021; doi:10.1371/journal.pbio.3001430 (englisch).
- ↑ Ruixuan Zhang, Masaharu Takemura, Kazuyoshi Murata, Hiroyuki Ogata: “Mamonoviridae”, a proposed new family of the phylum Nucleocytoviricota. In: Archives of Virology. Band 168, Nr. 80, 5. Februar 2023; doi:10.1007/s00705-022-05633-1, PMID 36740641 (englisch).
- ↑ a b C.F. Quispe, O. Sonderman, A. Seng, B. Rasmussen, G. Weber, C. Mueller, D. D. Dunigan, J. L. Van Etten: Three-year survey of abundance, prevalence and genetic diversity of chlorovirus populations in a small urban lake. In: Archives of Virology. Band 161. Jahrgang, Nr. 7, Juli 2016, S. 1839–1847, PMID 27068168 (englisch).
- ↑ S. M. Short: The ecology of viruses that infect eukaryotic algae. In: Environmental Microbiology. 14. Jahrgang, Nr. 9, September 2012, S. 2253–2271, doi:10.1111/j.1462-2920.2012.02706.x, PMID 22360532 (englisch).
- ↑ a b c d James L. Van Etten, David D. Dunigan, Richard C. Condit: Giant Chloroviruses: Five Easy Questions. In: PLOS Pathogens. 12. Jahrgang, Nr. 8, 18. August 2016, S. e1005751, doi:10.1371/journal.ppat.1005751, PMID 27536965, PMC 4990331 (freier Volltext) – (englisch).
- ↑ a b c d e James L. Van Etten, David D. Dunigan: Chloroviruses: not your everyday plant virus. In: Trends in Plant Science. 17. Jahrgang, Nr. 1, Januar 2012, S. 1–8, doi:10.1016/j.tplants.2011.10.005, PMID 22100667, PMC 3259250 (freier Volltext) – (englisch).
- ↑ a b J. P. DeLong, Z. Al-Ameeli, G. Duncan, James L. Van Etten, David D. Dunigan: Predators catalyze an increase in chloroviruses by foraging on the symbiotic hosts of zoochlorellae. In: Proceedings of the National Academy of Sciences of the United States of America. 113. Jahrgang, Nr. 48, November 2016, S. 13780–13784, doi:10.1073/pnas.1613843113, PMID 27821770, PMC 5137705 (freier Volltext) – (englisch, pnas.org).
- ↑ A. M. Long, S. M. Short: Seasonal determinations of algal virus decay rates reveal overwintering in a temperate freshwater pond. In: The ISME Journal. 10. Jahrgang, Nr. 7, Juli 2016, S. 1602–1612, doi:10.1038/ismej.2015.240, PMID 26943625, PMC 4918447 (freier Volltext) – (englisch, nature.com).
- ↑ a b GM Yanai: Transcription analysis of the chlorovirus Paramecium bursaria chlorella virus-1 (PhD). University of Nebraska at Lincoln, 2009.
- ↑ H. Song, M. Lavoie, X. Fan, H. Tan, G. Liu, P. Xu, Z. Fu, H. W. Paerl, H. Qian: Allelopathic interactions of linoleic acid and nitric oxide increase the competitive ability of Microcystis aeruginosa. In: The ISME Journal. 11. Jahrgang, Nr. 8, August 2017, S. 1865–1876, doi:10.1038/ismej.2017.45, PMID 28398349, PMC 5520033 (freier Volltext) – (englisch).
- ↑ M Rieper: Investigations on the relationships between algal blooms and bacterial populations in the Schlei Fjord (western Baltic Sea). In: Helgoländer wissenschaftliche Meeresuntersuchungen. 28. Jahrgang, Nr. 1, 1. März 1976, S. 1–18, doi:10.1007/BF01610792 (englisch).
- ↑ Chuan Xiao, Matthias G. Fischer, Duer M. Bolotaulo, Nancy Ulloa-Rondeau, Gustavo A. Avila, Curtis A. Suttle: Cryo-EM reconstruction of the Cafeteria roenbergensis virus capsid suggests novel assembly pathway for giant viruses. In: Nature Scientific Reports. Band 7, Nr. 5484, 14. Juli 2017; doi:10.1038/s41598-017-05824-w.
- ↑ a b Cristian F. Quispe, Ahmed Esmael, Olivia Sonderman, Michelle McQuinn, Irina Agarkova, Mohammed Battah, Garry A. Duncan, David D. Dunigan, Timothy P. L. Smith, Cristina De Castro, Immacolata Speciale, Fangrui Ma, James L. Van Etten: Characterization of a new chlorovirus type with permissive and non-permissive features on phylogenetically related algal strains. In: Virology. 500. Jahrgang, Januar 2017, S. 103–113, doi:10.1016/j.virol.2016.10.013, PMID 27816636, PMC 5127778 (freier Volltext) – (englisch).
- ↑ a b David M. Needham, Alexandra Z. Worden et al.:A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators. ( des vom 26. September 2019 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. In: PNAS, 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424, hier: Supplement 1 (xlsx) (Seite nicht mehr abrufbar, festgestellt im Oktober 2022. Suche in Webarchiven) Info: Der Link wurde automatisch als defekt markiert. Bitte prüfe den Link gemäß Anleitung und entferne dann diesen Hinweis.
- ↑ Jean-Michel Claverie, Chantal Abergel: „Mimiviridae“: An Expanding Family of Highly Diverse Large dsDNA Viruses Infecting a Wide Phylogenetic Range of Aquatic Eukaryotes. In: Viruses. 18. September 2018, Band 109, S. 506, doi:10.3390/v10090506, PMC 6163669 (freier Volltext), PMID 30231528, Tabelle 2.
- ↑ Elad Milrot, Eyal Shimoni, Tali Dadosh, Katya Rechav, Tamar Unger, James L. Van Etten, Abraham Minsky: Structural studies demonstrating a bacteriophage-like replication cycle of the eukaryote-infecting Paramecium bursaria chlorella virus-1. In: PLOS Pathogens. Version 2, 29. April 2017, doi:10.1371/journal.ppat.1006562.
- ↑ X. Zhang, Y. Xiang, D. D. Dunigan, T. Klose, P. R. Chipman, J. L. Van Etten, MG Rossmann: Three dimensional structure and function of the Paramecium bursaria chlorella virus capsid. In: Proceedings of the National Academy of Sciences of the United States of America. 2011, Band 108, S. 14837–14842, doi:10.1073/pnas.1107847108, PMID 21873222.
- ↑ G. Blanc, M. Mozar, I. V. Agarkova, J. R. Gurnon, G. Yanai Balser, J. M. Rowe, Y. Xia, J. J. Riethoven, D. D. Dunigan, J. L. Van Etten: Deep RNA sequencing reveals hidden features and dynamics of early gene transcription in Paramecium bursaria chlorella virus 1. In: PLoS ONE. 2014, Band 9, Artikel 90989, doi:10.1371/journal.pone.0090989, PMID 24608750.
- ↑ Siehe auch Jerry H.-C. Wang
Auf dieser Seite verwendete Medien
Autor/Urheber: Provided by James L. Van Etten, Irina V. Agarkova, David D. Dunigan. Created by Cherrier et al., Lizenz: CC BY-SA 4.0
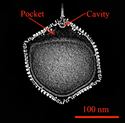
CryoEM structure of PBCV-1: Central cross-section of the cryo-EM density. A pocket is located between the virus internal membrane and the unique vertex and a cavity is located at the bottom of the spike structure.
Autor/Urheber: Provided by James L. Van Etten, Irina V. Agarkova, David D. Dunigan. Created by Blanc et al., Lizenz: CC BY-SA 4.0
Mapping of the PBCV-1 transcriptome at various times post infection. PBCV-1 genes on the forward and reverse strands are depicted by red and blue boxes, respectively. The green curves in the interior concentric circles represent the normalized read coverage for each time point (7, 14, 20, 40, and 60 min p.i.) of the experiment in logarithmic scale (base 10). Note: the PBCV-1 genome is a linear molecule with inverted repeats and closed hairpin ends that is depicted as a circle. The two ends are at the 12 o’clock position.
Autor/Urheber: Provided by James L. Van Etten, Irina V. Agarkova, David D. Dunigan. Created by: Milrot et al., Lizenz: CC BY-SA 4.0
Chlorella cells and chlorovirus Paramecium bursaria chlorella virus-1 (PBCV-1): A model depicting PBCV-1 assembly into infectious particles including generation of nuclei- derived cisternae decorated with ribosomes (red spheres), which serve as precursors (dark blue) for single bilayer viral membranes (light blue) in the viral assembly centers. Note, the membrane serves as the template for the capsid structures to form virus particles.
Autor/Urheber: Elad Milrot, Eyal Shimoni, Tali Dadosh, Katya Rechav, Tamar Unger, James L. Van Etten, Abraham Minsky, Lizenz: CC BY-SA 4.0
A model depicting early stages of PBCV-1 infection and highlighting the similarity between PBCV-1 infection and that of bacteriophages. A. The main components involved in PBCV-1 infection, including the PBCV-1 icosahedral capsid (yellow), internal membrane (dark blue), genome (gray) and spike (magenta) on the left side of the panel, as well as the cell wall (brown), cellular membrane (dark blue), thylakoid stacks (green), nuclear membrane (light blue) and nucleus (gray) of the host on the right side. B-D. Spike-mediated perforation of the host cell wall, protrusions of the viral and cellular internal membranes and their subsequent fusion into a membrane tunnel. D, E. Internalization of PBCV-1 genome into the host cytoplasm through a membrane tunnel, accompanied by the perforation of the photosynthetic thylakoid stacks and apparently promoted by viral DNA condensation. Significantly, perforation of the host cell wall, generation of a membrane portal and genome condensation represent crucial infection stages of bacteriophages (see text for details).
Autor/Urheber: Provided by James L. Van Etten, Irina V. Agarkova, David D. Dunigan, Lizenz: CC BY-SA 4.0
Chlorella cells and chlorovirus: Paramecium bursaria chlorella virus-1 (PBCV-1): Cross-section of a five-fold averaged cryo-EM of PBCV-1 as the virus is getting ready to release its DNA into the host cell.
Autor/Urheber: Chuan Xiao, Matthias G. Fischer, Duer M. Bolotaulo, Nancy Ulloa-Rondeau, Gustavo A. Avila, Curtis A. Suttle, Lizenz: CC BY-SA 4.0
Zentraler Querschnitt der Kryo-EM-Darstellung von CroV, überlagert mit der von PBCV-1. CroV hat nicht nur einen größeren Durchmesser der Virionen (300 nm vs. 180 nm), seine Kapsidschicht ist auch dicker (10,5 nm vs. 7,5 nm)
Autor/Urheber: Provided by James L. Van Etten, Irina V. Agarkova, David D. Dunigan. Created by Quispe, Jeanniard et al., Lizenz: CC BY-SA 4.0
Phylogenies of chloroviruses and algal hosts. (A) Phylogenetic tree shows the evolutionary relationships between 47 chloroviruses concatenated amino acid sequences (7762 gap-free sites). The Maximum Likelihood tree was constructed using the MEGA 6.0 software with the Maximum Likelihood algorithm and default setting. The bar length represents 0.2 substitutions per amino acid site. A recently characterized Osy chlorovirus species is indicated in red and resides between the separate phylogenetic subclades of NC64A viruses; one subclade contains PBCV-1 while the other subclade contains chlorovirus NY-2A. Both subclades share almost perfect gene colinearity and they replicate in the same host. Branch support was estimated from 1000 bootstrap replicates. Four Ostreococcus virus sequences served as outgroups to root the tree. (B) Maximum Likelihood tree of three algal hosts based on 18S RNA alignment (2266 gap-free sites). The phylogenetic tree was computed using the GTR + G + I substitution model. All interior branches received maximal support (100%). Parachlorella species served as the outgroup.
Autor/Urheber: James L. Van Etten, David D. Dunigan, Lizenz: CC BY-SA 4.0
(A) Paramecium bursaria and its symbiotic Chlorella cells. (B) Plaques formed by PBCV-1 on a lawn of Chlorella variabilis. (C) Five-fold averaged cryo-electron micrograph of PBCV-1 reveals a long narrow cylindrical spike structure at one vertex and fibers extending from one unique capsomer per trisymmetron. (D) PBCV-1 attached to the cell wall as viewed by the quick-freeze, deep etch procedure. Note the virions attached to the wall by fibers. (E) Surface view of the PBCV-1 spike structure and fibers. (G) Attachment of PBCV-1 to the algal wall and digestion of the wall at the point of attachment. This occurs within 1–3 minutes postinfection (PI) (H) Virion particles assemble in defined areas in the cytoplasm named virus assembly centers at ~4 hour PI. Note that both DNA containing (dark centers) and empty capsids. (J) Localized lysis of cell plasma membrane and cell wall and release of progeny viruses at ~8 hours PI.
Autor/Urheber: Provided by James L. Van Etten, Irina V. Agarkova, David D. Dunigan, Lizenz: CC BY-SA 4.0
Chlorella cells and chlorovirus: Paramecium bursaria chlorella virus-1 (PBCV-1): Cross section of a five-fold averaged cryo-EM image of PBCV-1 reveals a long narrow cylindrical spike structure at one vertex and the viral internal membrane (green) surrounding the viral genome asymmetrically.
Autor/Urheber: ViralZone, SIB Swiss Institute of Bioinformatics, Philippe Le Mercier et al., Lizenz: CC BY 4.0
Schemazeichnung eines Virions der Phycodnaviridae (nicht Coccolithovirus)
Autor/Urheber: Provided by James L. Van Etten, Irina V. Agarkova, David D. Dunigan. Created by Kang et al., Lizenz: CC BY-SA 4.0
Proposed replication cycle of PBCV-1. The virus binds to the surface of the alga and opens a portal to release the viral DNA and associated proteins; the viral DNA moves to the nucleus where early gene transcription begins at 5 to 10 min p.i. Early mRNAs move to the cytoplasm for translation, and at least some early proteins presumably return to the viral genome located at or near to the nucleus to initiate viral DNA replication, which begins ~60 p.i., followed by late gene transcription. Late mRNAs move to the cytoplasm for translation and many of these late proteins are targeted to the virus assembly centers, where virus capsids are formed and DNA is packaged. Mature infectious virus particlles appear in the cytoplasm of the cell ~45 min prior to virus release. The chlorella cell membrane and wall lyses, and infectious PBCV-1 progeny viruses are released at 6 to 8 h p.i. (→) Known events; (---->) hypothesized events.


![Genomkarte von PBCV-1.[2][Anm. 1]](http://upload.wikimedia.org/wikipedia/commons/thumb/7/7e/Viruses-12-00020-g005_PBCV-1_Genome_Map_%28frameless%29.png/182px-Viruses-12-00020-g005_PBCV-1_Genome_Map_%28frameless%29.png)
![Querschnitt der Kryo-EM-Darstellung des Cafeteriavirus CroV, überlagert mit der des Chlorovirus PBCV-1. CroV hat einen größeren Durchmesser der Virionen (300 nm vs. 180 nm) und seine Kapsidschicht ist dicker (10,5 nm vs. 7,5 nm). Umrechnung 10 Å = 1 nm[73][Anm. 1]](http://upload.wikimedia.org/wikipedia/commons/thumb/2/27/41598_2017_5824_Fig3_HTML_%28D%29_virions.jpg/200px-41598_2017_5824_Fig3_HTML_%28D%29_virions.jpg)
![Die frühen Stadien einer PBCV-1-Infektion von Chlorella ähneln der Infektion von Bakterien mit Phagen der Ordnung Caudovirales.[77][Anm. 1]](http://upload.wikimedia.org/wikipedia/commons/thumb/9/9f/Ppat.1006562.g008_PBCV-1_and_bacteriophage_infecting.tif/lossy-page1-200px-Ppat.1006562.g008_PBCV-1_and_bacteriophage_infecting.tif.jpg)
![Vorgeschlagener Replikationszyklus von PBCV-1.[2][Anm. 1]](http://upload.wikimedia.org/wikipedia/commons/thumb/6/6c/Viruses-12-00020-g008_PBCV-1_replicaton_cycle_%28frameless%29.png/189px-Viruses-12-00020-g008_PBCV-1_replicaton_cycle_%28frameless%29.png)
![Ein Modell der Assemblierung von PBCV-1 zu infektiösen Partikeln. Es zeigt die aus dem Zellkern (Nu) abgeleiteten Zisternen (englisch cisternae), die mit Ribosomen (rote Kügelchen) dekoriert sind. Sie dienen in den viralen Assemblierungszentren als Vorläufer (dunkelblau) für einlagige virale Membranen (hellblau), d. h. als Kapsidstrukturen.[2][Anm. 1]](http://upload.wikimedia.org/wikipedia/commons/thumb/e/e0/Viruses-12-00020-g001_%28H%29_assembly_schema.png/200px-Viruses-12-00020-g001_%28H%29_assembly_schema.png)
![Kryo-EM-Querschnitt eines Kapsids von PBCV-1. Zwischen der inneren Membran und dem einzigen Scheitelpunkt (Vertex) befindet sich eine Tasche und am Boden des Spikes ein Hohlraum.[2][Anm. 1]](http://upload.wikimedia.org/wikipedia/commons/thumb/6/64/Viruses-12-00020-g004_PBCV-1_CryoEM_%28B%29_anotated.png/183px-Viruses-12-00020-g004_PBCV-1_CryoEM_%28B%29_anotated.png)
![Kryo-EM-Querschnitt eines Kapsids von PBCV-1, das gerade seine DNA in die Wirtszelle freisetzt.[2][Anm. 1]](http://upload.wikimedia.org/wikipedia/commons/thumb/0/0d/Viruses-12-00020-g001_%28E%29_PBCV-1.png/175px-Viruses-12-00020-g001_%28E%29_PBCV-1.png)
![Chlorella-Zellen und Paramecium bursaria Chlorella virus 1 (PBCV-1)[67]](http://upload.wikimedia.org/wikipedia/commons/thumb/6/63/Chlorella_cells_and_chlorovirus_Paramecium_bursaria_chlorella_virus_%28PBCV-1%29_%28ABCDEGHJ%29_frameless.png/200px-Chlorella_cells_and_chlorovirus_Paramecium_bursaria_chlorella_virus_%28PBCV-1%29_%28ABCDEGHJ%29_frameless.png)