Atribacterota
| Atribacterota | ||||||
|---|---|---|---|---|---|---|
 | ||||||
| Systematik | ||||||
| ||||||
| Wissenschaftlicher Name | ||||||
| Atribacterota | ||||||
| Katayama et al. 2021[2] |

Atribacterota ist ein Phylum (Abteilung) thermophiler (wärmeliebender) Bakterien,[3][4][5] die in anoxischen (sauerstofffreien), methanreichen Sedimenten häufig vorkommen. Frühere provisorische Bezeichnungen sind „Candidatus Caldatribacteriota“,[1][6] „Candidatus Atribacteria“ (steht jetzt für die Typusklasse),[4] Candidate division JS1[4] und Candidate division OP9 (Obsidian Pool 9)[4] (jeweils im weiteren Sinn verstanden).
Die Atribacterota sind weltweit verbreitet (kosmopolitisch) und wurden einigen Fällen in anaeroben Meeressedimenten, Geothermalquellen und Ölvorkommen reichlich nachgewiesen. Die Typusart Atribacter laminatus und Metagenomdaten der Kandidatengattung Ca. Caldatribacterium sowie 16S-rRNA-Gen-Phylotypen von Mitgliedskandidaten des Phylums stammen aus einer Vielzahl von Umgebungen, darunter oberirdische und untermeerische geothermische Systeme, Erdöllagerstätten, anaerobe Fermenter und Abwasseraufbereitungsanlagen.[7]
Nach Genanalysen zeigen die Atribacterota Genexpressionsmuster, die auf einen fermentativen, acetogenen Stoffwechsel hindeuten. Diese Ergebnisse lassen auf einen heterotrophen Stoffwechsel schließen, bei dem Gärungsprodukte wie Acetat, Ethanol und Kohlendioxid entstehen. In der mikrobiellen Gemeinschaft des Sediments können von diesen Produkten wiederum Methanogene leben, was das häufige Vorkommen von Atribacterota in methanreichen anoxischen Sedimenten erklärt.[8][9]
Dank ihrer Eigenschaften sind die Atribacterota in der Lage, katabolische und anabolische Funktionen auszuführen, die für die Reproduktion der Zellen notwendig sind, selbst wenn die Energieversorgung aufgrund des Mangels an gelöstem Sauerstoff in Meeres-, Süß- oder Grundwasser eingeschränkt ist.[10]
Systematik
Nach phylogenetischen Analysen scheinen die Atribacterota mit mehreren thermophilen Phyla innerhalb der Terrabacteria[11] verwandt zu sein oder könnten basal (an der Basis) innerhalb der Gracilicutes stehen.[12]
Der nachstehenden inneren Systematik liegen folgende Quellen zugrunde (Stand 27. Juli 2022):
- L – List of Prokaryotic names with Standing in Nomenclature (LPSN)[3]
- N – National Center for Biotechnology Information (NCBI),[4] visualisiert durch Lifemap
- G – Genome Taxonomy Database (GTDB),[5] visualisiert durch AnnoTree[13]
Anmerkungen:
- Die Gattung Caldatribacterium ist nach LPSN und NCBI ohne Zuordnung (incertae sedis), wird aber in der GTDB einer Familie Caldatribacteriaceae innerhalb der Ordnung Atribacterales zugeordnet, es gibt keine eigene Ordnung „Caldatribacteriales“ (d. h. eine solche ist ein Synonym von Atribacterales).
- Nach der GTDB werden die Kladen OP9 und JS1 (im engeren Sinn verstanden) als die beiden Klassen innerhalb des gemeinsamen Phylums Atribacterota aufgefasst. Im weiteren Sinn sind Candidate division OP9 und JS1 identisch mit dem gesamten Phylum (Phylum alias lateinisch divisio, englisch division, deutsch ‚Abteilung‘).
- OneZoom verortet abweichend die Kladen OP9 und JS1 als Kandidatenphyla an verschiedenen Stellen des bakteriellen Stammbaums.[14][15]
- Da die Bezeichnung Atribacteria früher für das ganze Phylum verwendet wurde, haben auch Stämme der GTDB-Klasse JS1 bei NCBI oft provisorische Speziesnamen mit dieser Bezeichnung, obwohl sie nicht zur Klasse Atribacteria gehören.
- Vorschläge (etwa aus der Metagenomik) mit vorläufigen Bezeichnungen sind nicht komplett, sondern nur beispielsweise wiedergegeben.
Phylum Atribacterota Katayama et al. 2021,[2] veraltet auch
- „Candidatus Caldatribacteriota“ Hahn et al. 2020 (L,N,G),[1][6]
- „Candidatus Atribacteria“ Dodsworth et al. 2013 (steht jetzt für die Typusklasse),[4]
- Candidate division OP9 (Obsidian Pool 9) im weiteren Sinn als Phylum[4]
- Candidate division JS1 im weiteren Sinn als Phylum[4]
- OP9/JS1 als beide Teilkladen umfassendes Taxon[8]
- „Candidatus Atribacteria“ Dodsworth et al. 2013 (steht jetzt für die Typusklasse),[4]
- Klasse Atribacteria Katayama et al. 2021 (L,N,G),[14] veraltet Candidate division OP9 (Obsidian Pool 9) im Rang eines eigenständigen Phylums[14]
- Ordnung Atribacterales Katayama et al. 2021 (L,N,G), synonym mit „Caldatribacteriales“
- Familie Atribacteraceae Katayama et al. 2021 (L,N,G)
- Gattung Atribacter Katayama et al. 2021[16] (L,N,G)
- Spezies Atribacter laminatus Katayama et al. 2021[1] mit Stamm RT761 alias DSM 105538 oder NBRC 112890 (L,N,G)
- Spezies Atribacter sp002069605 (G) alias Ca. Atribacteria bacterium ADurb.Bin276 (N) mit ADurb.Bin276
- Spezies Atribacter sp003542295 (G) mit Stamm UBA11188
- Spezies Atribacter sp012517485 (G) mit MAG AS27yjCOA_181[17]
- Spezies Atribacter sp012517635 (G) mit MAG AS27yjCOA_164[18]
- Spezies Atribacter sp018056205 (G) mit MAG Gw_SlDig_bin_239[19]
- Gattung SKXZ01 (G)
- Spezies SKXZ01 sp003561145 (G) mit MAG CSSed11_91
- Spezies SKXZ01 sp007128145 (G) mit MAG CSSed162cmB_266
- Gattung Atribacter Katayama et al. 2021[16] (L,N,G)
- Familie Caldatribacteriaceae (G)
- Gattung „Candidatus Caldatribacterium“ Dodsworth et al. 2013,[7] früher OP9-1/Genus 33[20]
- Spezies „Ca. Caldatribacterium californiense“ corrig.Dodsworth et al. 2013 (N), mit Schreibvariante „Ca. C. californiensis“, alias Caldatribacterium californiense_A mit Stamm OP9-cSCG (G)
- Spezies Caldatribacterium californiense_B mit Stamm SpSt-747 (G)
- Spezies „Ca. Caldatribacterium saccharofermentans“ Dodsworth et al. 2013 mit Stämmen SpSt-82, SpSt-31 und Referenzstamm OP9-77CS alias MAG-77CS (N,G)
- Spezies Caldatribacterium sp014359405 mit MAG MAG-105 (G)
- Gattung „Candidatus Caldatribacterium“ Dodsworth et al. 2013,[7] früher OP9-1/Genus 33[20]
- Familie Atribacteraceae Katayama et al. 2021 (L,N,G)
- Ordnung Atribacterales Katayama et al. 2021 (L,N,G), synonym mit „Caldatribacteriales“
- Klasse JS1 (G), veraltet Candidate division JS1 im Rang eines eigenständigen Phylums[15]
- Ordnung SB-45 (G)
- Familie 34-128 (G)
- Gattung 34-128 (G) alias JS1-1/Genus 1[20]
- Spezies 34-128 sp000405345 (G) alias Atribacteria bacterium SCGC AAA255-N14, Candidate division OP9 bacterium SCGC AAA255-N14 (N)[21]
- Spezies 34-128 sp000405605 (G) alias Atribacteria bacterium SCGC AAA255-G05, Candidate division OP9 bacterium SCGC AAA255-G05 (N)[22]
- Spezies 34-128 sp002084865 (G) alias Ca. Atribacteria bacterium 4572_76 (N)[23] mit Stamm/MAG 4572_76 (Guaymas-Becken)[20]
- 10 weitere Spezies-Kandidaten (G)
- Gattung CG2-30-33-13 (G) alias JS1-7/Genus[20]
- Spezies CG2-30-33-13 sp002782675 (G) alias Ca. Atribacteria bacterium CG_4_8_14_3_um_filter_34_18 (N)[24] (CG = Crystal Geyser)
- Gattung UBA4082 (G) alias JS1-5[20]
- Spezies UBA4082 sp002383355 (G) alias Ca. Atribacteria bacterium UBA5772 (N)[25] mit Stamm UBA5772
- Gattung UBA6251 (G) alias JS1-2/Genus 5[20]
- Gattung UBA9904 (G)
- Gattung 34-128 (G) alias JS1-1/Genus 1[20]
- Familie UBA6794 (G)
- Familie 34-128 (G)
- Ordnung SB-45 (G)
Atribacter

Einzige offizielle Spezies der Gattung Atribacter ist A. laminatus mit Referenzstamm RT761 (notiert als RT761T). Zur Zeit seiner Entdeckung war dies der einzige bekannte Vertreter der Prokaryoten, dessen Zellen eine Kompartimentierung durch innere Membranen (insbesondere um das DNA-Genom herum) aufweisen, ein ansonsten für Eukaryoten (komplex-zelluläre Organismen: Tiere, Pilze, Pflanzen, Protisten) typisches Merkmal (inzwischen hat man zellinterne Membranen auch bei den Riesenbakterien der Gattung Thiomargarita gefunden).
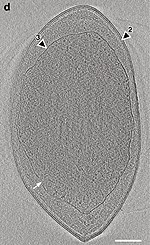
Der Stamm wurde 2020 von Katayama et al. isoliert. Der Stamm ist ubiquitär, d. h. in entsprechenden Habitaten über die ganze Erde verbreitet. Bei dem Isolat RT761 handelt es sich um ein unter der Oberfläche lebendes anaerobes Bakterium. Wie die Kryo-ET (Kryoelektronentomographie) zeigt, besitzt es offenbar drei lipidmembranähnliche Schichten (en. lipid membrane-like layers, LMLs), was von den Autoren gedeutet wird als die klassische gramnegative Struktur mit einer zusätzlichen intrazytoplasmatische (intrazellulären) Membran, die das Nukleoid zu umgeben scheint. Die Genomanalyse zeigt auch einen für gramnegative Bakterien sehr hohen Anteil an Transmembranproteinen.[1]
Die Proben wurden aus einem Absetzbecken in Mobara (Präfektur Chiba, Japan) entnommen, das angelegt wurde, um suspendierte Sandpartikel aus dem Wasser zu entfernen, das aus gasführenden Grundwasserleitern im Tiefenbereich von 490-900 m stammt. Die Wassertemperatur lag bei 24,4 °C, der pH-Wert bei 7,7.[1]
In Reinkultur sind die Endprodukte des Glucose-Abbaus Acetat, Wasserstoff und Kohlendioxid. Für das Wachstum war Hefeextrakt erforderlich. Eine Co-Kultivierung mit Wasserstoff verstoffwechselnde Methanogenen (wie Methanothermobacter thermoautotrophicus Stamm Delta H[30]) beschleunigte das Wachstum.[1]
- A. laminatus RT761T. Konfokalmikroskopie zeigt die Orte von DNA und RNA innerhalb der intrazytoplasmatischen Membran. DNA, RNA und Membranlipide wurden verschiedenen Methoden gefärbt, a ist Phasenkontrast-, b-d Konfokal-Bilder, e-g Bildüberlagerungen. Balken 1μm.
- Zellstruktur beispielhaft bei Atribacteria (A. laminatus RT761T), Thermotogae (Thermotoga maritima) und Dictyoglomi (Dictyoglomus thermophilum). Äußere Membran (schwarz), die Zytoplasmamembran (blau), die intrazytoplasmatische Membran (lila) und Nukleoid (gelb).
Caldatribacterium
Der Referenzstamm OP9-cSCG von „Ca. Caldatribacterium californiense“ wurde aus der Metagenomik von Sedimentproben der heißen Quelle LHC4 des Little Hot Creek in der Long Valley Caldera im US-Bundesstaat Kalifornien gefunden.[7]
Der Referenzstamm 77CS (CS = corn stover ‚Maisstoppel‘) von „Ca. C. saccharofermentans“ stammt ebenfalls aus heißen Quellen des Little Hot Creek (LHC) und aus einer heißen Quelle im Großen Beckens (Great Boiling Spring,[31][32] GBS, US-Bundesstaat Nevada).[7]
Die Genomdaten dieser beiden Stämme erwiesen sich als so ähnlich, dass eine Zuordnung zu einer gemeinsamen Gattung angebracht war.[7][5] Die GTDB ordnet diese Gattung inzwischen innerhalb einer eigenen Familie Caldatribacteraceae zusammen mit Atribacter der gemeinsamen Ordnung Atribacterales zu.[5]
Etymologie
Der Präfix des Phylums, der Klasse Atribacteria, und der Typusgattung kommt von lateinisch ater ‚schwarz‘, der Mittelteil ‚-bacter-‘ bezeichnet stäbchenförmige Bakterien; die jeweilige Endung verweist auf den taxonomischen Rang (Phylum, Klasse, … Gattung). Damit ist gemeint, dass diese Bakterien zur Mikrobiellen Dunkle Materie gehören.[3][33]
Das Namens-Epitheton der Typusspezies, Atribacter laminatus leitet sich ab von lat. lamina, deutsch ‚Schicht, Lage‘, englisch layer; laminatus ‚geschichtet‘, was auf die mehrschichtige Zellstruktur hindeutet.[3]
Der Name der Kandidatengattung Ca. Caldatribacterium leitet sich ab von lat. caldus ‚heiß‘, sowie ebenfalls von ater ‚schwarz‘. Er verweist damit auf ein stäbchenförmiges Bakterium aus einer heißen Umgebung, wobei „schwarz“ oder „dunkel“ sowohl auf die Mikrobielle Dunkle Materie als auch auf die dunklen, anaeroben Umgebungen verweist, in denen diese Bakterien gefunden werden.[7]
Das Artepitheton saccharofermentans leitet sich ab von altgriechisch σάκχαρονsákcharon und nachfolgend lat. saccharum ‚Zucker‘ und neulat. fermentans ‚gärend‘, verweist also auf die anaerobe Vergärung von Zucker; californiense ist neulateinisch und bedeutet aus oder zu Kalifornien gehörend.[7]
Literatur
- Chris Greening, Trevor Lithgow: Formation and function of bacterial organelles. In: Nature Reviews Microbiology. Band 18, Nr. 12, 24. Juli 2020, S. 1–13, 677–689; doi:10.1038/s41579-020-0413-0, ResearchGate.
Weblinks
Einzelnachweise
- ↑ a b c d e f g Taiki Katayama, Masaru K. Nobu, Hiroyuki Kusada, Xian-Ying Meng, Naoki Hosogi, Katsuyuki Uematsu, Hideyoshi Yoshioka, Yoichi Kamagata, Hideyuki Tamaki: Isolation of a member of the candidate phylum ‘Atribacteria’ reveals a unique cell membrane structure. In: Nature Communications, Band 11, Nr. 6381, 14. Dezember 2020; doi:10.1038/s41467-020-20149-5, ResearchGate, PMID 33318506, PMC 7736352 (freier Volltext).
- ↑ a b Aharon Oren, George M. Garrity: Valid publication of the names of forty-two phyla of prokaryotes. In: International Journal of Systematic and Evolutionary Microbiology. Band 71, Nr. 10, 25. Oktober 2021, ISSN 1466-5034, S. 5056, doi:10.1099/ijsem.0.005056, PMID 34694987.
- ↑ a b c d LPSN: Phylum Atribacterota Katayama et al. 2021. Aidan C. Parte, Sardà Carbasse, Jean P. Euzéby et al.: List of Prokaryotic names with Standing in Nomenclature (LPSN). Deutsche Sammlung von Mikroorganismen und Zellkulturen (DSMZ), abgerufen am 26. Juli 2022 (englisch).
- ↑ a b c d e f g h Sayers et al.: Atribacterota. National Center for Biotechnology Information (NCBI) Taxonomy Database, abgerufen am 20. März 2021 (englisch). Details: Atribacterota Katayama et al. 2021 (phylum), vernacular name: Candidate division JS1, Candidate division OP9 (Obsidian Pool 9). Graphisch: Candidatus Atribacteria. Lifemap NCBI Version.
- ↑ a b c d GTDB: Atribacterota (Phylum). GTDB release 07-RS220. In: Genome Taxonomy Database. (englisch).
- ↑ a b LPSN: Phylum „Candidatus Caldatribacteriota“ Hahn et al. 2020. Aidan C. Parte, Sardà Carbasse, Jean P. Euzéby et al.: List of Prokaryotic names with Standing in Nomenclature (LPSN). Deutsche Sammlung von Mikroorganismen und Zellkulturen (DSMZ), abgerufen am 26. Juli 2022.
- ↑ a b c d e f g Jeremy A. Dodsworth, Paul C. Blainey, Senthil K. Murugapiran, Wesley D. Swingley, Christian A. Ross, Susannah G. Tringe, Patrick S. G. Chain, Matthew B. Scholz, Chien-Chi Lo, Jason Raymond, Stephen R. Quake, Brian P. Hedlund: Single-cell and metagenomic analyses indicate a fermentative and saccharolytic lifestyle for members of the OP9 lineage. In: Nature Communications, Band 4, Nr. 1854, 14. Mai 2013; doi:10.1038/ncomms2884, PMID 23673639, PMC 3878185 (freier Volltext).
- ↑ a b Masaru K. Nobu, Jeremy A. Dodsworth, Senthil K. Murugapiran, Christian Rinke, Esther A. Gies, Gordon Webster, Patrick Schwientek, Peter Kille, R. John Parkes, Henrik Sass, Bo B. Jørgensen, Andrew J. Weightman, Wen-Tso Liu, Steven J. Hallam, George Tsiamis, Tanja Woyke, Brian P. Hedlund: Phylogeny and physiology of candidate phylum ‘Atribacteria’ (OP9/JS1) inferred from cultivation-independent genomics. In: Nature: The ISME Journal, Band 10, Nr. 2, Februar 2016, S. 273–286; doi:10.1038/ismej.2015.97, ResearchGate, PMID 26090992, PMC 4737943 (freier Volltext), Epub 19. Juni 2015.
- ↑ Stephanie A. Carr, Beth N. Orcutt, Kevin W. Mandernack, John R. Spear: Abundant Atribacteria in deep marine sediment from the Adélie Basin, Antarctica. In: Frontiers in Microbiology. Band 6, Sektion Extreme Microbiology. S. 872; doi:10.3389/fmicb.2015.00872, PMC 4549626 (freier Volltext), PMID 26379647, (ncbi.nlm.nih.gov PDF).
- ↑ Aurèle Vuillemin, Sergio Vargas, Ömer K. Coskun, Robert Pockalny, Richard W. Murray, David C. Smith, Steven D'Hondt, William D. Orsi: Atribacteria Reproducing over Millions of Years in the Atlantic Abyssal Subseafloor. In: mBio. Band 11, Nr. 5, 27. Oktober 2020, ISSN 2150-7511, S. e01937–20, doi:10.1128/mBio.01937-20, PMID 33024037, PMC 7542362 (freier Volltext) – (Epub 6. Oktober 2020).
- ↑ Christian Rinke, Patrick Schwientek, Alexander Sczyrba, Natalia N. Ivanova, Iain J. Anderson, Jan-Fang Cheng, Aaron Darling, Stephanie Malfatti, Brandon K. Swan, Esther A. Gies, Jeremy A. Dodsworth, Brian P. Hedlund, George Tsiamis, Stefan M. Sievert, Wen-Tso Liu, Jonathan A. Eisen, Steven J. Hallam, Nikos C. Kyrpides, Ramunas Stepanauskas, Edward M. Rubin, Philip Hugenholtz, Tanja Woyke: Insights into the phylogeny and coding potential of microbial dark matter. In: Nature. Band 499, 14. Juli 2013, S. 431–437; doi:10.1038/nature12352, ResearchGate, PMID 23851394.
- ↑ Laura A. Hug, Brett J. Baker, Karthik Anantharaman, Christopher T. Brown, Alexander J. Probst, Cindy J. Castelle, Cristina N. Butterfield, Alex W. Hernsdorf, Yuki Amano, Kotaro Ise, Yohey Suzuki, Natasha Dudek, David A. Relman, Kari M. Finstad, Ronald Amundson, Brian C. Thomas, Jillian F. Banfield: A new view of the tree of life. In: Nature Microbiology. Band 1, Nr. 5, 11. April 2016, S. 1–6, doi:10.1038/nmicrobiol.2016.48.
- ↑ Kerrin Mendler, Han Chen, Donovan H Parks, Briallen Lobb, Laura A Hug, Andrew C Doxey: AnnoTree: visualization and exploration of a functionally annotated microbial tree of life. In: Nucleic Acids Research. Band 47, Nr. 9, 2019, ISSN 0305-1048, S. 4442–4448, doi:10.1093/nar/gkz246, PMID 31081040.
- ↑ a b c OneZoom: Candidate division OP9.
- ↑ a b OneZoom: Candidate division JS1.
- ↑ UniProt: Taxonomy – Atribacter (genus).
- ↑ NCBI: MAG: Candidatus Atribacteria bacterium isolate AS27yjCOA_181.
- ↑ NCBI: MAG: Candidatus Atribacteria bacterium isolate AS27yjCOA_164.
- ↑ NCBI: MAG: Candidatus Atribacteria bacterium isolate Gw_SlDig_bin_239.
- ↑ a b c d e f g Yi-Fan Liu, Zhen-Zhen Qi, Li-Bin Shou, Jin-Feng Liu, Shi-Zhong Yang, Ji-Dong Gu, Bo-Zhong Mu: Anaerobic hydrocarbon degradation in candidate phylum ‘Atribacteria’ (JS1) inferred from genomics. In: Nature: The ISME Journal, Band 13, 6. Juni 2019, S. 2377–2390; doi:10.1038/s41396-019-0448-2. Siehe insbes. Fig. 1.
- ↑ NCBI: Atribacteria bacterium SCGC AAA255-N14 (species).
- ↑ NCBI: Atribacteria bacterium SCGC AAA255-G05 (species).
- ↑ NCBI: Candidatus Atribacteria bacterium 4572_76 (species).
- ↑ NCBI: Candidatus Atribacteria bacterium CG_4_8_14_3_um_filter_34_18 (species).
- ↑ NCBI: Candidatus Atribacteria bacterium UBA5772 (spezies).
- ↑ NCBI: Candidatus Atribacteria bacterium UBA4082 (species).
- ↑ NCBI: Candidatus Atribacteria bacterium UBA6251 (species).
- ↑ NCBI: MAG: Candidatus Atribacteria bacterium isolate AS21ysBPME_310.
- ↑ NCBI: Candidatus Atribacteria bacterium UBA6794 (species).
- ↑ NCBI: Methanothermobacter thermautotrophicus str. Delta H (strain).
- ↑ Great Boiling Spring. Geographic Names Information System. United States Geological Survey.
- ↑ Great Boiling Spring Park (historical). Geographic Names Information System. United States Geological Survey.
- ↑ LPSN: Phylum „Candidatus Atribacteria“ Dodsworth et al. 2013.
Auf dieser Seite verwendete Medien
Autor/Urheber: Taiki Katayama, Masaru K. Nobu, Hiroyuki Kusada, Xian-Ying Meng, Naoki Hosogi, Katsuyuki Uematsu, Hideyoshi Yoshioka, Yoichi Kamagata, Hideyuki Tamaki, Lizenz: CC BY-SA 4.0
Morphology and membrane structure in Atribacter laminatus (type strain RT761): Scanning electron microscopy. Scale bar, 1.5 μm.
Autor/Urheber: Taiki Katayama, Masaru K. Nobu, Hiroyuki Kusada, Xian-Ying Meng, Naoki Hosogi, Katsuyuki Uematsu, Hideyoshi Yoshioka, Yoichi Kamagata, Hideyuki Tamaki. Scale bar added from Fig. 1d., Lizenz: CC BY-SA 4.0
Morphology and membrane structure in Atribacter laminatus (type strain RT761) cells showing the presence of three lipid membrane-like layers (LMLs) with the innermost layer surrounding the nucleoid: Cryo-electron tomography. 3D-rendered reconstruction of a cell. Color code: outer LML, orange; middle LML, blue; inner LML, yellow; ribosome, green. Scale bar, 0.1 μm.
Autor/Urheber: Taiki Katayama, Masaru K. Nobu, Hiroyuki Kusada, Xian-Ying Meng, Naoki Hosogi, Katsuyuki Uematsu, Hideyoshi Yoshioka, Yoichi Kamagata, Hideyuki Tamaki, Lizenz: CC BY-SA 4.0
Morphology and membrane structure in Atribacter laminatus (type strain RT761) cells showing the presence of three lipid membrane-like layers (LMLs) with the innermost layer surrounding the nucleoid: Cryo-electron tomography. Black arrowheads indicate the outer (1), middle (2), and inner (3) LMLs. White arrowheads indicate the 2.2 nm thick layer (1) and faint layers (2). Scale bar, 0.2 μm.
Autor/Urheber: Taiki Katayama, Masaru K. Nobu, Hiroyuki Kusada, Xian-Ying Meng, Naoki Hosogi, Katsuyuki Uematsu, Hideyoshi Yoshioka, Yoichi Kamagata, Hideyuki Tamaki. Inside caption slightly moved., Lizenz: CC BY-SA 4.0
Atribacter laminatus, type strain RT761. Confocal-laser microscopy showing the localization of DNA and RNA within the intracytoplasmic membrane structure. DNA, RNA, and membrane lipids were stained by Hoechst (blue), SYTO RNAselect (green) and FM4-64 (red), respectively. Outlines of the cell from a are included in all panels. a Phase contrast image. b–d Confocal-laser images. e–g Image overlays. Scale bars, 1 μm.
Autor/Urheber: Taiki Katayama, Masaru K. Nobu, Hiroyuki Kusada, Xian-Ying Meng, Naoki Hosogi, Katsuyuki Uematsu, Hideyoshi Yoshioka, Yoichi Kamagata, Hideyuki Tamaki, Lizenz: CC BY-SA 4.0
Membrane-related features observed for phyla with unique cell structures. Cell structures of select species are shown for Atribacteria (Atribacter laminatus RT761, red/left), Thermotogae (Thermotoga maritima, green/center), and Dictyoglomi (Dictyoglomus thermophilum, orange/reight). The illustrations indicate the outer membrane (black), cytoplasmic membrane (blue), intracytoplasmic membrane (purple), and nucleoid (yellow). * For RT761, the shown schematic requires further investigation to conclude the identity/role of each layer.
Autor/Urheber: Taiki Katayama, Masaru K. Nobu, Hiroyuki Kusada, Xian-Ying Meng, Naoki Hosogi, Katsuyuki Uematsu, Hideyoshi Yoshioka, Yoichi Kamagata, Hideyuki Tamaki, Lizenz: CC BY-SA 4.0
Morphology and membrane structure in Atribacter laminatus (type strain RT761) cells showing the presence of three lipid membrane-like layers (LMLs) with the innermost layer surrounding the nucleoid: Cryo-electron tomography. White arrow indicates inner LML invagination. Scale bar, 0.1 μm.
Autor/Urheber: Taiki Katayama, Masaru K. Nobu, Hiroyuki Kusada, Xian-Ying Meng, Naoki Hosogi, Katsuyuki Uematsu, Hideyoshi Yoshioka, Yoichi Kamagata, Hideyuki Tamaki, Lizenz: CC BY-SA 4.0
Morphology and membrane structure in Atribacter laminatus (type strain RT761) cells showing the presence of three lipid membrane-like layers (LMLs) with the innermost layer surrounding the nucleoid. Transmission electron micrograph of a thin section of RT761 cells. N nucleoid. Scale bar, 0.5 μm.