Archaeen
| Archaeen | ||||
|---|---|---|---|---|
 Die vielfältige Morphologie von Archaeen veranschaulichende Bildkomposition | ||||
| Systematik | ||||
| ||||
| Wissenschaftlicher Name | ||||
| Archaea | ||||
| Otto Kandler & Mark L. Wheelis | ||||
| Überstämme | ||||
Die Archaeen (Archaea, Singular: Archaeon; von altgriechisch ἀρχαῖοςarchaĩos, deutsch ‚uralt‘, ‚ursprünglich‘),[1] früher auch Archaebakterien, Archebakterien oder Urbakterien genannt, bilden eine der drei Domänen, in die alle zellulären Lebewesen eingeteilt werden. Die anderen beiden Domänen sind die Bakterien (Bacteria), die mit den Archaeen zu den Prokaryoten zusammengefasst werden, und die Eukaryoten (Eukaryota), die im Unterschied zu den Prokaryoten einen Zellkern mit Kernmembran besitzen. Archaeen hingegen haben (wie auch die Bakterien) in sich geschlossene DNA-Moleküle (zirkuläre Chromosomen[2]), die im Zellplasma als Kernäquivalent ohne Hülle vorliegen. Die Archaea sind einzellige Organismen.
Bislang sind keine Krankheitserreger aus der Gruppe der Archaeen bekannt.
Eigenschaften
In vielen molekularbiologischen Eigenschaften sind die Archaeen den Eukaryoten ähnlicher als den Bakterien. Dennoch besitzen sie typisch bakterielle Eigenschaften, z. B. die Zellgröße, das Fehlen eines Zellkerns, die Art der Zellteilung, sie besitzen ein in sich geschlossenes DNA-Molekül, ebenfalls verhältnismäßig einfach aufgebaute Fortbewegungsorgane (Flagellen) und – wie die Bakterien – Ribosomen mit dem Sedimentationskoeffizienten 70S (allerdings sind die Archaeen-Ribosomen komplexer in ihrer Struktur). Die Gene beider Domänen sind in sogenannten Operons organisiert. Archaeen können auch Plasmide tragen, wie beispielsweise ein Archaeon (mit der vorläufigen Bezeichnung Sulfolobus NOB8H2) der Familie Sulfolobaceae im Phylum Crenarchaeota.[3]
Die zentralen molekularen Prozesse, zum Beispiel Translation und Transkription, sind dagegen denjenigen der Eukaryoten recht ähnlich: Archaeen benutzen ähnliche, aus mehreren Proteinuntereinheiten zusammengesetzte RNA-Polymerasen (Rifampicin- und Streptolydigin-resistent), bei der Translation kommen sehr ähnliche Initiations- und Elongationsfaktoren vor, der Beginn der Transkription wird durch eine sogenannte TATA-Box markiert.

Die Archaeen besitzen jedoch auch viele einzigartige Eigenschaften, besonders der Aufbau der Zellwand zeigt deutliche Unterschiede zu den anderen Domänen: Die Archaeen-Zellwände enthalten Pseudopeptidoglycan (Pseudomurein) und sind generell sehr vielfältig in ihrem Aufbau: Manchen Archaeen fehlt eine Zellwand völlig (Thermoplasma), andere besitzen hochkomplexe, aus vielen Schichten bestehende Zellwände (Methanospirillum). Aufgrund des anderen Aufbaus sind Archaeen generell gegen Zellwandantibiotika resistent. Auch die Zusammensetzung der Archaeen-Plasmamembran unterscheidet sich: In Bakterien und Eukaryoten sind Fettsäuren über eine Esterbindung an die Glycerolmoleküle gebunden, bei Archaeen findet man Glyceroldiether oder sogar Bis-Glycerol-Tetraether (einschichtige Membran, Monolayer) und verzweigte Isopreneinheiten statt einfacher Fettsäuren. Hyperthermophile Archaeen besitzen häufig derart stabilisierte Zellmembranen (Glycerol-Tetraether),[4] die nicht nur thermostabiler sind, sondern auch Anpassungen an saure Umgebungen darstellen können.[5]
Einige Archaeenarten können sich in Relation zu ihrer Größe sehr schnell fortbewegen. Mit 400 bis 500 „Körperlängen pro Sekunde“ (englisch bodies per second, abgekürzt „bps“) sind M. jannaschii und M. villosus der Gattung Methanocaldococcus die schnellsten bislang vermessenen Lebewesen.[6] Zum Vergleich: Ein Sportwagen mit 400 bps käme auf eine Geschwindigkeit von über 6000 km/h (nahezu Hyperschallgeschwindigkeit). Das Bakterium Escherichia coli bewegt sich dagegen mit rund 20 bps fort, ähnlich schnell wie ein Gepard.
Viele kultivierte Arten der Archaeen sind an extreme Milieubedingungen angepasst. So gibt es Arten, die bevorzugt bei Temperaturen von über 80 °C wachsen (hyper-thermophil), andere leben in hoch konzentrierten Salzlösungen (halophil) oder in stark saurem Milieu (pH-Wert zuweilen sogar unterhalb 0; acidophil) bzw. stark basischem Milieu (pH-Wert oberhalb von 10; alkaliphil). Thermoplasmatales der Gattung Picrophilus (P. oshimae und P. torridus) haben ein Wachstumsoptimum bei einem pH-Wert von 0,7 und können sogar bei einem pH-Wert von −0,06 noch überleben.
Archaeen sind in der Forschung von Interesse, da in ihnen vielleicht Merkmale des frühen Lebens auf der Erde erhalten geblieben sind. Aber auch ihr außergewöhnlicher Stoffwechsel ist von Interesse, zum Beispiel die Fähigkeit, bei 110 °C zu wachsen.
Stoffwechsel
Die meisten der bisher bekannten Archaeenarten sind autotroph, d. h., sie benötigen zum Wachstum keine organischen Stoffe, sie gewinnen den Kohlenstoff zum Aufbau ihrer Körperbestandteile ausschließlich durch Assimilation von Kohlenstoffdioxid. Aber auch Heterotrophie, die Gewinnung des Kohlenstoffs aus organischen Verbindungen, ist weit verbreitet.
Die Mehrzahl der bisher kultivierten Archaeen zeichnet sich durch einen anaeroben Stoffwechsel aus; für viele anaerobe Archaeen ist Sauerstoff (O2) toxisch.
Eine Besonderheit Archaeen-Stoffwechsels ist die Methanogenese, die ausschließlich von Methan produzierenden Archaeen, den sogenannten Methanogenen, vollbracht werden kann. Sie besitzen eine Reihe einzigartiger Cofaktoren, beispielsweise Coenzym F420 oder Methanofuran.
Die meisten hyperthermophilen Archaeen sind Anaerobier; der energiegewinnende Stoffwechsel ist entweder chemoorganotroph oder chemolithotroph (die Energie wird aus chemischen Umsetzungen organischer bzw. anorganischer Verbindungen gewonnen). Schwefelverbindungen spielen hierbei oft eine große Rolle: Während des Stoffwechsels wird der Schwefel reduziert und dabei Energie freigesetzt.
Bekannt ist aber der Schwefelstoffwechsel der extrem thermo- und acidophilen Art Acidianus ambivalens (früher: Desulfurolobus ambivalens) aus der Ordnung Sulfolobales, welche aerob Schwefel oxidieren kann.[7]
Halophile Archaeen sind meist aerob-chemoorganotroph, sie gewinnen ihre Energie aus chemischen Umsetzungen von organischen Verbindungen. Unter anoxischen Bedingungen oder bei Nährstoffmangel sind viele extrem Halophile sogar zur Nutzung von Lichtenergie fähig: Sie besitzen das Protein Bacteriorhodopsin, das Licht absorbiert und den Protonentransfer durch die Cytoplasmamembran katalysiert; der dadurch erzeugte elektrochemische Gradient treibt die ATPase und damit die ATP-Synthese an.
Morphologie
Wie die Bakterien sind auch die Archaeen in ihrer Form äußerst divers. Die Größen bzw. Längen der Archaeenzellen variieren von etwa 0,4 (Nanoarchaeum equitans) bis zu 100 µm (Methanospirillum hungatei), durchschnittlich sind die Zellen etwa 1 µm groß. Die Zellen zeigen verschiedene Formen, z. B.: Kokken (z. B. Methanococcus jannaschii), Stäbchen (Thermoproteus neutrophilus), Spirillen-förmig (Methanospirillum hungatei), gelappte Kokken (Archaeoglobus fulgidus), Scheiben (Thermodiscus maritimus), lange Filamente (Thermofilum pendens) oder sogar quadratisch (Haloquadratum walsbyi).
Sie besitzen oft Geißeln (Flagellen) zur Fortbewegung, oder auch fadenartige Anhängsel (Pili) zur Anheftung an Oberflächen.
Genetik
Archaeen weisen in der Regel ein einziges zirkuläres Chromosom auf,[8] wobei bei zahlreichen Methanobacteriati (früher „Euryarchaeota“ i. w. S. oder „Euryarchaeida“) nachgewiesen wurde, dass sie mehrere Kopien dieses Chromosoms besitzen.[9] Das größte bekannte Archaeen-Genom, das im Jahr 2002 ermittelt wurde, umfasste 5.751.492 Basenpaare in Methanosarcina acetivorans.[10] Das Genom von Nanoarchaeum equitans mit 490.885 Basenpaaren ist nur ein Zehntel so groß und ist das kleinste bekannte Archaeen-Genom; es enthält schätzungsweise nur 537 proteinkodierende Gene.[11] Kleinere unabhängige DNA-Stücke, sogenannte Plasmide, sind ebenfalls in Archaeen zu finden. Plasmide können in einem Prozess, der der bakteriellen Konjugation ähneln könnte, durch physischen Kontakt zwischen Zellen übertragen werden.[12][13]
Archaeen unterscheiden sich genetisch von Bakterien und Eukaryoten. Bis zu 15 % der Proteine, für die ein Archaeen-Genom kodiert, sind einzigartig für die Domäne; allerdings haben die meisten dieser Gene keine bekannte Funktion.[14] Die übrigen Proteine mit identifizierter Funktion gehören überwiegend zu den Methanobacteriati und sind an der Methanogenese beteiligt. Die Proteine, die von Archaeen, Bakterien und Eukaryoten gemeinsam aufgebaut werden, stellen einen gemeinsamen Grundbaustein der Zellfunktionen dar, der sich hauptsächlich auf die Transkription, die Translation und den Nukleotidstoffwechsel bezieht.[15] Andere charakteristische Merkmale der Archaeen sind die Organisation von Genen mit verwandter Funktion – wie Enzyme, die Schritte desselben Stoffwechselwegs in neuartige Operons katalysieren – sowie große Unterschiede in tRNA-Genen und deren Aminoacyl-tRNA-Synthetasen.[16]
Die Transkription in Archaeen ähnelt mehr der eukaryotischen als der bakteriellen Transkription, wobei die RNA-Polymerase der Archaeen ihrem Äquivalent in Eukaryoten sehr nahe kommt,[8] während die Translation der Archaeen Anzeichen sowohl bakterieller als auch eukaryotischer Äquivalente aufweist.[17] Obwohl Archaeen nur einen Typ von RNA-Polymerase haben, scheinen ihre Struktur und Funktion bei der Transkription der eukaryotischen RNA-Polymerase II sehr ähnlich zu sein, wobei ähnliche Proteinanordnungen (die allgemeinen Transkriptionsfaktoren) die Bindung der RNA-Polymerase an den Promotor eines Gens steuern.[18] Andere Transkriptionsfaktoren von Archaeen sind allerdings näher an denen von Bakterien.[19] Die posttranskriptionelle Modifikation ist einfacher als bei Eukaryonten, da den meisten Archaeengenen Introns fehlen, obwohl es viele Introns in ihren Transfer-RNA- und ribosomalen RNA-Genen gibt[20] und Introns in einigen wenigen proteinkodierenden Genen vorkommen können.[21][22]
Gentransfer
Haloferax volcanii, ein extrem halophiles Archaeon, bildet zytoplasmatische Brücken zwischen den Zellen, die offenbar für die Übertragung von DNA von einer Zelle zur anderen in beide Richtungen genutzt werden.[23]
Wenn die hyperthermophilen Archaeen Sulfolobus solfataricus[24] und Sulfolobus acidocaldarius[25] einer DNA-schädigenden UV-Bestrahlung oder den Wirkstoffen Bleomycin oder Mitomycin C ausgesetzt werden, wird eine artspezifische zelluläre Aggregation ausgelöst. Die Aggregation in S. solfataricus konnte nicht durch andere physikalische Stressfaktoren wie pH-Wert oder Temperaturveränderung ausgelöst werden,[26] was darauf hindeutet, dass die Aggregation speziell durch DNA-Schäden ausgelöst wird. Ajon et al.[27] zeigten, dass UV-induzierte zelluläre Aggregation den Austausch chromosomaler Marker in S. acidocaldarius mit hoher Frequenz vermittelt. Die Rekombinationsraten übertrafen die von nicht induzierten Kulturen um bis zu drei Größenordnungen. Frols et al.[26][28] und Ajon et al.[27] stellten die Hypothese auf, dass die zelluläre Aggregation den artspezifischen DNA-Transfer zwischen Sulfolobus-Zellen fördert, um die Reparatur beschädigter DNA durch homologe Rekombination zu verbessern. Bei dieser Reaktion könnte es sich um eine primitive Form der sexuellen Interaktion handeln, ähnlich wie bei besser untersuchten bakteriellen Transformationssystemen, die ebenfalls mit einem artspezifischen DNA-Transfer zwischen Zellen verbunden sind, der zu einer homologen Rekombinationsreparatur von DNA-Schäden führt.[29]
Fortpflanzung
Archaeen pflanzen sich ungeschlechtlich durch binäre oder multiple Fissiparie, Fragmentierung oder Knospung fort. Mitose und Meiose kommen nicht vor, sodass, wenn eine Archaeenart in mehr als einer Form existiert, alle dasselbe genetische Material haben.[30] Die Zellteilung wird durch einen Zellzyklus gesteuert; nachdem das Chromosom der Zelle repliziert wurde und sich die beiden Tochterchromosomen getrennt haben, teilt sich die Zelle.[31] In der Gattung Sulfolobus weist der Zyklus Merkmale auf, die sowohl bakteriellen als auch eukaryotischen Systemen ähneln. Die Chromosomen werden von mehreren Ausgangspunkten (Replikationsursprüngen) aus mit Hilfe von DNA-Polymerasen repliziert, die den entsprechenden eukaryotischen Enzymen ähneln.[32]
In Methanobacteriati („Euryarchaeota“) ähneln das Zellteilungsprotein FtsZ, das einen sich zusammenziehenden Ring um die Zelle bildet, und die Bestandteile des Septums, das quer durch das Zentrum der Zelle gebaut wird, ihren bakteriellen Äquivalenten.[31] In Cren-[33][34] und Thaumarchaea[35] erfüllt der Zellteilungsapparat Cdv eine ähnliche Rolle. Dieser Mechanismus ist mit dem eukaryotischen ESCRT-III-Zellteilungsapparat verwandt. Obwohl dieser am besten für seine Rolle bei der Zellsortierung bekannt ist, ist er auch an der Trennung von teilenden Zellen beteiligt, was auf eine ursprüngliche Funktion in der Zellteilung hindeutet.[36]
Sowohl Bakterien als auch Eukaryoten, nicht aber Archaeen, bilden Sporen.[37] Einige Haloarchaea-Arten durchlaufen eine phänotypische Umstellung und wachsen als mehrere verschiedene Zelltypen, einschließlich dickwandiger Strukturen, die gegen osmotischen Schock resistent sind und den Archaeen das Überleben in Wasser mit niedrigen Salzkonzentrationen ermöglichen; diese Strukturen sind jedoch nicht als reproduktive Strukturen zu bezeichnen, sondern könnten ihnen vielmehr dabei helfen, neue Lebensräume zu erreichen.[38]
Habitate

Die meisten der bisher bekannten Archaeen sind Extremophile, d. h. den extremen Bedingungen ihrer Biotope angepasst. Viele Vertreter besitzen die Fähigkeit, bei sehr hohen Temperaturen (Hyperthermophile über 80 °C), sehr niedrigen oder sehr hohen pH-Werten (Acidophile bzw. Alkaliphile), hohen Salzkonzentrationen (Halophile) oder hohen Drücken (Barophilie) zu leben.
Hyperthermophile Archaeen findet man häufig in vulkanischen Gebieten, marinen (Schwarzer Raucher) und terrestrischen (Geysire, Solfatarenfelder), so z. B. vulkanisch geprägten Habitaten des Yellowstone-Nationalparks. Halophile gedeihen gut in Umgebungen mit hohem Salzgehalt, so z. B. im Toten Meer oder auch in natürlich vorkommenden marinen Solen.
Auch methanogene Archaeen sind in gewisser Weise „extrem“: Sie wachsen ausschließlich unter anoxischen Bedingungen und benötigen häufig molekularen Wasserstoff für ihren Stoffwechsel. Diese Archaeenarten sind relativ weit verbreitet und kommen in Süßwasser, Meer und Boden vor, aber auch als Symbionten im Darmtrakt von Tieren und Menschen. Archaeen konnten sogar in den Falten des menschlichen Bauchnabels nachgewiesen werden, wobei dies selten ist.[39]
Wegen dieser „Extremophilie“ hat man die ökologische Bedeutung der Archaeen zunächst als relativ gering eingeschätzt. Erst in den letzten Jahren wurde durch Einsatz feinerer molekularbiologischer Nachweismethoden deutlich, dass Archaeen zu großen Anteilen in verhältnismäßig kaltem Meerwasser, aber auch in Böden und Süßwasser-Biotopen vorkommen. In bestimmten ozeanischen Bereichen machen z. B. Crenarchaeota bis zu 90 % der vorhandenen Lebewesen aus. Insgesamt schätzt man, dass in den Ozeanen etwa 1,3 × 1028 Archaeen und 3,1 × 1028 Bakterien vorkommen.
Die Mehrzahl der isolierten und als Reinkultur im Labor verfügbaren Archaeenarten ist allerdings nach wie vor „extremophil“; in einigen Fällen ist eine Kultivierung auch unter weniger extremen Bedingungen gelungen. Aus den bisherigen Untersuchungen geht hervor, dass die Archaeen eine bedeutende Rolle für den Stickstoff-, Kohlenstoff- und Schwefelkreislauf im Ökosystem der Erde spielen.
Systematik
Die separate Stellung der Archaeen als eine eigenständige Domäne ist begründet durch eine Reihe genetischer, physiologischer, struktureller und biochemischer Merkmale, insbesondere deutliche Unterschiede in der Sequenz der in den Ribosomen enthaltenen RNA (der kleinen ribosomalen Untereinheit, 16S rRNA).
Ende der 1970er Jahre wurde von den US-amerikanischen Mikrobiologen Carl Woese und George Fox die Eigenständigkeit der Archaeen und ihre Zugehörigkeit zu einer eigenen systematischen Einheit neben den Bakterien (Eubakterien) und Eukaryoten erkannt und beschrieben. In der Sequenz der ribosomalen RNA entdeckten die Forscher auffällige Unterschiede zu Bakterien. Auch die Struktur der Zellen und deren Eigenheiten im Stoffwechsel ließen auf eine separate Gruppe von Prokaryoten schließen.[40] Diese Ergebnisse wurden in den folgenden Jahren bestätigt, und weitreichende Fortschritte in der molekularen Biologie machten eine generelle Änderung der Taxonomie notwendig: Eubakterien wurden in Bacteria umbenannt und Archaebakterien in Archaea. Beide wurden 1990 im Rahmen eines Drei-Domänen-Systems als zwei eigenständige Domänen neben der Domäne der Eukarya beschrieben.[41] Dabei stehen die Archaea den Eukarya phylogenetisch vermutlich näher als die Bacteria. Zwar lassen sich in Archaeen keine Zellorganellen finden, doch können sie zur Stabilisierung ihrer Form besondere, einem Cytoskelett vergleichbare Filamente ausbilden.[42][43]
Für die Beschreibung von Genus und Art gibt es eine festgelegte Prozedur.[44] Durch Publikation oder Validierung im International Journal of Systematic and Evolutionary Microbiology (IJSEM)[45] sind Gattung und Art festgelegt. Höhere Taxa können hier auch beschrieben werden. Der aktuelle Stand kann in der List of Prokaryotic names with Standing in Nomenclature (LPSN),[46] gepflegt durch Jean Euzéby, eingesehen werden. Dies entspricht dem Internationalen Code der Nomenklatur der Prokaryoten (ICNP).[47] Taxa, die diesem Standard nicht entsprechen, werden in Anführungszeichen dargestellt.
Darüber hinaus wurde die globale Einteilung innerhalb der Archaeen und Bakterien mittels phylogenetischer Analyse des 16S-rRNS-Gens reformiert.[48] Eine aktuelle Zusammenstellung der Taxa aus dieser und zahlreichen weiterführenden Publikationen erscheint in Bergey’s Manual,[49] und in Taxonomic Outline of the Bacteria and Archaea,[50] wobei mittlerweile zusätzlich zum 16S-rRNS-Gen teilweise weitere phylogenetische Markergene hinzugezogen werden. Einige dieser Taxa haben ihre Berechtigung, sind aber bis heute nicht valide publiziert oder werden generell nicht vom ICNB erfasst. Solche Taxa werden in Anführungszeichen dargestellt.
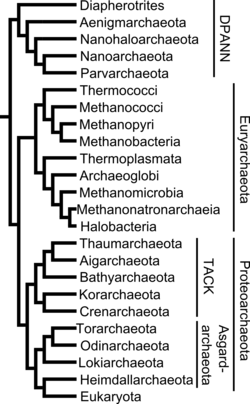

Systematik bis 2024
Die hier wiedergegebene herkömmliche Systematik enthält Taxa im Rang von Phylum bis herunter zur Familie. Bei manchen Taxa gibt es widersprüchliche Einträge. Diese wurden anhand von Originalliteratur und einer phylogenetischen Analyse auf Stichhaltigkeit geprüft.[51][52][53]
Vor einigen Jahren wurde die Beschreibung der zusätzlichen Phyla „Korarchaeota“ und „Nanoarchaeota“ veröffentlicht. Ein Vertreter des vorgeschlagenen Phylums „Nanoarchaeota“ konnte erfolgreich co-kultiviert[54] und sein Genom sequenziert werden,[55] das sogenannte Nanoarchaeum equitans. Vom vorgeschlagenen Phylum „Korarchaeota“, zunächst anhand seiner 16S rRNA-Gen-Basensequenzen in Proben heißer Quellen nachgewiesen,[56] gibt es Anreicherungskulturen. Daraus konnte nun die komplette Basensequenz des Genoms veröffentlicht werden,[57] mit dem informellen Namen „Candidatus Korarchaeum cryptofilum“ versehen. Ohne isolierte Stämme haben die Vertreter dieser Phyla nach den derzeitigen Regeln des ICSB keinen validierten Platz in der Taxonomie,[58] stellen jedoch zwei von vier bekannten Phyla der Archaeen dar.
- Superphylum „Euryarchaeota“ Woese et al. 1990 bzw. „Euryarchaeida“ Luketa 2012
- Klasse Archaeoglobi
- Klasse Halobacteria
- Klasse Methanobacteria
- Klasse „Methanococci“ Boone 2002
- Klasse „Methanomicrobia“
- Klasse „Methanopyri“ Garrity & Holt 2002
- Klasse Thermococci
- Klasse Thermoplasmata
- Klasse „Eurythermea“ Cavalier-Smith 2002[59]
- Klasse „Neobacteria“ Cavalier-Smith 2002[59]
- Klasse „Hadesarchaea“
- Superphylum „DPANN“
- Superphylum „Proteoarchaeota“ Petitjean et al. 2014
- Superphylum „TACK“ (Thermoproteota (GTDB),[60] „Filarchaeota“ Cavalier-Smith 2014[61])
- Supergruppe „Asgardarchaeota“ Violette Da Cunha et al. 2017 (Asgard-Gruppe)[62]
Systematik ab 2024
Um der hohen Diversität der Archaeen (wie auch der Bakterien) zu entsprechen, wurden seit dem Jahr 2024 von der List of Prokaryotic names with Standing in Nomenclature (LPSN) obige Supergruppen und -phyla als neue Prokaryoten-Reiche (englisch kingdoms) eingeführt[63][64] (eine ähnliche Megataxonomie war bereits einige Jahre zuvor vom International Committee on Taxonomy of Viruses (ICTV) für Viren eingeführt worden); das National Center for Biotechnology Information (NCBI) ist diesen Vorgaben gefolgt.[65][66][67]
Domäne Archaea Woese et al. 2024. [bzw. „Archaea“ Woese et al. 1990, „Archaebiota“ Luketa 2012, „Archaebacteria“ Woese et al.. „Mendosicutes“ Gibbons & Murray 1978] – Typusgattung: Methanobacterium Kluyver & van Niel 1936
- Reich Methanobacteriati (Garrity & Holt 2023) Oren & Göker 2024 [„Euryarchaeida“ Luketa 2012, früher: Superphylum Euryarchaeota s. l.]
- Phylum „Candidatus Hadarchaeota“ Chuvochina et al. 2019 bzw. 2023 [„Ca. Hadesarchaeota“ Da Cunha et al. 2017]
- Phylum „Candidatus Hydrothermarchaeota“ Jungbluth et al. 2017
- Phylum Methanobacteriota Garrity & Holt 2023 [bzw. „Methanobacteriota“ Whitman et al. 2018, „Methanobacteraeota“ Oren et al. 2015, „Euryarchaeota“ Garrity & Holt 2001, „Halobacteriota“ Rinke et al. 2021]
- Phylum „Candidatus Poseidoniota“ corrig.Rinke et al. 2019 [„Ca. Thermoplasmatota“ Rinke et al. 2019 bzw. „Thermoplasmatota“ Rinke et al. 2021 oder Thermoplasmatota Chuvochina et al. 2024]
- Reich Nanobdellati Rinke et al. 2024 [früher provisorisch DPANN-Archaeen]
- Phylum „Candidatus Aenigmatarchaeota“ corrig.Rinke et al. 2013 [„Ca. Aenigmarchaeota“ Rinke et al. 2013]
- Phylum „Candidatus Altarchaeota“ corrig.Seitz et al. 2016 [„Ca. Altiarchaeota“ Seitz et al. 2016, „Ca. Altarchaeia“ Dong et al. 2022]
- Phylum „Candidatus Huberarchaeota“ corrig.Probst et al. 2018 [„Ca. Huberarchaea“ Probst et al. 2018]
- Phylum „Candidatus Iainarchaeota“ corrig.Rinke et al. 2013 [„Ca. Diapherotrites“ Rinke et al. 2013]
- Phylum „Candidatus Micrarchaeota“ Baker & Dick 2013
- Phylum Microcaldota Sakai et al. 2023
- Phylum Nanobdellota Huber et al. 2023 [„Nanoarchaeota“ Huber et al. 2002]
- Phylum „Candidatus Nanohalarchaeota“ corrig.Rinke et al. 2013 [„Ca. Nanohaloarchaeota“ Rinke et al. 2013]
- Phylum „Candidatus Parvarchaeota“ Rinke et al. 2013
- Phylum „Candidatus Undinarchaeota“ Dombrowski et al. 2020
- Reich Promethearchaeati Imachi et al. 2024 [früher Asgard-Archaeen]
- Phylum Promethearchaeota Imachi et al. 2024 [„Ca. Lokiarchaeota“ corrig.Spang et al. 2015, bzw. „Ca. Lokiarchaeia“ Spang et al. 2015, „Ca. Lokiarchaeia“ Bulzu et al. 2019 — „Candidatus Baldrarchaeia“ Liu et al. 2021, „Ca. Borrarchaeota“ Liu et al. 2021, „Ca. Hermodarchaeota“ Liu et al. 2021, „Ca. Njordarchaeota“ Xie et al. 2022, „Ca. Odinarchaeota“ Zaremba-Niedzwiedzka et al. 2017, „Ca. Wukongarchaeota“ Liu et al. 2021]
- Reich Thermoproteati Guy & Ettema 2024 [„Crenarchaeida“ Luketa 2012, „Korarchaeota“ Barns et al. 1996, „Proteoarchaeota“ Petitjean et al. 2014, früher TACK-Superphylum]
- Phylum „Candidatus Augarchaeota“ corrig.Nunoura et al. 2011 [„Ca. Aigarchaeota“ Nunoura et al. 2011]
- Phylum „Candidatus Nezhaarchaeota“ Wang et al. 2019
- Phylum Thermoproteota Garrity & Holt 2021 [bzw. „Thermoproteota“ Whitman et al. 2018, „Thermoproteaeota“ Oren et al. 2015, „Crenarchaeota“ Garrity & Holt 2001 — „Ca. Geothermarchaeota“ Jungbluth et al. 2017, „Ca. Korarchaeota“ Ludwig & Klenk 2001, „Ca. Geothermarchaeum“ Adam et al. 2022, „Nitrososphaeraeota“ Oren et al. 2015 bzw. Nitrososphaerota Brochier-Armanet et al. 2021 oder „Nitrososphaerota“ Whitman et al. 2018, „Ca. Thaumarchaeota“ Brochier-Armanet et al. 2008, „Ca. Methanomethylicota“ corrig.Vanwonterghem et al. 2016, „Ca. Verstraetearchaeota“ Vanwonterghem et al. 2016]
- Archaeen ohne Zuordnung zu einem Reich
- Phylum „Candidatus Freyrarchaeota“ Xie et al. 2022
- Phylum „Candidatus Geoarchaeota“ Kozubal et al. 2013
- Phylum „Candidatus Hodarchaeota“ Liu et al. 2021
- Phylum „Candidatus Kariarchaeota“ Liu et al. 2021
- Phylum „Candidatus Sigynarchaeota“ Xie et al. 2022
- Klade Sukuna-clade
- Archaeen ohne Zuordnung zu einem Phylum oder einer Klade
- …
Anmerkungen:
- Dass einige Kandidatenphyla noch ohne Zuordnung zu einem der obigen Reiche sind, bedeutet nicht unbedingt, dass sie abseits dieser Großgruppen stehen, vorgeschlagene Zugehörigkeiten (etwa zu Asgard oder TACK) sind i. d. R. lediglich noch nicht offiziell anerkannt.
- Manche Taxa kommen mit gleicher Bezeichnung mehrfach vor, teilweise in Anführungszeichen oder als Kandidaten, mit unterschiedlicher Autorenschaft. Das liegt daran, dass Archaeengruppen in der Regel zunächst durch die Metagenomik und ähnliche Verfahren identifiziert wurden und die betreffenden Taxa erst später durch nach eine Kultivierung ihre gültige Veröffentlichung bekamen.
- Die vielen Synonyme einiger Phyla (Promethearchaeota, Thermoproteota) resultieren u. a. daher, dass einige Mitgliedskladen (Verwandtschaftsgruppen) früher im Rang eines Phylums gesehen wurden, und ihr Bezeichner ein entsprechendes Suffix (‚-ota‘, meist ‚-archaeota‘) trug. Diese Kladen werden heute oft als Klassen (mit einem entsprechenden neuen Suffix, etwa ‚-archaeia‘) im jeweiligen Phylum gesehen. Die alten Bezeichner auf der Rang-Ebene der Phyla werden damit formal zu Synonymen für das heutige Phylum. Die inhaltliche Entsprechung ist aber die eines Subsets. In der obigen Liste sind diese formalen Synonyme von den inhaltlichen durch einen Bindestrich (‚—‘) abgesetzt. Der Wortteil ‚bacter‘ ist eine Reminiszenz an die Zeit, in der die Archaeen noch als eine Gruppe der Bakterien angesehen wurden, er leitet sich üblicherweise von der Typusgattung ab.
- Die Reiche Thermoproteati (TACK-Superphylum) und Promethearchaeati (Asgard-Archaeen) werden von einigen Autoren in einer gemeinseman Klade, der Supergruppe „Proteoarchaea“ gesehen (auch „Proteoarchaeota“ genannt).
Archaeen und Menschen
Archaeen wurden beim Menschen im Darm (Colon), in der Mundhöhle (Zahnflora), im Bauchnabel[68] und in der Vagina nachgewiesen.[69] Archaeen machen etwa 10 % der anaeroben Gemeinschaft im menschlichen Darm aus.[70] Im Darm treten vor allem Archaeen auf, die der Gattung Methanobrevibacter zugehören, im Speziellen Methanobrevibacter smithii. Diese zählen zu den methanogenen Archaea. Nicht bei allen Menschen kommt M. smithii im Darm vor, und bei Säuglingen unter zwei Jahren wurden bisher noch nie Archaeen identifiziert. In einer Studie wurden Archaeen auch auf der Haut nachgewiesen, die zum Phylum Thaumarchaeota gehören.[71] Möglicherweise korreliert die Anzahl jener Archaeen mit der Häufigkeit des Schwitzens.
Methanogene der Arten Methanobrevibacter smithii und Methanosphaera stadtmanae leben vergesellschaftet mit syntrophen Bakterien im menschlichen Verdauungstrakt, sodass sie einen Einfluss auf die Verdauung ausüben.[72] Diese nutzen die beiden Produkte bakterieller Gärungen, Wasserstoff und Formiat, für die Methanogenese. Eine hohe Konzentration an Wasserstoff hemmt die ATP-Erzeugung anderer Bakterien. M. smithii baut unter Methanbildung auch Methanol ab, das für den Menschen toxisch ist. Daher haben die Methanogenen einen positiven Einfluss auf die menschliche Darmflora. Ob diese auch beeinflussen, wie viel Energie der Mensch aus der Nahrung aufnehmen kann, ist noch Gegenstand der Forschung.
Obwohl Archaeen in engem Kontakt zum Menschen stehen, gibt es keinen Hinweis auf humanpathogene Arten.[70][73] Es wurde aber eine Korrelation zwischen Erkrankung und Anzahl von methanogenen Archaeen nachgewiesen: Je mehr Archaeen beispielsweise im (entzündeten) Zahnfleisch vorhanden waren, desto stärker war eine entsprechende Parodontitis ausgeprägt. Hierbei treten insbesondere Archaeen der Art Methanobrevibacter oralis auf. Auch bei Patienten mit Darmkrebs bzw. Divertikulose war die Menge methanogener Archaeen in jenen Bereichen erhöht. Dennoch tragen diese Archaeen nur indirekt zur Erkrankung bei, indem sie das Wachstum echt pathogener Bakterien fördern – die Archaeen selbst sind es nicht. Wenn man Archaeen als „Kopathogene“ oder „Pathobionten“[74] (Symbionten, die unter bestimmten Bedingungen pathologisch werden) betrachtet, dann könnte die Erkrankung mit Medikationen therapiert werden, die diese Archaeen zum Ziel haben. So inhibieren beispielsweise Statine das Wachstum der bei einer Parodontitis vergesellschafteten Methanobrevibacter oralis.[71]
Warum die bekannten Archaeen nicht humanpathogen sind, ist noch nicht eindeutig geklärt. Auch wenn unter den Archaeen keine menschlichen Parasiten bekannt sind, so gibt es unter den Nanobdellati (DPANN-Archaeen) zumindest einige kleine Vertreter, die als Epibionten (Parasiten?) auf größeren Archaeen leben (etwa Nanoarchaeum equitans). Dagegen scheint Ca. Sukunaarchaeum mirabile ein echter Parasit zu sein. Das Fehlen vieler archaeenspezifischer Kofaktoren und Vitamine im Menschen ist nicht notwendigerweise die Ursache für das Nichtvorkommen humanpathogener Archaeen.[75][76] Selbst dass die durch pathogene Prozesse erzeugten Mikrohabitate besetzt werden, ist kein Alleinstellungsmerkmal der Archaeen – prinzipiell könnten auch Organismen mit ähnlichem (anaeroben, hydrogenotrophen) Stoffwechsel diese Habitate nutzen.[74]
Biotechnologisches Potential
Archaeelle Stoffwechselleistungen, Zellbestandteile oder Enzyme werden industriell angewendet. Vor allem die Extremophilen besitzen viele Eigenschaften, die sich biotechnologisch nutzen lassen. Einige Beispiele, die sich bereits in der Entwicklungsphase oder Anwendung befinden:
- Biotechnologie
- Biogas-Gewinnung (Methangewinnung in Biogasanlagen)
- Mikrobielle Erzlaugung („microbial ore leaching“ oder „bioleaching“): Bei diesem Prozess werden niederwertige, sulfidische Erze ausgelaugt (die Sulfidanteile werden mikrobiell zu Sulfat oxidiert und dadurch die Schwermetalle in einen löslichen Zustand überführt); dies wird zum Beispiel zur Gewinnung von Kupfer, Zink und Nickel angewendet.
- Recycling: Kunststoff-Zersetzung zu wiederverwertbaren Kunststoff-Rohstoffen (i.e. Monomeren) dank in Archaeen vorhandener Enzyme[77]
- Medizin: Verwendung von Zellwandbestandteilen (sogenannte S-Layer) als Träger für Impfstoffe
- Nanotechnologie
- Verwendung der S-Layer für die Ultrafiltration
- Bacteriorhodopsin/Purpurmembran phototropher halophiler Archaeen als Biosensoren
- Biologie
- Gewinnung hitzeresistenter Enzyme, z. B. α-Amylasen, proteolytische Enzyme, DNA-Polymerasen
- Gewinnung neuer Restriktionsenzyme
- Boden- und Gewässersanierung
Literatur
Archaeen in Standardwerken
- Georg Fuchs (Hrsg.): Allgemeine Mikrobiologie. (begr. von Hans G. Schlegel). 8. Auflage. Georg Thieme Verlag, Stuttgart/New York 2007, ISBN 978-3-13-444608-1 (englisch).
- Martin Dworkin, Stanley Falkow, Eugene Rosenberg, Karl-Heinz Schleifer, Erko Stackebrandt (Hrsg.) The Prokaryotes, A Handbook of the Biology of Bacteria. 7 Bände, 3. Auflage. Springer-Verlag, New York u. a., 2006, ISBN 0-387-30740-0 (englisch).
- Joseph W. Lengeler, Gerhart Drews, Hans G. Schlegel (Hrsg.) Biology of the Prokaryotes. Georg Thieme Verlag, Stuttgart 1999, ISBN 3-13-108411-1 (englisch).
- H. König: Archaea. In: Encyclopedia of Life Sciences. John Wiley & Sons, 2003; doi:10.1038/npg.els.0000443 (englisch).
- Michael T. Madigan, John M. Martinko, Thomas Lazar (Übersetzer) und Freya Thomm-Reitz (Übersetzer): Brock Mikrobiologie. 11., aktualisierte Auflage. Pearson Studium, 2009, ISBN 978-3-8273-7358-8 (englisch).
Beschreibungen in der wissenschaftlichen Literatur
- Carl R. Woese, Otto Kandler, Mark L. Wheelis: Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya. In: PNAS, Band 87, Nr. 12, 15. Juni 1990, S. 4576-4579; doi:10.1073/pnas.87.12.4576, PMC 54159 (freier Volltext), PMID 2112744, PDF (freier Volltextzugriff, englisch)
- B. M. Karner, E. F. DeLong, D. M. Karl: Archaeal dominance in the mesopelagic zone of the Pacific ocean. In: Nature. Band 409(6819), 2001, S. 507–510. PMID 11206545; doi:10.1038/35054051
- R. Cavicchioli: Cold-adapted archaea. In: Nat Rev Microbiol. 4(5), 2006, S. 331–343; doi:10.1038/nrmicro1390, PMID 16715049 (englisch).
- H. Huber, M. J. Hohn, R. Rachel, T. Fuchs, V. C. Wimmer, K. O. Stetter. A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont. In: Nature. Band 417(6884), 2002, S. 63–67; doi:10.1038/417063a, PMID 11986665 (englisch).
- E. Conway de Macario, Alberto J. L. Macario: Methanogenic archaea in health and disease: a novel paradigm of microbial pathogenesis. In: Int J Med Microbiol. Band 299, Nr. 2, 2009, S. 99–108; doi:10.1016/j.ijmm.2008.06.011, PMID 18757236 (englisch).
- R. Cavicchioli: Archaea - timeline of the third domain. In: Nat Rev Microbiol. 9(1), 2011, S. 51–61. PMID 21132019; doi:10.1038/nrmicro2482 (englisch)
- R. E. Valas, P. E. Bourne: The origin of a derived superkingdom: how a gram-positive bacterium crossed the desert to become an archaeon. In: Biology direct. Band 6, 2011, S. 16, ISSN 1745-6150; doi:10.1186/1745-6150-6-16, PMC 3056875 (freier Volltext), PMID 21356104 (englisch).
- Hans-Peter Horz: Archaeal Lineages within the Human Microbiome: Absent, Rare or Elusive? In: Life (Basel). Band 5, Nr. 2, 2015, S. 1333–1345; doi:10.3390/life5021333 (englisch).
Weblinks
- Archaeen wandeln in sauren Böden Ammonium um, Auf Pflanzenforschung.de vom 20. Dezember 2011: Archaeen oxidieren in sauren Böden Ammoniak zu Nitrat
- Nadja Podbregar: Unser Ur-Urahn war ein Archaeon. Auf wissenschaft.de vom 6. Mai 2015
- Josephine Franke: Mikrobiom des Darms ist diverser als gedacht – Archaeen machen 1,2 Prozent der mikrobiellen Darmbesiedelung aus. Auf scinexx.de vom 18. Januar 2022
- Chris Rinke: Behind the Paper: Standardising archaeal taxonomy, Nature Microbiology Community, 21. Juni 2021
- SIB: Archaeal cell – Archaeenzelle, Interaktive Graphik von SwissBioPics
- Christa Schleper, German Jurgens, Melanie Jonuscheit: Genomic studies of uncultivated archaea. In: Nat Rev Microbiol. 3. Jahrgang, Nr. 6, Juni 2005, S. 479–488, doi:10.1038/nrmicro1159, PMID 15931166 (englisch).
- Cindy J. Castelle, Jillian F. Banfield: Major New Microbial Groups Expand Diversity and Alter our Understanding of the Tree of Life. In: Cell, Band 172, Nr. 6, S. 1181–1197; doi:10.1016/j.cell.2018.02.016, PMID 29522741 (englisch).
- Chris Greening, Princess R. Cabotaje, Luis E. Valentin Alvarado, Pok Man Leung, Henrik Land, Thiago Rodrigues-Oliveira, Rafael I. Ponce-Toledo, Moritz Senger, Max A. Klamke, Michael Milton, Rachael Lappan, Susan Mullen, Jacob West-Roberts, Jie Mao, Jiangning Song, Marie Schoelmerich, Courtney W. Stairs, Christa Schleper, Rhys Grinter, Anja Spang, Jillian F. Banfield, Gustav Berggren: Minimal and hybrid hydrogenases are active from archaea. In: Cell, Band 187, Nr. 13, S. 3357-3372.e19, 20. Juni 2024; doi:10.1016/j.cell.2024.05.032, Epub 11. Juni 2024 (englisch). Dazu:
Einzelnachweise
- ↑ Merriam-Webster Online Dictionary
- ↑ A. L. Hartman et al.: The Complete Genome Sequence of Haloferax volcanii DS2, a Model Archaeon. In: PLOS ONE. Band 5, Nr. 3, 2010, S. e9605, PMC 2841640 (freier Volltext) – (englisch).
- ↑ Christa Schleper et al.: A multicopy plasmid of the extremely thermophilic archaeon „Sulfolobus“ effects its transfer to recipients by mating. In: J. Bacteriol. Band 177, 1995, S. 4417–4426. PMID 7635827.
- ↑ A. Pearson et al.: Factors Controlling the Distribution of Archaeal Tetraethers in Terrestrial Hot Springs. In: Appl. Environ. Microbiol. Band 74, 2008, S. 3523–3532, als (PDF)
- ↑ E. Boyd et al.: The role of tetraether lipid composition in the adaptation of thermophilic archaea to acidity. In: Front Microbiol. Band 4, Nr. 62, April 2013, doi:10.3389/fmicb.2013.00062, PMC 3615187 (freier Volltext) – (englisch).
- ↑ Bastian Herzog, Reinhard Wirth: Swimming Behavior of Selected Species of Archaea. In: Applied and Environmental Microbiology. Band 78, Nr. 6, 2012, S. 1670–1674; doi:10.1128/AEM.06723-11 (englisch).
idw-online.de vom 28. Februar 2012: Archaeen sind die schnellsten Lebewesen der Welt – Mikroorganismen glänzen mit Weltrekord. - ↑ T. Urich et al.: The sulphur oxygenase reductase from Acidianus ambivalens is a multimeric protein containing a low-potential mononuclear non-haem iron centre. In: Biochem J. Band 381, Teil 1, 2004, S. 137–146. doi:10.1042/BJ20040003
- ↑ a b Thorsten Allers, Moshe Mevarech: Archaeal genetics — the third way. In: Nature Reviews Genetics. Band 6, Nr. 1, Januar 2005, ISSN 1471-0064, S. 58–73, doi:10.1038/nrg1504 (nature.com [abgerufen am 4. Juni 2024]).
- ↑ Catherina Hildenbrand, Tilmann Stock, Christian Lange, Michael Rother, Jörg Soppa: Genome Copy Numbers and Gene Conversion in Methanogenic Archaea. In: Journal of Bacteriology. Band 193, Nr. 3, Februar 2011, ISSN 0021-9193, S. 734–743, doi:10.1128/JB.01016-10, PMID 21097629, PMC 3021236 (freier Volltext).
- ↑ James E. Galagan, Chad Nusbaum, Alice Roy, Matthew G. Endrizzi, Pendexter Macdonald, Will FitzHugh, Sarah Calvo, Reinhard Engels, Serge Smirnov, Deven Atnoor, Adam Brown, Nicole Allen, Jerome Naylor, Nicole Stange-Thomann, Kurt DeArellano, Robin Johnson, Lauren Linton, Paul McEwan, Kevin McKernan, Jessica Talamas, Andrea Tirrell, Wenjuan Ye, Andrew Zimmer, Robert D. Barber, Isaac Cann, David E. Graham, David A. Grahame, Adam M. Guss, Reiner Hedderich, Cheryl Ingram-Smith, H. Craig Kuettner, Joseph A. Krzycki, John A. Leigh, Weixi Li, Jinfeng Liu, Biswarup Mukhopadhyay, John N. Reeve, Kerry Smith, Timothy A. Springer, Lowell A. Umayam, Owen White, Robert H. White, Everly Conway de Macario, James G. Ferry, Ken F. Jarrell, Hua Jing, Alberto J. L. Macario, Ian Paulsen, Matthew Pritchett, Kevin R. Sowers, Ronald V. Swanson, Steven H. Zinder, Eric Lander, William W. Metcalf, Bruce Birren: The Genome of M. acetivorans Reveals Extensive Metabolic and Physiological Diversity. In: Genome Research. Band 12, Nr. 4, 1. April 2002, ISSN 1088-9051, S. 532–542, doi:10.1101/gr.223902, PMID 11932238 (cshlp.org [abgerufen am 4. Juni 2024]).
- ↑ Elizabeth Waters, Michael J. Hohn, Ivan Ahel, David E. Graham, Mark D. Adams, Mary Barnstead, Karen Y. Beeson, Lisa Bibbs, Randall Bolanos, Martin Keller, Keith Kretz, Xiaoying Lin, Eric Mathur, Jingwei Ni, Mircea Podar, Toby Richardson, Granger G. Sutton, Melvin Simon, Dieter Soll, Karl O. Stetter, Jay M. Short, Michiel Noordewier: The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism. In: Proceedings of the National Academy of Sciences of the United States of America. Band 100, Nr. 22, 28. Oktober 2003, ISSN 0027-8424, S. 12984–12988, doi:10.1073/pnas.1735403100, PMID 14566062.
- ↑ C Schleper, I Holz, D Janekovic, J Murphy, W Zillig: A multicopy plasmid of the extremely thermophilic archaeon Sulfolobus effects its transfer to recipients by mating. In: Journal of Bacteriology. Band 177, Nr. 15, August 1995, ISSN 0021-9193, S. 4417–4426, doi:10.1128/jb.177.15.4417-4426.1995, PMID 7635827, PMC 177192 (freier Volltext).
- ↑ Sota M, Top EM: Plasmids: Current Research and Future Trends. Caister Academic Press, 2008, abgerufen am 4. Juni 2024 (englisch).
- ↑ David E. Graham, Ross Overbeek, Gary J. Olsen, Carl R. Woese: An archaeal genomic signature. In: Proceedings of the National Academy of Sciences. Band 97, Nr. 7, 28. März 2000, ISSN 0027-8424, S. 3304–3308, doi:10.1073/pnas.97.7.3304.
- ↑ Terry Gaasterland: Archaeal genomics. In: Current Opinion in Microbiology. Band 2, Nr. 5, Oktober 1999, ISSN 1369-5274, S. 542–547, doi:10.1016/s1369-5274(99)00014-4 (sciencedirect.com [abgerufen am 4. Juni 2024]).
- ↑ Terry Gaasterland: Archaeal genomics. In: Current Opinion in Microbiology. Band 2, Nr. 5, Oktober 1999, ISSN 1369-5274, S. 542–547, doi:10.1016/s1369-5274(99)00014-4 (sciencedirect.com [abgerufen am 4. Juni 2024]).
- ↑ Patrick P Dennis: Ancient Ciphers: Translation in Archaea. In: Cell. Band 89, Nr. 7, Juni 1997, S. 1007–1010, doi:10.1016/S0092-8674(00)80288-3 (elsevier.com [abgerufen am 4. Juni 2024]).
- ↑ Finn Werner: Structure and function of archaeal RNA polymerases. In: Molecular Microbiology. Band 65, Nr. 6, September 2007, ISSN 0950-382X, S. 1395–1404, doi:10.1111/j.1365-2958.2007.05876.x.
- ↑ L Aravind: DNA-binding proteins and evolution of transcription regulation in the archaea. In: Nucleic Acids Research. Band 27, Nr. 23, 1. Dezember 1999, S. 4658–4670, doi:10.1093/nar/27.23.4658, PMID 10556324, PMC 148756 (freier Volltext).
- ↑ J Lykke-Andersen: Archaeal introns: splicing, intercellular mobility and evolution. In: Trends in Biochemical Sciences. Band 22, Nr. 9, September 1997, ISSN 0968-0004, S. 326–331, doi:10.1016/s0968-0004(97)01113-4 (cell.com [abgerufen am 4. Juni 2024]).
- ↑ Yoh-ichi Watanabe, Shin-ichi Yokobori, Toshiro Inaba, Akihiko Yamagishi, Tairo Oshima, Yutaka Kawarabayasi, Hisasi Kikuchi, Kiyoshi Kita: Introns in protein‐coding genes in Archaea. In: FEBS Letters. Band 510, Nr. 1–2, 2. Januar 2002, ISSN 0014-5793, S. 27–30, doi:10.1016/S0014-5793(01)03219-7.
- ↑ Shigeo Yoshinari, Takashi Itoh, Steven J. Hallam, Edward F. DeLong, Shin-ichi Yokobori, Akihiko Yamagishi, Tairo Oshima, Kiyoshi Kita, Yoh-ichi Watanabe: Archaeal pre-mRNA splicing: A connection to hetero-oligomeric splicing endonuclease. In: Biochemical and Biophysical Research Communications. Band 346, Nr. 3, August 2006, ISSN 0006-291X, S. 1024–1032, doi:10.1016/j.bbrc.2006.06.011 (sciencedirect.com [abgerufen am 4. Juni 2024]).
- ↑ Ilan Rosenshine, Ronen Tchelet, Moshe Mevarech: The Mechanism of DNA Transfer in the Mating System of an Archaebacterium. In: Science. Band 245, Nr. 4924, 22. September 1989, ISSN 0036-8075, S. 1387–1389, doi:10.1126/science.2818746.
- ↑ Sabrina Fröls, Malgorzata Ajon, Michaela Wagner, Daniela Teichmann, Behnam Zolghadr, Mihaela Folea, Egbert J. Boekema, Arnold J. M. Driessen, Christa Schleper, Sonja‐Verena Albers: UV‐inducible cellular aggregation of the hyperthermophilic archaeon Sulfolobus solfataricus is mediated by pili formation. In: Molecular Microbiology. Band 70, Nr. 4, November 2008, ISSN 0950-382X, S. 938–952, doi:10.1111/j.1365-2958.2008.06459.x.
- ↑ Małgorzata Ajon, Sabrina Fröls, Marleen van Wolferen, Kilian Stoecker, Daniela Teichmann, Arnold J. M. Driessen, Dennis W. Grogan, Sonja-Verena Albers, Christa Schleper: UV-inducible DNA exchange in hyperthermophilic archaea mediated by type IV pili: UV-inducible DNA exchange in hyperthermophilic archaea. In: Molecular Microbiology. Band 82, Nr. 4, November 2011, S. 807–817, doi:10.1111/j.1365-2958.2011.07861.x.
- ↑ a b Sabrina Fröls, Malgorzata Ajon, Michaela Wagner, Daniela Teichmann, Behnam Zolghadr, Mihaela Folea, Egbert J. Boekema, Arnold J. M. Driessen, Christa Schleper, Sonja‐Verena Albers: UV‐inducible cellular aggregation of the hyperthermophilic archaeon Sulfolobus solfataricus is mediated by pili formation. In: Molecular Microbiology. Band 70, Nr. 4, November 2008, ISSN 0950-382X, S. 938–952, doi:10.1111/j.1365-2958.2008.06459.x.
- ↑ a b Małgorzata Ajon, Sabrina Fröls, Marleen van Wolferen, Kilian Stoecker, Daniela Teichmann, Arnold J. M. Driessen, Dennis W. Grogan, Sonja-Verena Albers, Christa Schleper: UV-inducible DNA exchange in hyperthermophilic archaea mediated by type IV pili: UV-inducible DNA exchange in hyperthermophilic archaea. In: Molecular Microbiology. Band 82, Nr. 4, November 2011, S. 807–817, doi:10.1111/j.1365-2958.2011.07861.x.
- ↑ Sabrina Fröls, Malcolm F. White, Christa Schleper: Reactions to UV damage in the model archaeon Sulfolobus solfataricus. In: Biochemical Society Transactions. Band 37, Nr. 1, 20. Januar 2009, ISSN 0300-5127, S. 36–41, doi:10.1042/bst0370036 (portlandpress.com [abgerufen am 4. Juni 2024]).
- ↑ Harris Bernstein, Carol Bernstein: Sexual Communication in Archaea, the Precursor to Eukaryotic Meiosis. In: Biocommunication of Archaea. Springer International Publishing, Cham 2017, ISBN 978-3-319-65536-9, S. 103–117, doi:10.1007/978-3-319-65536-9_7.
- ↑ Krieg N: Bergey's Manual of Systematic Bacteriology. (PDF) In: ResearchGate. Springer, 2005, S. 21–26, abgerufen am 4. Juni 2024 (englisch).
- ↑ a b Rolf Bernander: Archaea and the cell cycle. In: Molecular Microbiology. Band 29, Nr. 4, August 1998, ISSN 0950-382X, S. 955–961, doi:10.1046/j.1365-2958.1998.00956.x.
- ↑ L KELMAN, Z KELMAN: Multiple origins of replication in archaea. In: Trends in Microbiology. Band 12, Nr. 9, September 2004, ISSN 0966-842X, S. 399–401, doi:10.1016/j.tim.2004.07.001 (cell.com [abgerufen am 4. Juni 2024]).
- ↑ Ann-Christin Lindås, Erik A. Karlsson, Maria T. Lindgren, Thijs J. G. Ettema, Rolf Bernander: A unique cell division machinery in the Archaea. In: Proceedings of the National Academy of Sciences. Band 105, Nr. 48, 2. Dezember 2008, ISSN 0027-8424, S. 18942–18946, doi:10.1073/pnas.0809467105.
- ↑ Rachel Y. Samson, Takayuki Obita, Stefan M. Freund, Roger L. Williams, Stephen D. Bell: A Role for the ESCRT System in Cell Division in Archaea. In: Science. Band 322, Nr. 5908, 12. Dezember 2008, ISSN 0036-8075, S. 1710–1713, doi:10.1126/science.1165322 (englisch).
- ↑ Erik A. Pelve, Ann‐Christin Lindås, Willm Martens‐Habbena, José R. de la Torre, David A. Stahl, Rolf Bernander: Cdv‐based cell division and cell cycle organization in the thaumarchaeon Nitrosopumilus maritimus. In: Molecular Microbiology. Band 82, Nr. 3, November 2011, ISSN 0950-382X, S. 555–566, doi:10.1111/j.1365-2958.2011.07834.x.
- ↑ Yaron Caspi, Cees Dekker: Dividing the Archaeal Way: The Ancient Cdv Cell-Division Machinery. In: Frontiers in Microbiology. Band 9, 2. März 2018, ISSN 1664-302X, doi:10.3389/fmicb.2018.00174, PMID 29551994, PMC 5840170 (freier Volltext).
- ↑ Rob U. Onyenwoke, Julia A. Brill, Kamyar Farahi, Juergen Wiegel: Sporulation genes in members of the low G+C Gram-type-positive phylogenetic branch (Firmicutes). In: Archives of Microbiology. Band 182, Nr. 2, 1. Oktober 2004, ISSN 1432-072X, S. 182–192, doi:10.1007/s00203-004-0696-y.
- ↑ N. A. Kostrikina, I. S. Zvyagintseva, V. I. Duda: Cytological peculiarities of some extremely halophilic soil archaeobacteria. In: Archives of Microbiology. Band 156, Nr. 5, 1. Oktober 1991, ISSN 1432-072X, S. 344–349, doi:10.1007/BF00248708.
- ↑ Hulcr J, Latimer AM, Henley JB, Rountree NR, Fierer N, et al. (2012) A Jungle in There: Bacteria in Belly Buttons are Highly Diverse, but Predictable. PLoS ONE 7(11): e47712. doi:10.1371/journal.pone.0047712
- ↑ Carl R. Woese, G. E. Fox: Phylogenetic structure of the prokaryotic domain: the primary kingdoms. In: Proc. Natl. Acad. Sci. U.S.A. Band 74, Heft 11, 1977, S. 5088–5090; PMID 270744 (englisch).
- ↑ Carl R. Woese et al.: Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya. In: Proc. Natl. Acad. Sci. U.S.A. Band 87, Heft 12, 1990, S. 4576–4579; PMID 2112744 (englisch).
- ↑ J. D. Trent et al.: Chaperonin filaments: the archaeal cytoskeleton? In: PNAS. Band 94, Nr. 10, 1997, S. 5383–5388. PMID 9144246; PDF (freier Volltextzugriff, engl.)
- ↑ F. Hara et al.: An actin homolog of the archaeon Thermoplasma acidophilum that retains the ancient characteristics of eukaryotic actin. In: J. Bacteriol. Band 189, Nr. 5, 2007, S. 2039–2045; PMID 17189356; PDF (freier Volltextzugriff, englisch).
- ↑ B. J. Tindall et al.: Valid publication of names of prokaryotes according to the rules of nomenclature: past history and current practice. In: Int J Syst Evol Microbiol. Band 56, Heft 11, 2006, S. 2715–2720. PMID 17082418. doi:10.1099/ijs.0.64780-0.
- ↑ International Journal of Systematic and Evolutionary Microbiology. (IJSEM)
- ↑ J. P. Euzéby: List of bacterial names with standing in nomenclature: a folder available on the Internet. In: Int. J. Syst. Bacteriol. Band 47, 1997, S. 590–592. PMID 9103655 -List of Prokaryotic names with Standing in Nomenclature ( vom 30. Dezember 2010 im Internet Archive) (LPSN)
- ↑ S. P. Lapage et al.: International Code of Nomenclature of Bacteria, 1990 Revision. (Bacteriological Code). ASM Press, Washington, D.C. 1992, ISBN 1-55581-039-X.
- ↑ C. R. Woese et al.: A phylogenetic definition of the major eubacterial taxa. In: Syst. Appl. Microbiol. Band 6, 1985, S. 143–151. PMID 11542017.
- ↑ Bergey’s Manual Trust, Department of Microbiology, 527 Biological, Sciences Building, University of Georgia, Athens, GA 30602-2605, USA - Bergey’s Manual
- ↑ George M. Garrity et al.: Taxonomic Outline of the Bacteria and Archaea. Release 7.7, 6. März 2007, Michigan State University Board of Trustees, www.taxonomicoutline.org
- ↑ W. Ludwig et al.: ARB: a software environment for sequence data. In: Nucleic Acids Research, Band 32, 2004, S. 1363–1371 (englisch).
- ↑ E. Pruesse et al.: SILVA: a comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB. In: Nucleic Acids Research, Band 35, Nr. 21, 2007, S. 7188–7196 (englisch).
- ↑ P. Yarza et al.: The All-Species Living Tree Project: a 16S rRNA-based phylogenetic tree of all sequenced type strains. In: Syst. Appl. Microbiol. 2008; doi:10.1016/j.syapm.2008.07.001 (englisch).
- ↑ H. Huber et al.: A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont. In: Nature, Band 417, 2002, S. 63–67; PMID 11986665 (englisch).
- ↑ E. Waters et al.: The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism. In: Proc Natl Acad Sci U.S.A. 100, 2003, S. 12984–12988; PMID 14566062 (englisch).
- ↑ S. M. Barns et al.: Perspectives on archaeal diversity, thermophily and monophyly from environmental rRNA sequences. In: Proc Natl Acad Sci U.S.A. 93, 1996, S. 9188–9193; PMID 8799176 (englisch).
- ↑ J. G. Elkins et al.: A korarchaeal genome reveals insights into the evolution of the Archaea. In: Proc Natl Acad Sci U.S.A. 105, 2008, S. 8102–8107; PMID 18535141 (englisch).
- ↑ P. De Vos, H. G. Trüper: Judicial Commission of the International Committee on Systematic Bacteriology. IXth International (IUMS) Congress of Bacteriology and Applied Microbiology. Minutes of the meetings, 14, 15 and 18 August 1999, Sydney, Australia. In: Int. J. Syst. Evol. Microbiol. Band 50, 2000, S. 2239–2244 (englisch).
- ↑ a b Taxa above the rank of class. List of Prokaryotic names with Standing in Nomenclature (LPSN, bacterio.net). Memento im Webarchiv vom 8. August 2017 (englisch).
- ↑ GTDB: Thermoproteota (phylum).
- ↑ Thomas Cavalier-Smith: The neomuran revolution and phagotrophic origin of eukaryotes and cilia in the light of intracellular coevolution and a revised tree of life. In: Cold Spring Harb. Perspect. Biol. 6. Jahrgang, Nr. 9, 2014, S. a016006, doi:10.1101/cshperspect.a016006, PMID 25183828, PMC 4142966 (freier Volltext) – (englisch).
- ↑ Paul-Adrian Bulzu, Adrian-Stefan Andrei, Michaela M. Salcher, Maliheh Mehrshad, Keiichi Inoue, Hideki Kandori, Oded Beja, Rohit Ghai, Horia L. Banciu: Casting light on Asgardarchaeota metabolism in a sunlit microoxic niche. In: Nature Microbiology, Band 4, 1. April 2019, S. 1129–1137, doi:10.1038/s41564-019-0404-y (englisch).
- ↑ LPSN: Domain Bacteria Name: Bacteria Woese et al. 2024.
- ↑ LPSN: Domain Bacteria Name: Archaea Woese et al. 2024.
- ↑ NCBI Taxonomy Browser: Bacteria, Details: Bacteria. Bacteria Woese et al. 2024 in [Göker M & Oren A (2024)]. Rank: domain.
- ↑ NCBI Taxonomy Browser: Archaea, Details: Archaea. Archaea Woese et al. 2024 in [Göker M & Oren A (2024)]. Rank: domain.
- ↑ NCBI Taxonomy Browser: Viruses.
- ↑ Souri Somphanith: Plunging into the Unknown: Belly Button Bacteria and You. In: PLOS Blogs.
- ↑ E. Conway de Macario, Alberto J. L Macario: Methanogenic archaea in health and disease: a novel paradigm of microbial pathogenesis. In: Int J Med Microbiol. 299 (2), 2009, S. 99–108. PMID 18757236
- ↑ a b Jennifer Wirth, Mark Young: The intriguing world of archaeal viruses. In: PLOS Pathog. 16. Jahrgang, Nr. 8, 13. August 2020, S. e1008574, doi:10.1371/journal.ppat.1008574, PMID 32790746, PMC 7425843 (freier Volltext) – (englisch).
- ↑ a b Mor N. Lurie-Weinberger und Uri Gophna: Archaea in and on the Human Body: Health Implications and Future Directions. In: PLOS Pathogens. Band 11, Nr. 6, 11. Juni 2015, S. e1004833, doi:10.1371/journal.ppat.1004833, PMID 26066650.
- ↑ Joan L. Slonczewski, John W. Foster: Mikrobiologie: Eine Wissenschaft mit Zukunft. 2. Auflage. Spektrum Akademischer Verlag, Berlin/Heidelberg 2012, ISBN 978-3-8274-2909-4, S. 854.
- ↑ R. Cavicchioli et al.: Pathogenic archaea: do they exist? In: Bioessays. Band 25, Nr. 11, 2003, S. 1119–1128; doi:10.1002/bies.10354, PMID 14579252 (englisch).
- ↑ a b Rustam I. Aminov: Role of archaea in human disease. In: Frontiers in Cellular and Infection Microbiology. Band 3, 1. Januar 2013, doi:10.3389/fcimb.2013.00042, PMID 23964350.
- ↑ W. Martin: Pathogenic archaebacteria: do they not exist because archaebacteria use different vitamins? In: Bioessays, Band 26, Nr. 5, 2004, S. 592–593; PMID 15112239, doi:10.1002/bies.20044.
- ↑ R. Cavicchioli, P. Curmi: Response to William Martin's letter. In: Bioessays, Band 26, Nr. 5, 2004, S. 593; doi:10.1002/bies.20043.
- ↑ Tageszeitung 28. Februar 2024, Ilse Bullerdiek: „Mikrobiologische Forschung: Ur-Enzyme fressen Plastik“
Auf dieser Seite verwendete Medien
Autor/Urheber: (of code) -xfi-, Lizenz: CC BY-SA 3.0
The Wikispecies logo created by Zephram Stark based on a concept design by Jeremykemp.
Autor/Urheber: Carsten Steger, Lizenz: CC BY-SA 4.0
Luftbild der Grand Prismatic Spring im Yellowstone-Nationalpark (Ansicht von Süden)
Autor/Urheber: Angels Tapias. Microscopist: Jeril, Lizenz: CC BY 3.0
Microphotograph of the archaeon Thermococcus gammatolerans
Autor/Urheber: created according information provided in the study, Lizenz: CC BY-SA 4.0
Outline of a Phylogenetic Tree of the Archaea
Archaeal membrane structure Up: archaeal membrane, 1-isoprene sidechain, 2-ether linkage, 3-L-glycerol, 4-phosphate moieties. Medium: bacterial and eukaryan membrane: 5-fatty acid, 6-ester linkage, 7-D-glycerol, 8-phosphate moieties. Bottom: 9-lipid bilayer in bacteria, eukarya and most archaea, 10-lipid monolayer in some archaea.
Autor/Urheber: Maulucioni, Lizenz: CC BY-SA 4.0
Die vielfältige Morphologie von Archaeen veranschaulichende Bildkomposition
Autor/Urheber: Daniel R. Colman, Melody R. Lindsay, Eric S. Boyd, Lizenz: CC BY-SA 4.0
Maximum likelihood phylogenetic reconstruction of archaeal population level MAGs from 'Smokejumper 3' ('SJ3') in Yellowstone National Park, Smokejumper Geyser Basin. Trees representing the major archaeal order- (or class/phylum-) level clades were constructed using up to 104 phylogenetic marker genes (>50% in any genome). Clades are collapsed as triangles with the taxonomic designation provided to the right. The numbers of MAGs that were identified in a specified clade are indicated in parentheses. Clades encompassing MAGs from 'SJ3' that were >50% complete are shown in blue, those with MAGs >75% are shown in red, and those representing multiple MAGs that ranged in completeness from 50 to >75% are shown in purple. The scale bar shows the expected number of substitutions per site. Proposed names have been added for some clades originally identified by acronymes, subsequently.






