Aminopolycarbonsäuren
Die Aminopolycarbonsäuren oder Komplexone sind eine Gruppe von Komplexbildnern, welche aus einem oder mehreren stickstoffhaltigen Gruppen und mehreren Carboxygruppen bestehen.[1] EDTA und NTA werden dabei in den größten Mengen eingesetzt. EDTA ist jedoch nicht biologisch abbaubar[1] und NTA gilt als krebserregend.[2] Daher gewinnen neue Chelatoren wie Tetranatriumiminodisuccinat, Methylglycindiessigsäure und β-Alanindiessigsäure an Bedeutung.
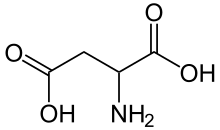
Die natürlichen Aminosäuren Asparaginsäure und Glutaminsäure sind Aminodicarbonsäuren und zählen so auch zu den Aminopolycarbonsäuren.
Sie werden für die Chelatometrie benötigt[3] und in Waschmitteln verwendet.
Natürliche Aminopolycarbonsäuren
Einige Lebewesen verwenden Aminopolycarbonsäuren als Siderophore. So stellen Bakterien (S,S-Enantiomer) und Pilze (R,R-E.) Rhizoferrin und Rhizobactine her. Bei den Süßgräsern nutzt Hafer die Aveninsäure[4] und Gerste die Mugineinsäure[5] um Eisen aus dem Boden aufzunehmen.[6] Bei praktisch allen höheren Pflanzen ist Nicotianamin vorhanden.[7]
- Rhizoferrin
- Rhizobactin DM4
- Rhizobactin 1021
- Mugineinsäure
- Aveninsäure
Diese bilden teilweise deutlich stabilere Komplexe mit Eisen als die synthetischen Aminopolycarbonsäuren.
Synthetische Aminopolycarbonsäuren
 |  | |
| Aminomalonsäure[8] | Iminodiessigsäure (IDA)[9] | Nitrilotriessigsäure (NTA) |
 |  |  |
| Ethylendiamintetraessigsäure (EDTA) | Diethylentriaminpentaessigsäure (DTPA)[9] | Ethylenglycol-bis(aminoethylether)-N,N,N',N'-tetraessigsäure (EGTA) |
 |  |  |
| BAPTA | Tetranatriumiminodisuccinat (IDS) | 1,4,7,10-Tetraazacyclododecan-1,4,7,10-tetraessigsäure (DOTA)[9] |
 |  |  |
| HEDTA | Ethylendiamindibernsteinsäure (EDDS) | Polyasparaginsäure |
 |  |  |
| β-Alanindiessigsäure (β-ADA) | Methylglycindiessigsäure (MGDA) | Fura-1/2/3 |
Einzelnachweise
- ↑ a b Margarete Bucheli-Witschel, Thomas Egli: Environmental fate and microbial degradation of aminopolycarboxylic acids. In: FEMS Microbiology Reviews. Band 25, Nr. 1, Januar 2001, S. 69–106, doi:10.1111/j.1574-6976.2001.tb00572.x.
- ↑ Zahmer Teufel. In: Der Spiegel. Nr. 42, 1971, S. 198 (online – 11. Oktober 1971).
- ↑ Complexometric Titration with Aminopolycarboxylic Acids (EDTA and Analogs). Abgerufen am 11. Juli 2014.
- ↑ Shinji Fushiya, Yoshikazu Sato, Shigeo Nozoe, Kyosuke Nomoto, Tsunematsu Takemoto, Sei-ichi Takagi: Avenic acid, a new amino acid possessing an iron chelating activity. In: Tetrahedron Letters. Band 21, Nr. 32, Januar 1980, S. 3071–3072, doi:10.1016/s0040-4039(00)77410-7.
- ↑ Mugineic acid, a phytosiderophore
- ↑ Waldemar Ternes: Biochemie der Elemente: Anorganische Chemie biologischer Prozesse. Springer DE, 2013, ISBN 3-8274-3020-8, S. 115 (eingeschränkte Vorschau in der Google-Buchsuche).
- ↑ Miloš Buděšínský, Herbert Budzikiewicz, Želimír Procházka, Helmut Ripperger, Axel Römer, Günter Scholz, Klaus Schreiber: Nicotianamine, a possible phytosiderophore of general occurrence. In: Phytochemistry. Band 19, Nr. 11, Januar 1980, S. 2295–2297, doi:10.1016/s0031-9422(00)91014-8.
- ↑ G. Schwarzenbach, E. Kampitsch, R. Steiner: Komplexone II. Das Komplexbildungsvermögen von Iminodiessigsäure, Methylimino-diessigsäure, Aminomalonsäure und Aminomalonsäure-diessigsäure. In: Helvetica Chimica Acta. Band 28, Nr. 1, 1945, S. 1133–1143, doi:10.1002/hlca.194502801158.
- ↑ a b c Giorgio Anderegg, Francoise Arnaud-Neu, Rita Delgado, Judith Felcman, Konstantin Popov: Critical evaluation of stability constants of metal complexes of complexones for biomedical and environmental applications* (IUPAC Technical Report). In: Pure and Applied Chemistry. 77, 2005, doi:10.1351/pac200577081445.
Auf dieser Seite verwendete Medien
Struktur von Asparaginsäure
allg. Schema für Fura-1, Fura-2 und Fura-3
Autor/Urheber: Dominik-jan, Lizenz: CC0
Structure of polyaspartic acid with alpha and beta repeating units
Struktur von Diethylentriaminpentaessigsäure (DTPA)
Autor/Urheber: ChemDoc 2010, Lizenz: CC BY-SA 3.0
beta-alaninediacetic acid structural formula
Aveninsäure A
Struktur von BAPTA
Strukturformel von HEDTA
Structure of 2,2'-(ethane-1,2-diylbis(azanediyl))disuccinic acid
Aminomalonsäure
The chemical structure of rhizoferrin. In bacteria it exists as the S,S enantiomer; in fungi it exists as the R,R enantiomer.
Struktur von EGTA
Structure of 2,2'-azanediyldiacetic acid
Die Strukturformel von DOTA = 1,4,7,10-Tetraazacyclododecan-1,4,7,10-tetraessigsäure
Struktur von Tetranatriumiminodisuccinat
The chemical structure of rhizobactin DM4 or (N2)-[2-[(1-carboxyethyl)amino]ethyl]-N6-(3-carboxy-3-hydroxy-1-oxypropyl)lysine, as indicated in Smith, M. J. et al. (1985) "Rhizobactin, a structurally novel siderophore from Rhizobium meliloti" J. Am. Chem. Soc. 107 (6):1739–1743 doi:10.1021/ja00292a047
Mugineinsäure
The chemical structure of rhizobactin 1021 or (E)-4-[[3-(acetylhydroxyamino)propyl]-amino]-2-hydroxy-2-[2-[3-[hydroxy(1-oxo-2-decenyl)amino]propyl]amino]-2-oxoethyl]-4-oxobutanoic acid, as indicated in Persmark, M. et al. (1993) "Isolation and structure of rhizobactin 1021, a siderophore from the alfalfa symbiont Rhizobium meliloti 1021." J. Am. Chem. Soc. 115 (10):3950–3956 doi:10.1021/ja00063a014
Struktur von 2,2',2''-Nitrilotriessigsäure
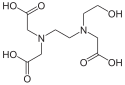
Struktur von EDTA